Escolar Documentos
Profissional Documentos
Cultura Documentos
Resumo - Estrutura e Função Do Genoma
Enviado por
humbertosouza1alves0 notas0% acharam este documento útil (0 voto)
8 visualizações11 páginasTítulo original
Resumo - Estrutura e Função do Genoma
Direitos autorais
© © All Rights Reserved
Formatos disponíveis
DOCX, PDF, TXT ou leia online no Scribd
Compartilhar este documento
Compartilhar ou incorporar documento
Você considera este documento útil?
Este conteúdo é inapropriado?
Denunciar este documentoDireitos autorais:
© All Rights Reserved
Formatos disponíveis
Baixe no formato DOCX, PDF, TXT ou leia online no Scribd
0 notas0% acharam este documento útil (0 voto)
8 visualizações11 páginasResumo - Estrutura e Função Do Genoma
Enviado por
humbertosouza1alvesDireitos autorais:
© All Rights Reserved
Formatos disponíveis
Baixe no formato DOCX, PDF, TXT ou leia online no Scribd
Você está na página 1de 11
ESTRUTURA E FUNCIONAMENTO DE
ÁCIDOS NUCLÉICOS
1. O DNA É O MATERIAL GENÉTICO
No início das pesquisas pela macromolécula celular capaz de armazenar a
informação hereditária e passá-la com fidelidade para a geração seguinte, os
pesquisadores estavam tentando identificar uma possível proteína ou proteínas
com estas funções. Até aquela data, as principais funções das células como
transporte, citoesqueleto, catálise etc., eram realizadas por proteínas ou complexos
proteicos. Não seria nenhuma surpresa se proteínas com função hereditária fossem
descobertas pelos pesquisadores, mas nenhum conseguia tal feito.
Em 1928, o bacteriólogo inglês Frederick Griffith descobriu um dos
mecanismos pelo qual bactérias podem captar do meio de cultura alguma
macromolécula capaz de alterar uma característica fenotípica, e esta alteração ser
transmitida para as células-filhas. A este fenômeno Griffith deu o nome
de transformação bacteriana. Brevemente, os experimentos de Griffith foram os
seguintes:
Griffith possuía no laboratório duas linhagens diferentes de Streptococcus
pneumoniae. Uma linhagem denominada S apresentava duas características
fenotípicas singulares – quando crescida em meio de cultura sólido formava
colônias com aspecto liso e eram virulentas, isto é, capazes de causar pneumonia
em camundongos se inoculadas nestes animais e matá-los (Figura 1, tratamento 2
- controle). A outra linhagem era denominada R e apresentava como características
fenotípicas, colônias de aspecto rugoso e eram avirulentas, isto é, inócuas para os
camundongos caso injetadas nestes animais (Figura 1, tratamento 2 - controle). No
seu experimento, Griffith cresceu as duas linhagens bacterianas em meio líquido e
matou por aquecimento as bactérias S. As bactérias S mortas pelo calor eram agora
inócuas para os camundongos (Figura 1, tratamento 3). Griffith misturou então as
duas bactérias, a linhagem R mais a linhagem S morta pelo calor, e inoculou em
camundongos. A mistura foi capaz de causar pneumonia nos camundongos e
matá-los (Figura 1, tratamento 4). Griffith então sugeriu que a linhagem R,
avirulenta, era capaz de captar alguma macromolécula da linhagem S morta pelo
calor e este fator era responsável pela transformação do fenótipo avirulento da
linhagem R para virulento. Quando ele isolou a bactéria responsável pela morte dos
camundongos ele viu que o fenótipo destas em meio sólido era de colônias lisas.
Griffith imaginou que esta macromolécula pudesse ser o carboidrato da cápsula
bacteriana que é responsável pelos dois fenótipos observados na linhagem S e que
adsorveu na superfície das bactérias R, ou uma outra macromolécula distinta da
linhagem S. Ele imaginava que pudesse ser uma proteína, mas era cético quanto as
demais macromoléculas como os ácidos nucleicos devido a simplicidade da
composição destes, baseada em quatro nucleotídeos. O fator ‘transformador’ de
Griffith ficou então sem identificação.
Figura 1. Transformação bacteriana
Em 1944, três pesquisadores americanos, Oswald Avery, Colin MacLeod e
Maclyn McCarty, trabalhando com o modelo de Griffith, chegaram ao indício que o
DNA seria a macromolécula celular responsável pela transformação do
fenótipo R em S de S. pneumoniae. Ao invés de misturar a linhagem S morta pelo
calor às células R, eles primeiramente fracionaram estas células S mortas por calor e
eliminaram enzimaticamente os polissacarídeos da cápsula, lipídeos, RNAs,
proteínas ou DNAs. Então eles adicionaram estas frações tratadas na cultura da
linhagem R e foram ver qual era capaz de transformar o fenótipo rugoso de R em
liso. A eliminação dos polissacarídeos da cápsula bacteriana S não interferiu na
transformação do fenótipo mostrando que não era apenas por adsorção destes na
superfície da linhagem R. A eliminação dos lipídeos, RNAs e proteínas também não
impediu a transformação do fenótipo rugoso em liso. Já a eliminação de DNAs do
extrato S por DNAse levou ao desaparecimento de colônias de aspecto liso nas
placas de cultura das células R. Desta forma, esta foi a primeira evidência
experimental que o DNA, mesmo com sua simplicidade de composição química,
era a macromolécula celular responsável pela manutenção da informação
hereditária. Ainda assim havia ceticismo na comunidade científica (Figura 2).
Figura 2. Experimento de Avery, MacLeod e McCarty.
Alguns anos mais tarde, em 1952, Hershey e Chase trabalhando num
modelo de infecção bacteriana por bacteriófago T2, tiveram também indícios de
que o DNA seria a macromolécula viral que guarda todas as informações
necessárias para a replicação do vírus. Eles propagaram vírus em meio com fontes
radioativas para a marcação seletiva do capsídeo proteico ou do DNA. Como não
há enxofre no DNA e há dois aminoácidos sulfonados nas proteínas, metionina e
cisteína, eles marcaram as proteínas virais com metionina contendo 35S. Em outro
lote de vírus, eles marcaram o DNA com o isótopo radioativo do fósforo, 32P. Apesar
de algumas proteínas serem fosforiladas, esta fosforilação na maior parte do casos
é transitória. Já o DNA tem um fósforo em cada nucleotídeo que o compõe, sendo
então marcado com a incorporação de algum dos nucleotídeos contendo 32P. Eles
então misturaram os vírus com sua bactéria hospedeira e após um período de
incubação, desfizeram o contato entre eles com a ajuda de um liquidificador e
centrifugaram a mistura a altas rotações. Os vírus permanecem no sobrenadante e
as bactérias vão para o fundo do tubo na forma de precipitado. A radioatividade
recuperada quando os vírus tinham o capsídeo protéico marcado estava toda
associada ao sobrenadante e quase nada no precipitado bacteriano, contudo as
bactérias estavam infectadas e produzindo novos vírus. A radioatividade
recuperada quando os vírus tinham o DNA marcado estava toda associada com o
precipitado bacteriano, indicando que o DNA viral foi introduzido dentro da
bactéria e era o responsável por codificar novas partículas virais (Figura 3).
Figura 3. Experimento de Hershey e Chase (Transdução viral)
2. ESTRUTURA DO DNA
Quanto sua composição química, o DNA é um ácido nucléico composto por 4
nucleotídeos que diferem na base nitrogenada
(adenina, guanina, citosina ou timina). Cada nucleotídeo contém um
fosfato, uma desoxirribose (açúcar) e uma das quatro bases
nitrogenadas sendo denominados dAMP, dGMP, dCMP e dTMP, respectivamente.
Duas bases nitrogenadas são do tipo PURINA (adenina e guanina) e duas do
tipo PIRIMIDINA (citosina e timina). As bases nitrogenadas são unidas à
desoxirribose pelo carbono 1’ por uma ligação glicosídica, o carbono 2’ tem um
hidrogênio (na ribose esta posição é ocupada por uma hidroxila, o que torna o RNA
uma molécula mais reativa e instável que o DNA), o carbono 3’ tem uma hidroxila
que faz uma ligação fosfodiéster com o fosfato presente no carbono 5’ do
nucleotídeo adjacente (Figura 4).
Figura 4. Nucleotídeos do DNA.
O bioquímico austríaco Erwin Chargaff, estudando a composição do DNA de
diferentes organismos, estabeleceu regras empíricas sobre as quantidades de cada
componente do DNA:
a quantidade de nucleotídeos pirimídicos (T + C) sempre é igual a quantidade
de nucleotídeos purínicos (A + G);
a quantidade de adenina (A) é sempre igual a de timina (T) e a quantidade de
guanina (G) é sempre igual a de citosina (C).
Porém, nos diferentes DNAs a quantidade de A + T não é igual a quantidade de G
+ C, por exemplo:
Staphylococcus aureus 18% G + C e 82% A + T
Homo sapiens 40% G + C e 60% A + T
Escherichia coli 51% G + C e 49% A + T
Micrococcus luteus 75% G + C e 25% A + T
Figura 5. Pareamentos legítimos de nucleotídeos (pares de Watson e Crick)
Dois outros bioquímicos, Rosalind Franklin e Maurice Wilkins, trabalhando com
cristalografia de raios X, obtiveram cristais de DNA em condições de alta salinidade
ou em condições fisiológicas, e a posição dos átomos no cristal puderam ser
mapeadas com precisão pelas imagens obtidas pela difração dos raios X. Os dados
obtidos mostravam o DNA como longo e fino, com um esqueleto de fosfatos e
pentoses no exterior da molécula, com duas partes semelhantes e paralelas ao
longo da molécula, e uma estrutura helicoidal.
Com os achados de Chargaff mais os resultados de Franklin e Wilkins (mesmo
ainda não publicados, mas apresentados informalmente em um seminário de
pesquisa), Watson e Crick em 1953 propuseram como seria a estrutura
tridimensional do DNA e como ele replicaria. Segundo sua teoria, o DNA é uma
dupla hélice composta de cadeias de nucleotídeos ligados por ligações
fosfodiéster formando um esqueleto externo de fosfato-pentose (só possível com
os resultados experimentais de cristalografia de raios-X); as duas hélices são
mantidas juntas por pontes de hidrogênio entre as bases nitrogenadas
complementares - o par A+T forma duas pontes de hidrogênio e o par G+C
forma três pontes de hidrogênio (só possível com os resultados de Chargaff);
as duas hélices têm polaridade inversa sendo chamadas de filamento 5´→3´ e a
outra de filamento 3´→5´; o empilhamento dos pares de bases na dupla hélice
forma dois sulcos na sequência pentose-fosfato, chamados de sulco maior e menor
(só possível com os resultados experimentais de cristalografia de raios-X) (Figura
6).
Figura 6. O empilhamento dos pares de bases na dupla hélice do DNA, filamento
5´→3´ e filamento 3´→5´.
O DNA apresenta três formas estruturais: o DNA A é menos hidratado e mais
compacto que o DNA B e ambos produzem hélices com giro para a direita; o
DNA Z tem estrutura em ziguezague e produz uma hélice com giro para a
esquerda.
A replicação proposta por Watson e Crick ocorreria de
maneira semiconservativa, isto é, os dois DNAs filhos resultantes seriam
compostos por um dos filamentos parentais (utilizado como molde) e um filamento
novo de DNA recém-sintetizado. Novamente, havia a necessidade de dados
experimentais para a comprovação desta hipótese.
3. REPLICAÇÃO DO DNA
Na época da elucidação da estrutura do DNA houve então a proposição de três
teorias de como as moléculas de DNA se duplicariam. Havia a teoria proposta por
Watson e Crick da replicação ser semiconservativa, mas devido a não
comprovação experimental duas outras teorias foram também propostas: a
replicação conservativa, o dúplex-filho contém dois filamentos recém-sintetizados
e o filamento parental é conservado, e a replicação dispersiva, os dúplices-filhos
contém segmentos do filamento parental e DNA recém-sintetizado. Destas teorias,
a menos provável seria a dispersiva pois é a única que envolveria a quebra das duas
fitas do DNA parental para se ajuntarem e formarem novamente os novos DNAs
filhos. As teorias semiconservativa e conservativa propunham apenas a abertura da
dupla hélice por rompimento das pontes de hidrogênio entre os pares de bases
complementares, sem haver quebra do esqueleto de fosfato-pentose (Figura 7).
Figura 7 . Modelos teóricos para a replicação do DNA
Em 1958 veio a comprovação experimental de que a replicação do DNA
ocorreria de maneira semiconservativa. No experimento de Meselson-
Stahl, Escherichia coli foi crescida em meio com o isótopo pesado do nitrogênio 15N
ao invés do isótopo normal leve 14N por várias gerações e então postas em meio
com 14N. O DNA foi extraído das bactérias após uma ou duas multiplicações
celulares (1a e 2a gerações, respectivamente) e separados por densidade em
gradiente de CsCl por ultracentrifugação. O resultado obtido na 1 a geração foi uma
única banda de DNA de densidade intermediária aos DNAs pesado (com 15N) e leve
(com 14N) e na 2a geração DNAs de densidades intermediária e leve (Figura 8).
Figura 8. Experimento de Meselson-Stahl demonstrando a replicação
semiconservativa do DNA.
Mecanismo Molecular de Replicação do DNA
O DNA se autoduplica a partir de pontos específicos conhecidos
como origens de replicação. A característica mais marcante destas origens é a
riqueza em pares A+T, o que significa menor gasto de energia para a abertura da
hélice nestes pontos. A replicação pode ser uni ou bidirecional, segundo se
formem, na origem, uma ou duas forquilhas de replicação que migram em
direções opostas (Figura 9).
Figura 9. A replicação do DNA se inicia em várias Origens de replicação, que
viram Bolhas de Replicação com duas Forquilhas de Replicação em direções
opostas, até a fusão dos replicons.
Várias proteínas estão envolvidas na replicação do DNA:
1. DNA polimerases: sintetizam a fita nova de DNA de 5´ para 3´ adicionando o
novo dNTP pelo seu grupo fosfato 5´ à hidroxila na posição 3´ da desoxirribose do
precedente; além da atividade de síntese do novo DNA, a DNA polimerase também
possui atividades de exonuclease de 5´ para 3´, necessária para a remoção dos
primers de RNA, e de exonuclease de 3´ para 5´, necessária para a revisão do
último nucleotídeo adicionado (atividade revisora que garante a fidelidade da
cópia pois a enzima só prossegue se as pontes de hidrogênio do par de bases se
formarem);
2. Helicase: desenrola o duplex de DNA desfazendo as pontes de hidrogênio
entre os pares de bases complementares às custas de energia (hidrólise de ATP);
3. DNA primase: sintetiza os primers (iniciadores) de RNA necessários como
substrato para o início da síntese do novo filamento de DNA pela DNA polimerase;
4. Topoisomerases: introduzem cortes no duplex de DNA à frente da forquilha
de replicação para liberar a tensão rotacional na molécula ainda dupla-fita gerada
pela abertura da hélice pela helicase;
5. Proteínas SSB (ligação a DNA fita simples): estabilizam os filamentos de fita
única desenrolados pela helicase, evitando que estes voltem a se associar
espontaneamente;
6. DNA ligase: liga os fragmentos de Okasaki (fosfato 5´ à hidroxila na posição 3´
do nucleotídeo seguinte).
A síntese do novo filamento de DNA ocorre de maneira diferente nos dois
filamentos parentais de polaridade inversa. Na fita 3´ para 5´ do molde, o novo
filamento vai sendo sintetizado na direção 5´ para 3´ continuamente pela DNA
polimerase e o filamento é denominado condutor ou leading. Na fita 5´ para 3´ do
molde, o novo filamento vai sendo sintetizado na direção 5´ para 3´ de
forma descontinua pela DNA polimerase usando curtos iniciadores ou primers de
RNA que fornecem o grupo 3´OH necessário à ação da enzima, e o filamento é
denominado retardatário ou lagging (Figura 10). Nas forquilhas de replicação que
estão indo em direções opostas, o filamento condutor numa forquilha será
retardatário na outra e vice-versa. Assim, se considerarmos a replicação do DNA
como um todo podemos concluir que a replicação das duas fitas de polaridade
inversa é descontínua.
Figura 10. Esquemas da replicação descontínua de DNA por fragmentos de
Okasaki no filamento lagging (molde 5´para 3') e contínua no
filamento líder (molde 3´para 5') considerando apenas uma das forquilhas de
replicação da bolha de replicação.
Você também pode gostar
- Codigos de Falhas Rede Can Fiat PDFDocumento13 páginasCodigos de Falhas Rede Can Fiat PDFtadeu87% (30)
- Equipamentos IndustriaisDocumento473 páginasEquipamentos IndustriaisLuis Gomes100% (2)
- Ficha Replicação DNADocumento4 páginasFicha Replicação DNAAnatole Enes100% (1)
- Genética Médica para Médicos e Estudantes de Medicina: OncologiaNo EverandGenética Médica para Médicos e Estudantes de Medicina: OncologiaAinda não há avaliações
- Ficha de Trabalho 11.º AnoDocumento6 páginasFicha de Trabalho 11.º AnoBruna7MarquesAinda não há avaliações
- Resumo & Guia De Estudo - Cancerlândia: Memórias De Um Médico Sobre Câncer E Pesquisa Com Células-TroncoNo EverandResumo & Guia De Estudo - Cancerlândia: Memórias De Um Médico Sobre Câncer E Pesquisa Com Células-TroncoAinda não há avaliações
- Teste 1 11o B Versao 1Documento6 páginasTeste 1 11o B Versao 1Filipa OliveiraAinda não há avaliações
- Memorial Regularizacao PDFDocumento2 páginasMemorial Regularizacao PDFCaroliny SabasiAinda não há avaliações
- Plano de Prevenção e Proteção Contra IncêndioDocumento8 páginasPlano de Prevenção e Proteção Contra IncêndioDavi Alves BatalhaAinda não há avaliações
- Fabricação de HidromelDocumento11 páginasFabricação de HidromelwelberromeroAinda não há avaliações
- Salame ArtesanalDocumento6 páginasSalame ArtesanalHedge TraderAinda não há avaliações
- Manual Autoclave Vitale PlusDocumento34 páginasManual Autoclave Vitale PlusAna Clara Costa50% (4)
- Ficha 4 - Replicação Do DNA - CópiaDocumento2 páginasFicha 4 - Replicação Do DNA - CópiaLeonor MariaAinda não há avaliações
- Proteg TCC - TriclorocarbanDocumento2 páginasProteg TCC - TriclorocarbanallanHF100% (1)
- Guia Prático Do Pêndulo - D. Jurriaanse - Doc - IlustradoDocumento56 páginasGuia Prático Do Pêndulo - D. Jurriaanse - Doc - IlustradoTerezinha Flores Canabarro100% (1)
- QD1 - Construindo o Conceito de DNA Correcao PDFDocumento3 páginasQD1 - Construindo o Conceito de DNA Correcao PDFfernando100% (1)
- Fotossintese e RespiraçãoDocumento31 páginasFotossintese e RespiraçãoCharlesmaffraAinda não há avaliações
- B 01Documento49 páginasB 01Rita Brito100% (1)
- Verticalização de Molar Com Mini ImplanteDocumento4 páginasVerticalização de Molar Com Mini ImplanterfbsAinda não há avaliações
- Biologia 11 AnoDocumento9 páginasBiologia 11 AnoFrancisca Fonseca100% (4)
- Biologia Molecular TEXTO 1 O ÁCIDO DESOXIRRIBONUCLÉICO - DNA. A Descoberta Do DNA Experimentos ClássicosDocumento22 páginasBiologia Molecular TEXTO 1 O ÁCIDO DESOXIRRIBONUCLÉICO - DNA. A Descoberta Do DNA Experimentos ClássicosjcferreiraAinda não há avaliações
- Ficha Nº1 PDFDocumento12 páginasFicha Nº1 PDFLuis FilipeAinda não há avaliações
- Escola Secundaria Cacilhas Teste 1 ADocumento10 páginasEscola Secundaria Cacilhas Teste 1 AjpfixolasAinda não há avaliações
- Ficha Nº 3 - Crescimento e Renovação CelularDocumento6 páginasFicha Nº 3 - Crescimento e Renovação CelularRodissauro 1Ainda não há avaliações
- Texto de Apoio II de GGP 2017Documento17 páginasTexto de Apoio II de GGP 2017CAPAINA BENCILARIO ANDRE FAinda não há avaliações
- Unidade 1 - o DogmaDocumento28 páginasUnidade 1 - o DogmaPaulo ViniciusAinda não há avaliações
- Resumo Ácidos NucleicosDocumento9 páginasResumo Ácidos NucleicosTuanne Dos SantosAinda não há avaliações
- Apostila BioMolDocumento25 páginasApostila BioMolAriádine CasasAinda não há avaliações
- Biologia 2021Documento8 páginasBiologia 2021sunaraAinda não há avaliações
- 1 - Expressão Da Informação GenéticaDocumento30 páginas1 - Expressão Da Informação GenéticaHelioAinda não há avaliações
- 1718 F1 DNA 11anoDocumento11 páginas1718 F1 DNA 11anoRicardo VasconcelosAinda não há avaliações
- Resumoglobalbiologia 11 AnoDocumento25 páginasResumoglobalbiologia 11 Anocarlbisp100% (1)
- Acidos NucleicosDocumento18 páginasAcidos NucleicosLeonor NevesAinda não há avaliações
- Exercicios - Farmácia - Biomol 18.05.2021 para EntregaDocumento4 páginasExercicios - Farmácia - Biomol 18.05.2021 para EntregaWilliam CarraraAinda não há avaliações
- Dna Apostila 1Documento9 páginasDna Apostila 1Clebson SantanaAinda não há avaliações
- Apotila - DNA - Histórico e EstruturaDocumento10 páginasApotila - DNA - Histórico e EstruturaJustino ClaudiaAinda não há avaliações
- DNA - História e EstruturaDocumento42 páginasDNA - História e EstruturamarianaAinda não há avaliações
- Experimento de GriffithDocumento7 páginasExperimento de GriffithIsabela SousaAinda não há avaliações
- Lista de Biologia - 2º Ano Do Ensino MédioDocumento8 páginasLista de Biologia - 2º Ano Do Ensino MédioGuilherme HennaAinda não há avaliações
- Apontamentos Biologia 11 AnoDocumento24 páginasApontamentos Biologia 11 AnoAfonso FreireAinda não há avaliações
- 1.1. Acidos Nucleicos e Replicacao Do DNA 2022Documento46 páginas1.1. Acidos Nucleicos e Replicacao Do DNA 2022SofiaAinda não há avaliações
- Resumoglobalbiologia 11 AnoDocumento5 páginasResumoglobalbiologia 11 Anomiguel2007mendes25Ainda não há avaliações
- Aula - 2 Material Genética Estrutura e Organização Do DNADocumento20 páginasAula - 2 Material Genética Estrutura e Organização Do DNARaner santanaAinda não há avaliações
- Tarefa Mar2024 - 2serie2024Documento5 páginasTarefa Mar2024 - 2serie2024gabriel.kenzo18Ainda não há avaliações
- C3 CursoE Biologia A 20aulasDocumento16 páginasC3 CursoE Biologia A 20aulasHancock86Ainda não há avaliações
- Apontamentos de BiologiaDocumento6 páginasApontamentos de Biologialara albinoAinda não há avaliações
- 8vii Exercicios Porto Editora BG 11o Ano XXDocumento3 páginas8vii Exercicios Porto Editora BG 11o Ano XXJoaquina JúliaAinda não há avaliações
- Crescimento e Renovacao CelularDocumento33 páginasCrescimento e Renovacao CelularLaura PintoAinda não há avaliações
- Tema 5 - Biologia 11ºanoDocumento16 páginasTema 5 - Biologia 11ºanoSilvia RibeiraAinda não há avaliações
- Cenas Raras.Documento5 páginasCenas Raras.Leo JesusAinda não há avaliações
- Acidos NucleicosDocumento16 páginasAcidos NucleicosRick DouglasAinda não há avaliações
- Localizacao Da Informacao GeneticaDocumento5 páginasLocalizacao Da Informacao GeneticaNareida MonteiroAinda não há avaliações
- Bg11 Teste 1 FinalDocumento10 páginasBg11 Teste 1 FinalBeatriz Silva100% (1)
- Ácidos Nucleicos e Síntese ProteicaDocumento5 páginasÁcidos Nucleicos e Síntese ProteicaThaís AraújoAinda não há avaliações
- Breve Histórico Da Descoberta Do DnaDocumento17 páginasBreve Histórico Da Descoberta Do DnaGiovani Hideo TutihashiAinda não há avaliações
- Projeto Exame - Unidades 4, 5 e 6Documento11 páginasProjeto Exame - Unidades 4, 5 e 6pepeAinda não há avaliações
- Material de Leitura GMBDocumento124 páginasMaterial de Leitura GMBnelson daniel fermengaAinda não há avaliações
- Resumo Biologia MolecularDocumento169 páginasResumo Biologia MolecularPatrícia SequeiraAinda não há avaliações
- O DNA em OrigamiDocumento4 páginasO DNA em Origamipassosjeanne9858Ainda não há avaliações
- Fichaa 3Documento7 páginasFichaa 3maria araújoAinda não há avaliações
- VOD - Biologia - Ácidos NucleicosDocumento11 páginasVOD - Biologia - Ácidos NucleicosSidney MendesAinda não há avaliações
- Genética PPT 1 9Documento62 páginasGenética PPT 1 9franciscojulio2005Ainda não há avaliações
- 2 Dna e Replicação ExperiênciasDocumento22 páginas2 Dna e Replicação ExperiênciasPedro BaetaAinda não há avaliações
- Ácidos NucleicosDocumento34 páginasÁcidos NucleicosTomás SilvaAinda não há avaliações
- Ficha de Exercícios ADocumento3 páginasFicha de Exercícios A64dxb6mqp8Ainda não há avaliações
- I - Importância Do Dna PDFDocumento21 páginasI - Importância Do Dna PDFSandra Nascimento0% (1)
- DnaDocumento8 páginasDnaDaniela RodriguesAinda não há avaliações
- Conversas com o ChatGPT sobre Ciência para jovens: A Biologia Molecular!: Descubra os segredos da vida com a ajuda da inteligência artificialNo EverandConversas com o ChatGPT sobre Ciência para jovens: A Biologia Molecular!: Descubra os segredos da vida com a ajuda da inteligência artificialAinda não há avaliações
- FISPQ SGQ VT20 Limpa Estofados PDFDocumento6 páginasFISPQ SGQ VT20 Limpa Estofados PDFjsbarcellosAinda não há avaliações
- Estufa de Esterilização e Secagem Solid SteelDocumento6 páginasEstufa de Esterilização e Secagem Solid SteelLeandro GonçalvesAinda não há avaliações
- NBR 09479 1994 Cura de CP de ConcretoDocumento2 páginasNBR 09479 1994 Cura de CP de ConcretoDavi FonsecaAinda não há avaliações
- AULA 3 BIOLOGIA CELULAR - Membranas Biologicas-1Documento81 páginasAULA 3 BIOLOGIA CELULAR - Membranas Biologicas-1Levi CamoesAinda não há avaliações
- Ficha de Emergência - FET - Standak® TopDocumento1 páginaFicha de Emergência - FET - Standak® TopPCdoB de PinheirosAinda não há avaliações
- Descrição Dos Principais Materiais de LaboratórioDocumento4 páginasDescrição Dos Principais Materiais de LaboratórioAna Carolina Monteiro100% (1)
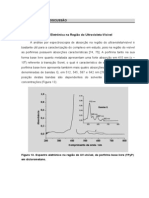
- Discussão Dos Resultados - UV - VisDocumento5 páginasDiscussão Dos Resultados - UV - VisPatrick Rodrigues BatistaAinda não há avaliações
- Bomba CentrifugaDocumento18 páginasBomba CentrifugaMichael CamargoAinda não há avaliações
- Apostila Orgânica 4º SemestreDocumento16 páginasApostila Orgânica 4º Semestreanon_226130384Ainda não há avaliações
- Álcool A Partir de MilhoDocumento10 páginasÁlcool A Partir de MilhoMargareth CunhaAinda não há avaliações
- Relatório 10 - Determinação de Água de Critalização Por Gravimetria de VolatilizaçãoDocumento5 páginasRelatório 10 - Determinação de Água de Critalização Por Gravimetria de VolatilizaçãoandresaAinda não há avaliações
- Reciclagem IsoporDocumento7 páginasReciclagem IsoporeusebiopAinda não há avaliações
- Fispq Borden CleanDocumento6 páginasFispq Borden CleanMarcos AntonioAinda não há avaliações
- Tecnologia de Aplicação de Defensivos - UNESPDocumento28 páginasTecnologia de Aplicação de Defensivos - UNESPsartooriAinda não há avaliações
- Velocidade de Hidratação Da CalDocumento4 páginasVelocidade de Hidratação Da CalVictor ReginoAinda não há avaliações
- AglomeranteDocumento272 páginasAglomeranteelisa120Ainda não há avaliações
- BP Gasolina Ultimate 98 Gasolina Schumbo 98DDocumento39 páginasBP Gasolina Ultimate 98 Gasolina Schumbo 98DIsabelDrummondAinda não há avaliações
- Betao LeveDocumento2 páginasBetao LeveJose Manuel DomingosAinda não há avaliações