Escolar Documentos
Profissional Documentos
Cultura Documentos
Sebenta PDF
Enviado por
FCiênciasTítulo original
Direitos autorais
Formatos disponíveis
Compartilhar este documento
Compartilhar ou incorporar documento
Você considera este documento útil?
Este conteúdo é inapropriado?
Denunciar este documentoDireitos autorais:
Formatos disponíveis
Sebenta PDF
Enviado por
FCiênciasDireitos autorais:
Formatos disponíveis
2006/2007
1 ANO 2 SEMESTRE
BIOLOGIA MOLECULAR
Jorge Paulos
Biologia Molecular - 1 Ano - FFUP
2006/2007
Captulo 1 - Genes |
Biologia Molecular 1 Ano - FFUP
2006/2007
Captulo 1 - Genes
1. Conceito de gene
A vida depende da capacidade das clulas em armazenar, obter e traduzir as instrues genticas necessrias para manter o organismo vivo. Esta informao hereditria passada de uma clula s suas clulas-filhas, durante a diviso celular, e de uma gerao de um organismo para outro, por meio das suas clulas reprodutoras. Essas instrues so armazenadas, em todas as clulas vivas, nos genes, os elementos que contm a informao que determina as caractersticas de uma espcie como um todo, bem como as de um indivduo. A maioria dos genes humanos consiste de longos segmentos de exes e de intres alternados, sendo que a maior parte do gene consiste de intres. Por outro lado, a maioria dos genes de um organismo com genomas compactos no possui introes. Isso explica o tamanho bastante menor dos seus genes, bem como a grande proporo de DNA codificante nos seus cromossomas. Alm dos exes e dos intres, cada gene est associado s sequncias de DNA reguladoras, as quais so responsveis por garantir que o gene seja expresso nos nveis, no momento e no tipo celular adequados. No homem, as sequncias reguladoras de um gene tpico esto dispersas por cerca de dezenas de milhares de pares de nucletidos. Essas sequncias reguladoras so mais comprimidas em organismos com genoma compacto.
2. Informao gentica em eucariotas e procariotas
Por meio de um microscpio simples, possvel observar claramente que os organismos vivos podem ser classificados, de acordo com as estruturas internas da clula, em dois grupos: os eucariotas e os procariotas. Os eucariotas mantm o seu DNA num compartimento limitado por uma membrana, chamado ncleo.
| Captulo 1 - Genes
Biologia Molecular - 1 Ano - FFUP
2006/2007
Os procariotas no possuem um ncleo compartimentado limitado para abrigar o seu DNA. A maioria das clulas procariticas pequena e simples e, geralmente, vivem como indivduos independentes, tal como os organismos multicelulares. Os procariotas so tipicamente esfricos ou em forma de bastes, medindo poucos micrmetros de comprimento. Frequentemente, apresentam uma capa protectora flexvel, chamada parede celular, seguida da membrana plasmtica que envolve um nico compartimento citoplasmtico que contm DNA, RNA, protenas e uma grande quantidade de molculas pequenas necessrias vida.
3. Informao gentica nos eucariotas
As clulas eucariticas, em geral, so maiores e mais elaboradas que as clulas procariticas e, consequentemente, os seus genomas so maiores e mais elaborados tambm. Esta diferena acompanhada por diferenas radicais nas estruturas e nas funes celulares. Alm disso, muitas classes de clulas eucariticas formam organismos multicelulares que atingem um nvel de complexidade que no alcanado pelos procariotas. As informaes genticas das clulas eucariticas tm uma origem hbrida. A maior parte dessa informao armazenada no ncleo, mas uma pequena quantidade permanece dentro da mitocndria e,
Captulo 1 - Genes |
Biologia Molecular 1 Ano - FFUP
2006/2007
em clulas vegetais, dentro dos cloroplastos. O DNA mitocondrial e o DNA dos cloroplastos podem ser separados do DNA nuclear, analisados e sequenciados individualmente. Os genes encontram-se no interior dos cromossomas, que contm cromatina que est includa no ncleo celular. Basicamente, a cromatina pode incluir polmeros de DNA podendo ser eucromatina ou heterocromatina, ou ento pode incluir apenas protenas histnicas ou no histnicas. Os estudos por microscopia ptica, distinguiram dois tipos de cromatina no ncleo interfsico de muitas clulas eucariticas superiores: Heterocromatina forma altamente condensada, que inclui protenas adicionais e, provavelmente, um nvel mais compacto de organizao, que est apenas a comear a ser analisado actualmente. Normalmente, corresponde a 10% do genoma de uma clula e est concentrada em diversas regies como o centrmero e o telmero. A maior parte do DNA compactado em heterocromatina no contm genes, mas o que contm resistente expresso, porque a heterocromatina se encontra compactada de forma no-usual. Eucromatina restante cromatina menos condensada, composta por tipos de estruturas cromossmicas como as fibras de 30 nm. Os cromossomas de um indivduo podem ser cromatdeos irmos (ou cromtidas irms), ou seja, quando so duas cpias de um dado cromossoma, replicado durante a fase S do ciclo celular, que se separam durante a mitose. Existem tambm os cromossomas homlogos que emparelham durante a meiose ou correspondem a cromossomas de espcies diferentes que mantm o mesmo nmero de genes durante a evoluo. Os cromossomas de um indivduo encontram-se organizados num caritipo atravs do seu nmero, tamanho e forma. No caso do caritipo humano, este inclui 23 pares, sendo 22 pares de autossomas (ou cromossomas somticos) e 1 par de heterossomas (ou cromossomas sexuais).
| Captulo 1 - Genes
Biologia Molecular - 1 Ano - FFUP
2006/2007
Tendo em conta estes conceitos, pode definir-se genoma como sendo o conjunto do material gentico de um determinado conjunto de cromossomas de um organismo. Num genoma, os genes pode encontrarse em vrias posies, sendo que cada uma delas se designa locus. Para alm disso, cada forma alternativa de um determinado gene, designa-se alelo. Os alelos podem ser de dois tipos: Alelo dominante alelo que expressa o seu fentipo mesmo quando um heterozigtico com um alelo recessivo presente. Alelo recessivo alelo que no expresso fenotipicamente num determinado heterozigtico. Esta informao gentica de um organismo podem ser expressas de duas formas: Gentipo a composio gentica de um organismo. Fentipo a expresso fsica de um determinado gentipo (morfologia, comportamento, fisiologia e relaes ecolgicas). Quanto aos alelos presentes no genoma, os indivduos podem ser de vrios tipos: Homozigticos quando os dois alelos so idnticos Heterozigticos quando os dois alelos so diferentes.
4. Transformaes laboratoriais que envolvem Gentica
Fred Griffit injectou a forma rugosa da bactria num ratinho que morreu de pneumonia e injectou a forma lisa da bactria noutro ratinho que sobreviveu. Noutra experincia seguinte, inactivou as formas rugosas com temperaturas elevadas e s depois que as injectou noutro ratinho, o qual sobreviveu pois as bactrias inactivas, deixam de ter caractersticas patognicas. Numa ltima experincia, utilizou clulas rugosas inactivas pelo calor e lisas simultaneamente, sendo que como o DNA se encontra presente, entra para a bactria de forma rugosa, passa a conferir-lhe caractersticas patognicas e o ratinho pode vir a desenvolver uma pneumonia.
Captulo 1 - Genes |
Biologia Molecular 1 Ano - FFUP
2006/2007
Alfred Hershey um fago quando infecta uma bactria, tem de injectar o seu material gentico, manter-se no interior da bactria e modificar o DNA. As clulas podem ser marcadas com compostos radioactivos, sendo o mais comum a utilizao de fsforo-32. Para efectuar esta marcao tambm necessrio utilizar algo que faa parte da constituio da protena, como o enxofre-35. Quando o fago infecta a bactria, vai injectar o material e como a replicao do DNA semiconservativa e existem duas cadeias de DNA, vo ser originadas 4 cadeias, existindo uma cadeia radioactiva e outra no radioactiva, devido utilizao do fsforo. Desta forma, analisam-se as cadeias das descendncias vricas e consegue concluir-se acerca da importncia da utilizao destes compostos na infeco de material gentico.
| Captulo 1 - Genes
Biologia Molecular - 1 Ano - FFUP
2006/2007
5. Estrutura do DNA
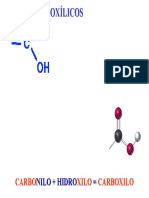
A molcula de DNA consiste de duas longas cadeias polipeptdicas compostas de quatro tipos de subunidades nucleotdicas. Cada uma dessas cadeias conhecida como uma cadeia de DNA. As pontes de hidrognio entre as regies das bases azotadas mantm as duas cadeias juntas. Os nucletidos so compostos de acares com cinco carbonos, aos quais um ou mais grupos fosfato esto ligados, e de uma base contendo azoto. No caso dos nucletidos do DNA, o acar uma desoxirribose ligada a um nico grupo fosfato (por isso o nome cido desoxirribonucleico), e a base pode ser adenina (A), timina (T), guanina (G) e citosina (C). Os nucletidos esto covalentemente ligados numa cadeia atravs dos acares e dos fosfatos, os quais permitem formar a estrutura principal alternada de acar-fosfato-acar-fosfato. Cada cadeia polinucleotdica de DNA semelhante a um colar constitudo por quarto contas diferentes, correspondentes s quatro bases azotadas, pois apneas as bases diferem nos quatro tipo de subunidades. Esses mesmos smbolos so usados para representar os quatro diferentes nucletidos, que so as bases ligadas com os seus grupos fosfato e acar. A estrutura tridimensional do DNA a dupla hlice decorrente das caractersticas qumicas e estruturais das suas cadeias polipeptdicas. Uma vez que essas duas cadeias so mantidas juntas atravs de pontes de hidrognio entre as bases das duas cadeias complementares, todas as bases encontram-se no interior da dupla hlice, e os acares e os fosfatos encontram-se na regio externa. Em cada um dos casos, a base mais robusta, com dois anis (purina), emparelha com uma base com um nico anel (pirimidina). A emparelha sempre com T e G com C. Essa complementaridade de bases permite que os pares de bases sejam dispostos num arranjo energtico mais favorvel no interior da dupla hlice. Neste arranjo, cada par de bases apresenta largura similar, mantendo a estrutura de acar-fosfato equidistante ao longo da molcula de DNA. Para maximizar a eficcia do empacotamento pelo emparelhamento, as duas estruturas enrolam-se uma em torno da outra para formar a dupla hlice, com uma volta completa a cada 10 pares de bases. Os membros de cada par de bases encaixam-se na dupla hlice apenas se as duas cadeias da hlice estiverem na posio antiparalela, isto , apenas se a polaridade de uma cadeia estiver em orientao oposta da outra cadeia. A consequncia desta caracterstica que cada cadeia de uma molcula de DNA contm uma sequncia de nucletidos exactamente complementar da outra cadeia. 8 Captulo 1 - Genes |
Biologia Molecular 1 Ano - FFUP
2006/2007
6. Conformaes do DNA
A maior parte do DNA celular encontra-se sob a forma de hlice. A anlise desta molcula atravs de raios-X informa-nos que as bases azotadas se encontram, geralmente, espaadas de 0.36 nm entre si, ao longo dos eixos da hlice da molcula de DNA. As trs principais conformaes de DNA conhecidas so: B-DNA a forma de DNA mais esperada de encontrar nas clulas. o DNA que respeita completamente as regras de complementaridade de bases azotadas e que conduz existncia de cadeias complementares e antiparalelas. A-DNA existe em condies de baixa humidade, onde a estrutura principal bastante mais compacta, exibindo grandes pores de bases que se encontram a seguir os eixos da hlice de DNA. Z-DNA inclui pequenas molculas de DNA que alternam nucletidos de purinas e pirimidinas, adoptando uma estrutura mais instvel que a estrutura em hlice. As bases criam como que um zig-zag quando visualizadas de lado, sendo que quando o DNA adquire esta conformao particular, ainda apresenta funo desconhecida.
7. Metilao do DNA
A metilao no DNA vertebrado restrita aos nucletidos de citosina na sequncia CG, que faz o emparelhamento de bases com a mesma sequncia (na direco oposta), na outra cadeia de DNA de dupla hlice. Consequentemente, um mecanismo simples permite a existncia de um padro de metilao de DNA a ser herdado directamente pelas cadeias-filhas de DNA. Uma enzima designada metiltransferase de manuteno actua preferencialmente nas sequncias CG referidas anteriormente, que esto emparelhadas com uma sequncia CG j metilada. Como resultado, o padro de metilao de DNA da cadeia-filha de DNA parental serve como molde para a metilao da cadeia-filha de DNA, tornando esse padro directamente herdvel aps a replicao do DNA. Logo aps a fertilizao, ocorre uma ampla onda de desmetilao do genoma, quando a grande maioria dos grupos metilo so perdidos da cadeia de DNA. Essa desmetilao pode ocorrer tanto pela supresso da actividade das metiltransferases de manuteno do DNA, resultando numa perda passiva de grupos metilo durante cada ciclo de replicao de DNA, como por uma enzima especfica de desmetilao.
| Captulo 1 - Genes
Biologia Molecular - 1 Ano - FFUP
2006/2007
8. Tautomerismo
Geralmente, durante o enrolamento das cadeias de DNA, estamos perante um emparelhamento A=T e GC, sendo que as formas tautomricas podem emparelhar com bases que normalmente no emparelham, como por exemplo A com C e G com T. Este processo ocorre normalmente no nosso organismo, mas na coexistncia de sistemas que nos defendem dessas isomerizaes, como os sistemas de reparao de DNA, que quando entram em funcionamento, removem os emparelhamentos de isomerismo e o DNA volta ao seu estado normal. No entanto, quando o emparelhamento de isomerismo no retirado por esses sistemas, quando ocorrer a replicao de DNA seguinte, o erro vai ser corrigido e as bases voltam a emparelhar segundo as regras normais.
9. Sequncias invertidas - Palindromas
Geralmente, um palindroma entendido como uma sequncia de DNA de cadeia dupla, que lida da mesma forma quando as duas cadeias so lidas numa direco definida. Formam-se estruturas cruciformes, apresentando zonas que podem emparelhar por serem perfeitamente idnticas, e quando isso se verifica, adquirem forma de alfinete, conferindo o aspecto de uma cruz molecula de DNA na sua totalidade. Este acontecimento tambm pode ocorrer no RNA, sendo que as estruturas formadas se designam por estruturas secundrias, adquiridas pela paragem da transcrio. Podem tambm adquirir estruturas tercirias, sendo estas caractersticas de um RNA particular, o tRNA, que geralmente apresenta forma de folha de trevo, formando um pseudo-lao.
10
Captulo 1 - Genes |
Biologia Molecular 1 Ano - FFUP
2006/2007
10. Desnaturao e renaturao do DNA
Durante a replicao e a transcrio do DNA, as cadeias de dupla hlice da molcula separam-se para permitir o emparelhamento das partes internas das bases, com as bases dos nucletidos que vo ser polimerizadas em novas cadeias polinucleotdicas. O desenrolamento e a separao do DNA, referido normalmente como desnaturao pode ser induzido experimentalmente atravs de um aumento da temperatura de uma soluo de DNA. medida que a energia trmica aumenta, o aumento do movimento molecular resulta numa quebra das pontes de hidrognio e de outras foras que pudessem estabilizar a dupla hlice. De seguida, as cadeias separam-se e so conduzidas individualmente atravs da repulso electrosttica do grupo fosfato que est contido nas desoxirriboses de cada cadeia. Perto da temperatura de desnaturao, um pequeno aumento da temperatura causa uma perda das fracas interaces que mantinham as cadeias juntas ao longo de todo o comprimento da molcula de DNA, conduzindo a uma mudana abrupta na absorvncia de ultravioletas. A temperatura de fuso (Tm) qual as cadeias de DNA se separam, depende de diversos factores. As molculas que contm uma maior proporo de pares G-C requerem uma temperatura superior para desnaturarem devido existncia de trs pontes de hidrognio entre as duas bases, tornando-as mais estveis que as A-T que apenas apresentam duas pontes de hidrognio nas suas ligaes. No entanto, a percentagem de bases G-C numa determinada molcula de DNA pode ser estimada atravs da temperatura de fuso da molcula, sendo que quanto maior a percentagem de pares G-C, maior a temperatura de fuso Tm.
| Captulo 1 - Genes
11
Biologia Molecular - 1 Ano - FFUP
2006/2007
11. Compactao do DNA
Nucleossomas
As protenas que se ligam ao DNA para formar o cromossoma eucaritico so, tradicionalmente, divididas em duas classes gerais: as histonas e as protenas cromossmicas no-histnicas. O complexo das duas classes de protenas com o DNA nuclear conhecido por cromatina. As histonas esto presentes em enormes quantidades nas clulas, de forma a que a sua massa total de cromatina seja quase igual massa final de DNA. As histonas so responsveis pelo primeiro e mais bsico nvel de organizao cromossmica, o nucleossoma, o qual foi descoberto em 1974. Quando o ncleo interfsico delicadamente rompido e o seu contedo examinado atravs de um microscpio electrnico, a maior parte da cromatina est na forma de uma fibra com 30 nm de dimetro. A organizao estrutural dos nucleossomas foi determinada depois de estes serem isolados, da cromatina compactada pela digesto com enzimas especficas (nucleases) que quebram o DNA cortando-o entre os cernes dos nucleossomas. Aps a digesto por um curto perodo, o DNA exposto entre as partculas dos nucleossomas, o DNA de ligao degradado. Cada partcula do cerne nucleossmico individual consiste de um complexo de oito protenas histnicas, duas molculas de cada uma das histonas H2A, H2B, H3 e H4 e a dupla cadeia de DNA, que tem 146 nucletidos de comprimento. O octmero de histonas forma, ento, um cerne proteico ao redor do qual se enrola a dupla cadeia de DNA. Cada partcula do cerne do nucleossoma separada por um segmento de DNA de ligao, o qual pode variar em comprimento desde poucos, at cerca de 80 pares de nucletidos. Este octmero de histonas , finalmente, selado por uma outra protena histnica, H1.
Digesto com a nuclase do micrococcus
Os nucleossoma so as unidades bsicas de compactao do DNA, sendo que cada um deles ligado a outro atravs de molculas de DNA linker. O DNA est protegido com protenas e passa a no ser digerido pelas enzimas de restrio ou exonucleases. Quando cada nucleossoma era constitudo por 200 pares de bases e o DNA linker tem o seu tamanho, a nuclease do micrococcus corta as molculas de DNA nas zonas onde o DNA est livre, no estando associado a protenas. Quando a enzima actua durante mais tempo, digere todo o DNA que corresponde ao DNA linker (8 a 114 pares de bases). 12 Captulo 1 - Genes |
Biologia Molecular 1 Ano - FFUP
2006/2007
Compactao da cromatina
O DNA dos eucariotas consiste em sequncias nicas e repetidas. Apenas uma percentagem inferior a 5% do DNA humano codifica protenas e RNA funcionais e as sequncias reguladoras que controlam a sua expresso. O restante maioritariamente constituinte do DNA existente entre os genes e os intres que se encontram inseridos em genes. Uma grande quantidade deste DNA (cerca de 50% nos humanos) derivado de elementos mveis de DNA que contriburam para a evoluo dos genomas contemporneos. Cada cromossoma consiste numa molcula individual e comprida de DNA com um numero de 210 Mb nos humanos, organizada em nveis sucessivamente superiores de condensao, atravs de protenas histnicas e no histnicas, com as quais se encontra altamente complexada. As molculas de DNA de dimenses inferiores encontram-se nas mitocndrias e nos cloroplastos.
| Captulo 1 - Genes
13
Biologia Molecular - 1 Ano - FFUP
2006/2007
12. Cromossoma metafsico
Um cromossoma uma molcula de DNA associada a protenas histnicas e no histnicas. constitudo por dois cromatdeos ligados pelo centrmero que se caracteriza por ser uma zona altamente compacta. Para que um cromossoma seja funcional, tem de possuir: Origem de replicao local onde se inicia a replicao do DNA; Centrmero regio especializada e complexa dos cromossomas que apresenta poucos ou nenhuns genes. o ponto de unio dos cromatdeos irmos e contm uma estrutura (o cinetocoro) a que as fibras do fuso se ligam durante a mitose e a meiose, pelo que tem um papel importante no movimento dos cromossomas em direco aos plos;
Telmero sequncias de DNA existentes nas zonas terminais dos cromossomas, que impedem um encurtamento de DNA pela aco de uma enzima especfica. O telmero pode ter o comprimento de algumas centenas de pares de bases e participa na estabilidade e na replicao do cromossoma. Um cromossoma normal possui dois telmeros. A enzima que protege os telmeros designa-se telomerase, que j no est presente na maioria das clulas adultas. Nas clulas embrionrias, existe sempre a telomerase, na medida em que esto em constante desenvolvimento.
Bandeamento dos cromossomas
Os cromossomas so submetidos a um tratamento proteoltico para depois serem colocados em contacto com reagente de Giemsa, produzindo bandas distintas em locais caractersticos. Anlises microgrficas dos cromossomas 4 e 5, mostram condies em locais onde as bandas surgem em locais levemente assinalados de forma microgrfica. Geralmente, as bandas G apresentam colorao violeta, sendo que as regies assinaladas a verde no cromossoma 4 apresentam comprimento varivel e so mais comuns na regio do centrmero. Por conveno, a regio p delimita o brao curto do cromossoma e a regio q , o brao longo. Cada um deles est dividido em seces de maiores dimenses (1, 2, etc.) e subseces. Existem ainda outros tipos de bandas de cromossomas:
14
Captulo 1 - Genes |
Biologia Molecular 1 Ano - FFUP
2006/2007
O bandeamento de cromossomas permite revelar a existncia de determinadas sequncias importantes, mas tambm se o cromossoma contm eucromatina ou heterocromatina em determinadas regies.
13. Tipos de DNA
Existem trs principais tipos de molculas de DNA: Sequncias nicas contm os genes integrados na eucromatina. Sequncias muito repetitivas encontram-se maioritariamente no centrmero e no telmero, existindo sob a forma de uma sequncia simples de DNA designada DNA satlite. Sequncias moderadamente repetitivas caractersticas dos elementos de DNA mveis. Estas sequncias terminam, geralmente, com pequenas sequncias repetidas invertidas, sendo capazes de gerar outras pequenas sequncias no local de insero. So responsveis por rearranjos do genoma e no podem viver fora dele, sendo designadas por parasitas moleculares e por selfish DNA. Podem ser de dois tipos: transposes e retrotransposes.
Sequncias moderadamente repetitivas
Depois de pesquisas sucessivas acerca destes elementos mveis, puderam ser maioritariamente criadas duas categorias: aqueles que transpem directamente o DNA e os que transpem atravs de um intermedirio de RNA, transcrito do elemento mvel por uma RNA polimerase, para depois ser convertido numa molcula de DNA de cadeia dupla por uma transcriptase reversa. Os elementos mveis que transpem atravs de um intermedirio de DNA so os transposes. Os elementos mveis que transpem para novos locais do genoma atravs de um intermedirio de RNA so os retrotransposes porque o seu movimento anlogo ao do processo infeccioso dos retrovrus. Isto acontece porque os retrovrus podem ser identificados como retrotransposes que evoluram genes atravs da codificao de capas virais, permitindo, consequentemente, a sua transposo entre as clulas.
| Captulo 1 - Genes
15
Biologia Molecular - 1 Ano - FFUP
2006/2007
Transposes a regio central relativamente comprida, que corresponde a um elemento IS, que codifica uma ou duas enzimas necessrias para a transposio, est bloqueada por uma sequncia repetitiva invertida em cada extremidade. Estas sequncias so aparentemente idnticas, mas esto orientadas em direces opostas, sendo que cada uma delas caracterstica de um elemento IS em particular. As sequncias de 5 para 3 no so transpostas com o elemento de insero, sendo consideradas como sequncias que vo ser duplicadas, com uma cpia em cada extremidade, durante a insero do elemento mvel. O comprimento destas sequncias 5- 3 constante para um determinado elemento IS mas a sua sequncia depende do locl de insero e ainda varia com cada transposio do elemento IS.
Retrotransposes podem ser divididos em vricos e no vricos: Vricos a regio central que codifica protenas est delimitada por duas sequncias terminais de grande comprimento, as sequncias LTR, que so especficas. Tal como outros elementos mveis, os retrotransposes vricos apresentam sequncias de destino bastante curtas em cada extremidade. Estes retrotransposes contribuem com cerca de 4% para a constituio do DNA humano, sendo que a replicao feita via transcriptase reversa e DNA polimerase. Para alm disso, nestes retrotransposes tambm intervm uma protease, uma Rnase H e uma integrase que permite a integrao do DNA de cadeia dupla no genoma da clula hospedeira.
No vricos contrariamente aos vricos, no possuem sequncias LTR, mobilizam-se na forma de RNA, repetem-se bastantes vezes no genoma e no esto agregados como o DNA satlite. So de dois tipos principais: LINEs (long interspersed elements) o comprimento da sequncia de destino 5- 3 varia de acordo com as cpias do elemento em diferentes locais do genoma. Ainda que a sequncia L1 apresente aproximadamente 6 kb de comprimento, quantidades variveis na extremidade esquerda esto ausentes em 90% dos locais onde este elemento mvel encontrado. A ORF (open reading frame) mais pequena, a ORF1, codifica uma protena RNA-binding protein e a maior, a ORF2, codifica uma protena bifuncional, que pode actuar tanto como transcriptase reversa ou como DNA endonuclase. Este retrotransposo no vrico forma-se a partir dos transcriptos de RNA polimerase II, sendo o mais comum, o L1
16
Captulo 1 - Genes |
Biologia Molecular 1 Ano - FFUP
2006/2007
LINE. Apresenta regies ricas em A/T e se houver um elemento L1, ento ele vai assegurar a transposio dos restantes. SINEs (short interspersed elements) constituem aproximadamente 13% do DNA humano, apresentando um comprimento compreendido entre 100 e 400 pb. Estes retrotransposes no codificam protenas, mas a maior parte deles contm uma sequncia rica em A/T na extremidade 3, tal como os LINEs. Os SINEs so transcritos pela RNA polimerase III, a mesma que transcreve genes que codificam tRNAs e outras pequenas categorias de RNAs. De todos os locais do genoma huamno em que existem SINEs, uma grande quantidade so os Alu que adquirem esta designao pois contm um nico sinal de reconhecimento para a enzima de restrio Alu-I. Os elementos Alu apresentam uma homologia considervel com o 7SL RNA que um componente de uma partcula de reconhecimento sinal (SRP).
Transposio mediada por DNA Elementos IS da bactria - cerca de 50bp nas repeties invertidas a ladear a transposase; - fazem a exciso ou copiam o DNA e a insero no local alvo; - IS1 e IS10. Transposes bacterianos - contm gene que confere resistncia a antibiticos entre os elementos IS; - copiam o DNA e a insero do local alvo; - Tn9. Transposes eucariotas - tm repeties invertidas a ladear a regio codificante que contm intres; - exciso do DNA e insero no local alvo; - elementos P (Drosophila) e elementos Ac e DS (milho).
Transposio mediada por RNA Retrotransposes vricos - cerca de 250 a 600bp nas sequncias repetidas directas (LTR), que ladeiam a transcriptase reversa, integrase e protenas retrovricas tipo Gag; - saltam via intermedirio de RNA feito a partir da RNA polimerase II, que se liga ao do promotor de LTR esquerdo, e inserem-se no local alvo na forma de DNA de cadeia dupla (transcriptase reversa); - elemento Ty (levedura) e elemento copia (Drosophila).
Retrotransposes no vricos - comprimento varivel com regies ricas em A/T, codificam para a transcriptase reversa; - transcrio para RNA a partir de promotor interno, transcriptase reversa forma DNA de cadeia dupla e insero no local alvo; - elementos F e G (Drosophila), elementos LINEs e SINEs (mamferos), sequncia Alu (humanos).
| Captulo 1 - Genes
17
Biologia Molecular - 1 Ano - FFUP
2006/2007
Captulo 2 Cdigo gentico
O cdigo gentico o conjunto de regras atravs das quais a informao codificada no material gentico (sequncias da DNA ou RNA) traduzida para protenas (sequncias de aminocidos) por clulas vivas. Mais especificamente, o cdigo gentico define uma correspondncia entre codes e aminocidos: cada tripleto de nucletidos numa sequncia de cido nucleico especifica apenas um aminocido.
1. Caractersticas do cdigo gentico
Degenerado O cdigo altamente degenerado na medida em que mais do que um codo pode especificar o mesmo aminocido (excepo feita metionina e ao triptofano, que so ambos especificados por um nico codo). Os diferentes codes que especificam o mesmo aminocido so designados por sinnimos. Devido degenerescncia do cdigo, podem ocorrer vrias alteraes num gene (mutaes) que no tero qualquer efeito na composio do produto gnico (mutaes silenciosas ou mutaes com o mesmo sentido). Universal O cdigo gentico parecia inicialmente aplicar-se a todos os genes, quer em procariotas, quer em eucariotas. Foi, portanto, considerado um cdigo universal. Contudo, mais tarde, foram encontradas algumas excepes no DNA mitocondrial de alguns organismos, que sero desenvolvidas em 2.4. No tem vrgulas O cdigo gentico lido seguido, na totalidade, desde o codo de iniciao at um dos codes de terminao. Codo especfico de iniciao A iniciao da traduo d-se sempre com o codo AUG e com um tRNA iniciador que transporta o aminocido metionina (logo, todas as protenas vo ter a metionina como primeiro aminocido). Codes especficos de terminao Existem trs codes que no so reconhecidos por nenhum tRNA: UAA (ochre), UAG (amber) e UGA (opal). So designados por codes sem significado, codes de terminao ou codes de paragem, porque actuam como o ponto final no fim duma frase, isto , constituem parte do sinal de que a sntese da protena dever parar nesse ponto. 64 codes 61 deles codificantes, 3 deles codes de terminao 20 aminocidos e 4 nucletidos so necessrios trs grupos de quatro bases diferentes para se constiturem os 20 aminocidos existentes.
18
Captulo 2 Cdigo gentico |
Biologia Molecular 1 Ano - FFUP
2006/2007
2. Constituio do cdigo gentico
8 famlias de codes em que os dois primeiros nucletidos so idnticos e tm o mesmo significado e o terceiro no tem qualquer significado na especificao do aminocido. 7 pares de codes que representam o mesmo aminocido independentemente do terceiro nucletido poder ser qualquer uma das pirimidinas. 5 pares de codes em que qualquer purina pode preencher o terceiro nucletido sem que haja alterao do aminocido codificado. 3 codes cujo significado determinado pela presena de uma base particular na terceira posio: AUG (met), UGG (trp) e UGA (stop).
3. Conceito de wobble
Se o emparelhamento de bases perfeito de Watson-Crick fosse exigido entre codes e anticodes, as clulas conteriam exactamente 61 espcies diferentes de tRNA, uma para cada codo que especificasse um aminocido. Contudo, muitas clulas contm menos de 61 tRNAs (tm, na realidade, 32). A explicao para o pequeno nmero assenta na capacidade de um nico anticodo de tRNA reconhecer mais do que um (mas no necessariamente todos) codo correspondente a um dado aminocido. Este reconhecimento pode ocorrer devido ao emparelhamento no regular entre bases na que conhecida por posio wobble (terceira base do codo de mRNA, correspondendo primeira no seu anticodo). A primeira e segunda bases do codo formam, normalmente, pares de bases Watson-Crick com a terceira e segunda do anticodo correspondente, respectivamente.
| Captulo 2 Cdigo gentico
19
Biologia Molecular - 1 Ano - FFUP
2006/2007
Particularmente importante o par das bases G-U, que estruturalmente adapta-se to bem como o par G-C. Portanto, um dado anticodo em tRNA com G na primeira posio (wobble) pode corresponder aos dois codes que tenham qualquer pirimidina na terceira posio, desde que as outras duas correspondam. Apesar de a adenina raramente ser encontrada na posio wobble do anticodo, muitos tRNAs de plantas e animais contm inosina (I), um produto desaminado da adenina, nessa posio. Esta pode formar pares no regulares com A, C e U. Assim, um tRNA com inosina na posio wobble pode reconhecer os codes correspondentes de mRNA com A, C ou U na terceira posio (wobble). Por esta razo, tRNAs contendo inosina so largamente utilizados na traduo de codes sinnimos que especificam um nico aminocido.
4. Cdigo gentico mitocondrial
O cdigo gentico usado nas mitocndrias diferente do cdigo padro usado em genes nucleares procariticos e eucariticos, diferindo, at dentro dos vrios tipos de mitocndrias.
As principais alteraes so: Metionina iniciadora codificada por AUG, AUA, AUU e AUC Metionina interna codificada por AUG e AUA Codes de terminao UAA, UAG, AGA, AGG Os tRNAs tm estruturas diferentes, nomeadamente na haste D e T Existem 22 tRNAs Todos os tRNAs tm na posio wobble um resduo de U que pode emparelhar com qualquer outra das quatro bases passveis de estar na terceira posio no codo correspondente.
20
Captulo 2 Cdigo gentico |
Biologia Molecular 1 Ano - FFUP
2006/2007
Captulo 3 Replicao do DNA
A replicao do DNA genmico um processo de grande complexidade que assegura ao organismo a passagem das suas caractersticas especficas descendncia atravs da replicao do material gentico. Este processo est intimamente ligado ao ciclo celular e alvo de uma estrita regulao, de modo a assegurar que cada clula se divide s o nmero de vezes necessrio ao desenvolvimento e crescimento do organismo.
1. Dogma central da Biologia Molecular
2. Ciclo celular e replicao
O tipo mais simples de reproduo envolve a diviso de uma clula-me em duas clulas-filhas, Isto ocorre como parte do ciclo celular, uma srie de eventos que preparam a clula para se dividir, seguido da prpria diviso, chamada mitose. O ciclo da clula eucaritica normalmente representado em 4 fases. Os cromossomas e o DNA que contm so copiados durante a fase S (sntese). Os cromossomas replicados separam-se durante a fase M (mittica), com cada uma das clulas-filhas a receber uma cpia de cada cromossoma durante a diviso da clula. As fases M e S so separadas por duas fases G, a fase G1 e a fase G2, durante as quais os mRNAs e as protenas so sintetizados.
| Captulo 3 Replicao do DNA
21
Biologia Molecular - 1 Ano - FFUP
2006/2007
3. Caractersticas da replicao
3.1. Semi-conservativa
Cada cadeia parental serve de modelo para a sntese de uma cadeia filha que lhe complementar, processo que culmina com a obteno de duas molculas filha idnticas ao duplex inicial.
3.2. ARS
Qualquer segmento de DNA que tenha uma origem deve conseguir replicar-se. Ento, apesar de os plasmdeos serem raros nos eucariotas, pode ser possvel constru-los a partir de manipulao in vitro. At agora foi conseguido nas leveduras. Um fragmento transformante de alta frequncia possui uma sequncia que confere a capacidade para replicar eficientemente nas leveduras, sequncia essa denominada ARS (autonomously replicating sequence), que deriva de origens de replicao.
3.3. Replicao bidireccional e unidireccional
A origem pode ser usada para se iniciar replicao unidireccional ou bidireccional. O tipo de evento determinado determinado pela quantidade (um ou dois) de forquilhas de replicao que partem da origem. Na replicao unidireccional, uma forquilha de replicao parte da origem e continua ao longo do DNA. Na bidireccional, duas forquilhas de replicao so formadas e continuam a partir da origem em direco opostas.
4. Replices
Replico uma molcula de DNA ou RNA, ou uma regio no DNA ou RNA que replica a partir de uma nica origem de replicao. o local onde se d um acto individual de replicao. O replico definido pela sua posse no controlo de elementos necessrios replicao, e tem uma origem na qual a replicao comea. Pode tambm ter um local de terminao, onde a replicao pra. Para a maior parte dos cromossomas procariotas, o replico um cromossoma completo. Plasmdeos e bacterifagos tambm so replicados como replices nicos. 22 Captulo 3 Replicao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
Para os cromossomas eucariotas, h mltiplos replices por cromossoma.
5. Protenas necessrias replicao
| Captulo 3 Replicao do DNA
23
Biologia Molecular - 1 Ano - FFUP
2006/2007
6. Replicao em procariotas
6.1.
Incio da replicao
Nos procariotas, a iniciao da replicao o nico evento, envolvendo um nico local no cromossoma bacteriano, e o processo de diviso conseguido atravs do desenvolvimento de um septo que cresce a partir da clula e a divide em dois. Antes da replicao, o local-alvo palindromtico metilado nas adeninas de cada cadeia. A replicao insere as bases normais nas cadeias-filhas, dando origem a DNA hemimetilado, no qual s uma cadeia metilada. Ento, a replicao converte locais-alvo da Dam metilase de totalmente metilados para a condio de hemimetilados. Qual a consequncia para a replicao? A capacidade de um plasmdeo poder replicar a partir do oriC depende do estado de metilao. Se o plasmdeo est metilado, inicia um ciclo de replicao, e quando est hemimetilado os produtos acumulam-se, da que uma regio hemimetilada no possa ser usada para iniciar um ciclo de replicao.
24
Captulo 3 Replicao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
A replicao do DNA em E. coli bidirecional e originada numa nica origem do replicao (OriC). A iniciao da replicao mediada pela DnaA, uma protena que se liga a uma regio da origem conhecida como a DnaA box. Na E. coli, h 5 DnaA boxes, cada qual contm uma sequncia altamente conservada 5 ' TTATCCACA - 3 ' do consenso 9bp. A ligao da DnaA a esta regio faz com que se torne negativamente superenrolada. Depois desta, uma regio de OriC acima das DnaA boxes separa-se. H trs destas regies, e cada uma tem o comprimento de 13bp, e so ricas em AT (que facilita obviamente a quebra, porque requerida menos energia para quebrar as duas ligaes do hidrognio que se formam entre nucletidos de A e de T). Esta regio tem a sequncia 5 ' - GATCTNTTNTTTT - 3 do consenso. A quebra das DnaB boxes requer ATP (que hidrolisado pela DnaA). Depois da quebra, a DnaA recruta um hexmero de helicases (seis protenas de DnaB) s extremidades opostas do DNA quebrado. Este o local onde a forquilha de replicao se formar. O recrutamento da helicase requer seis protenas de DnaC, cada uma unida a uma subunidade da helicase. Uma vez que este complexo formado, cinco protenas adicionais de DnaA ligamse s cinco protenas originais de DnaA para formar cinco dmeros de DnaA. A DnaC , ento, liberta e o complexo prepriming est completo. Para que a replicao continue, necessrio SSB para impedir que as duas cadeias de DNA originem estruturas secundrias e se religuem, e DNA girase para aliviar o stress (criando superenrolamentos negativos) criado pela aco da helicase de DnaB. O desenrolar do DNA pela helicase de DnaB permite que a primase (DnaG) e a polimerase do RNA preparem cada molde do DNA de modo que a sntese do DNA possa comear.
6.2.
Protenas e enzimas requeridas
| Captulo 3 Replicao do DNA
25
Biologia Molecular - 1 Ano - FFUP
2006/2007
Origem de replicao activa sequncia particular de DNA na qual a replicao iniciada. A partir deste ponto a replicao pode proceder-se unidireccionalmente ou bidireccionalmente. Helicases II e III (5-3) e Rep (3-5) - quebram as pontes de hidrognio entre bases complementares das duas cadeias. Protenas que se ligam ao DNA: SSB (ligam-se cadeia de modo a que no se restabelea a dupla hlice, enquanto as outras enzimas no esto ainda a actuar), DnaA (factor de iniciao da replicao que promove a desnaturao do DNA no local de replicao activa) e DnaC (protena reguladora da DnaB). Primer ou iniciador - sequncia de bases de RNA que vo iniciar a sntese. Primases (forma de RNA polimerase que se liga DNA helicase, formando o primossoma) e protenas associadas: DnaB (enzima que abre a forquilha de replicao durante a replicao), DnaT, DnaC, priA, priB, priC DNA polimerases I, II e III I, de 5 para 3 apresenta actividade de DNA polimerase, de 3para 5 apresenta actividade de exonuclease, estando envolvida na correco e reparao de erros, sintetiza nos locais em que a RNase H removeu o primer; II est envolvida na reparao de erros de 5 para 3 e tem a funo de exonuclease de 3 para 5; holoenzima DNA polimerase III o principal complexo enzimtico envolvido na replicao de DNA procaritico e trabalha em conjunto com outras DNA polimerases, reparando erros, sintetiza a partir do primer. Ribonuclease H (RNase H) endonuclease no especfica que cataliza a quebra a ligao 3-O-P do RNA, transformando num duplex DNA/RNA para levar a produtos 3-hidroxilados e 5-fosfatados. Topoisomerases I e II (girase) I hidrolisa uma cadeia de cada vez, II hidrolisa duas cadeias de cada vez. Removem (decatenizao) ou induzem (catenizao) o superenrolamento da cadeia cortando-a em locais estratgicos. DNA ligase liga os nucletidos de modo a formar-se uma cadeia.
6.3.
Replissoma
O replissoma constitudo por duas DNA polimerase III que, por sua vez, so constitudas por trs subunidades: uma com actividade de polimerizao, uma com capacidade de detectar erros e uma que estimula a procura de erros. O objectivo destas subunidades ou protenas replicar o genoma do organismo correcta e rapidamente.
6.4.
Enzima Klenow
A enzima Klenow o fragmento maior (C-terminal) da DNA polimerase I. Apresenta actividade de polimerase 5-3 e actividade de exonuclease 3-5, removendo nucletidos pr-codificados.
26
Captulo 3 Replicao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
usada em laboratrio para sntese de DNA in vitro por um processo designado por nick translation (com incorporao de nucletidos radioactivos).
6.5.
Sntese do DNA
Devido ao antiparalelismo da cadeia de DNA parental, das duas cadeias filhas a sintetizar, s um a poder ser feita de modo contnuo na direco 5 para 3 a partir da regio da cadeia principal imediatamente adjacente origem de replicao esta ser a cadeia avanada (cadeia contnua ou leading). A outra cadeia filha no poder ser sintetizada de forma contnua, pois estar condicionada pelo facto da DNA polimerase ter um a nica direco de sntese (de 5 para 3). Assim, esta cadeia atrasada (cadeia descontnua ou lagging) ir ser sintetizada na direco oposta ao avano da forquilha de replicao, atravs da sntese e posterior ligao de mltiplos segmentos de DNA, todos iniciados por um pequeno fragmento de RNA iniciador colocado pela primase os fragmentos de Okazaki. O processo de juno de dois fragmentos de Okazaki implica a remoo do RNA iniciador existente no fragmento de Okazaki a partir da sua extremidade 5 por uma enzima do tipo RNAse com actividade exonuclesica 5-3. Ao mesmo tempo, para preencher esse espao, so adicionados novos nucletidos na extremidade 3 do fragmento de DNA que lhe fica adjacente, com a ajuda de uma das DNA polimerases que constituem o complexo de replicao. Os dois fragmentos de DNA so finalmente ligados um ao outro pela DNA ligase, que estabelece a ligao fosfodiester final entre o grupo 3-OH do ltimo nucletido do primeiro fragmento de Okazaki e o alfa-P da extremidade 5 do fragmento de Okazaki adjacente que acabou de ser sintetizado. De modo a aliviar a tenso de toro das cadeias durante o seu desenrolar pela helicase, as topoisomerases vo igualmente actuar neste processo.
| Captulo 3 Replicao do DNA
27
Biologia Molecular - 1 Ano - FFUP
2006/2007
7. Replicao em eucariotas
7.1.
Protenas e enzimas requeridas
Helicases - quebram as pontes de hidrognio entre bases complementares das 2 cadeias. Protenas que reconhecem os locais de origem e que fazem parte do complexo de pr-replicao Protenas que se ligam cadeia simples de DNA (RPA replication protein A) Primases - sintetizam uma pequena molcula de RNA (primer). Exonuclease MF1 remove o primer. DNA polimerases eucariotas
7.2.
DNA polimerases
Localizao Replicao Reparao Funes: 5-3 polimerase 3-5 exonuclease 5-3 exonuclease Primase Associada com PCNA2 Prossessivity Sntese de cadeias
1
Nucl. no sim
Mit. sim no
Nucl. sim no
Nucl. (no) sim
3
Nucl. Sim No
sim No No sim No Baixa Lagging Leading
sim No No No No
sim sim No No No
sim sim No No Sim Alta Leading Lagging
Sim sim No No No
actividade presente nas protenas associadas Proliferanting Cell Nuclear Antigen envolvida na reparao relacionada com a transcrio
28
Captulo 3 Replicao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
7.3.
Protenas da forquilha de replicao em humanos
Protenas SSB (single stranded binding proteins) ligam-se cadeia de modo a que no se restabelea a dupla hlice, enquanto as outras enzimas no esto ainda a actuar. PCNA desloca a DNA polimerase alfa e permite a ligao da DNA polimerase delta nas duas cadeias. RFC (replication factor C) estimula a actividade de DNA polimerase alfa
7.4.
Incio da replicao
ORC (origin-recognition complex) determina onde ocorre o incio de replicao. Requer as protenas ORC2-6 e ORC1.
1 Durante o incio da fase G1, os factores de iniciao de replicao desfoforilados fazem com que o ORC se ligue a uma origem de replicao para gerar um complexo de prreplicao. 2 na fase S, as CDKs da fase S complexam e o DDK fosforila componentes do complexo de pr-replicao? 3 Isto faz com que o Cdc45 se ligue, activao das helicases Mcm, que desenrolam as cadeias de DNA e libertao de Cdc6 fosforilado e factores de iniciao Ctd1. RPA liga-se s cadeias simples resultantes. 4 Um complexo de DNA polimerase Pol e primase inicia a sntese das cadeias-filhas. 5 DNA polymerase e os seus factores acessrios (PCNA e Rfc) elongam as cadeias filhas iniciadas pela Pol--primase. ORC liga-se sequncia de origem na cadeia dupla de DNA resultante, mas os factores de iniciao fosforilados no podem montar um complexo de pr-replicao no local. Complexos da ciclina CDK do tipo B mantm os factores de iniciao num estado fosforilado durante a fase S e G2 e no incio da anafase. Estes factores no podem iniciar novos complexos at que as ciclinas sejam degradadas.
| Captulo 3 Replicao do DNA
29
Biologia Molecular - 1 Ano - FFUP
2006/2007
7.5.
Telmeros:
Telmeros e telomerase
Parte terminal dos cromossomas Mantm a estabilidade dos cromossomas Repeties em srie de 5-26 bps O tamanho varia muito, mas controlado de forma especfica Implicados no envelhecimento, cancro e variao antignica
Telomerase: necessria para a manuteno do tamanho dos telmeros da linha germinativa Actividade de transcriptase reversa Clulas somticas no tm telomerase Clilas germinativas e embries tm telomerase Stem cells e clulas cancergenas tm telomerase, o que pode explicar a capacidade de diviso contnua
8. Defeitos na replicao e reparao do DNA
Esto todas associadas a uma grande frequncia de mutaes gnicas e cromossmicas, a maior parte est tambm associada a predisposio para cancro (especialmente leucemia).
Sndrome de Werner - provocado por mutaes na DNA polimerase Ataxia telangiectasia - maior risco de raios-X, associado ao aumento de cancro da mama em grvidas Sndrome de Cockayne - provocado por um defeito na reparao de DNA associada transcrio, causa sensibilidade luz solar, mas no predispe ao cancro Sndrome de Bloom - provocado por mutaes no gene da helicase, induz um maior risco de raiosX e uma maior sensibilidade luz solar Pigmentose Xeroderma - provocado por mutaes em genes relacionados com a reparao de cortes de nucletidos, estando associada a um grande aumento de cancro induzido pela luz solar e com outros tipos de cancro, como o melanoma. Anemia Fanconi causa sensibilidade luz solar e maior risco de raios-X
30
Captulo 3 Replicao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
9. Tipos de replicao
9.1.
Replicao tipo olho
Ocorre na bactria Ao M.E. forma uma estrutura tipo
9.2.
Replicao em crculos rolantes
Ocorre no rDNA de ocitos de Xenopus e em alguns bacterifagos Corte na origem de replicao numa das cadeias do DNA A cadeia deslocada serve de molde para a sntese da cadeia complementar atravs de fragmentos de Okazaki
9.3.
Replicao atravs de formas replicativas
Ocorre em certos vrus de cadeia de DNA simples (M13, 174) Cadeia de DNA (+) serve de molde para sintetizar a cadeia de DNA (-) DNA de cadeia dupla (RF I) Protena A (codificada pelo genoma do vrus) corta o DNA no local de origem de replicao RF II Protena Rep = helicase
| Captulo 3 Replicao do DNA
31
Biologia Molecular - 1 Ano - FFUP
2006/2007
9.4.
Replicao tipo D-loop
Ocorre na mitocndria H dois locais de origem de replicao No h formao de fragmentos de Okazaki Sntese comea no local de origem da cadeia H Sntese na cadeia L comea quando cerca de 2/3 da cadeia H j replicou Alteraes ou mutaes neste tipo de replicao conduzem a citopatias mitocondriais.
32
Captulo 4 Transcrio |
Biologia Molecular 1 Ano - FFUP
2006/2007
Captulo 4 Transcrio
1. Sntese do RNA
A transcrio comea com a abertura e despiralizao de uma pequena poro da dupla hlice de DNA, para expr as bases em cada cadeia de DNA. Uma das duas cadeias da dupla hlice de DNA age, ento, como um molde para a sntese de uma molcula de RNA. Tal como no processo de replicao do DNA, a sequncia de nucletidos da cadeia de RNA determinada pela complementaridade do emparelhamento de bases entre os nucletidos a serem incorporados e o DNA-molde. A cadeia de RNA produzida por transcrio o transcrito , portanto, aumentanda num nucletido por processo e possui uma sequncia de nucletidos exactamente complementar cadeia de DNA utilizada como molde.
1.1. Diferenas entre DNA e RNA
O primeiro passo executado pela clula para ler a informao necessria das suas instrues genticas a cpia de uma parcela especfica da sequncia de nucletidos de DNA um gene sob a forma de uma sequncia de nucletidos de RNA. A informao na forma de RNA, embora copiada de uma forma quimicamente distinta, tambm escrita essencialmente na mesma linguagem do DNA, ou seja, a linguagem de uma sequncia de nucletidos, de onde deriva a designao do processo de transcrio. Tal como o DNA, o RNA um polmero linear composto por quatro tipos diferentes de subunidades nucleotdicas unidas entre si por uma ligao fosfodister. O RNA difere quimicamente do DNA em dois aspectos principais: Os nucletidos do RNA so ribonucletidos, isto , contm o acar ribose em vez de desoxirribose; Embora, assim como o DNA, o RNA contenha as bases adenina, guanina e citosina, ele contm a base uracilo (U), em vez da timina, que ocorre no DNA. Uma vez que o uracilo, tal como a timina, pode formar pares com estabelecimento de pontes de hidrognio com a adenina, as propriedades de complementaridade por emparelhamento de bases descritas para o DNA anteriormente, tambm se aplicam ao RNA. No entanto, no raro encontrar outros tipos de emparelhamento de bases no RNA, como por exemplo a guanina a emparelhar com o uracilo, ocasionalmente.
| Captulo 4 Transcrio
33
Biologia Molecular - 1 Ano - FFUP
2006/2007
Apesar destas pequenas diferenas qumicas, o DNA e o RNA diferem drasticamente nas suas estruturas como um todo. Enquanto o DNA ocorre sempre nas clulas como uma cadeia de dupla hlice, o RNA apresenta-se com uma cadeia simples. Portanto, as cadeias de RNA podem dobrar-se sob diversas formas, do mesmo modo que uma cadeia de polipptidos pode dobrar-se, resultando na conformao final de uma protena.
1.2. Enzimas que realizam transcrio
As enzimas que realizam a transcrio so denominadas de RNA polimerases. Assim como a DNA polimerase catalisa a replicao do DNA, as RNA polimerases catalisam a formao de pontes fosfodister que ligam os nucletidos entre si para formar uma cadeia linear. A libertao quase imediata da cadeia de RNA do DNA, conforme a primeira est a ser sintetizada, significa que muitas cpias de RNA podem ser produzidas a partir do mesmo gene num perodo de tempo relativamente pequeno; a sntese de molculas de RNA adicionais pode ser iniciada antes que a do primeiro RNA tenha terminado. Quando vrias molculas de RNA polimerase usam a mesma regio como molde, deixando um pequeno intervalo entre si, cada uma sintetizando aproximadamente 20 nucletidos por segundo, podem ser sintetizados mais de mil transcritos numa hora, a partir de um nico gene. Apesar de a RNA poliermase catalisar essencialmente a mesma reaco qumica que a DNA polimerase, existem algumas diferenas importantes entre estas duas enzimas: A RNA polimerase catalisa a ligao de ribonucletidos e no de desoxirribonucletidos como a DNA polimerase. Ao contrrio das DNA polimerases envolvidas na replicao de DNA, as RNA polimerases podem comear a sintetizar uma cadeia de RNA sem um iniciador. Isto pode acontecer porque a transcrio no precisa de ser to precisa quanto a replicao. O RNA no mantm permanentemente a informao gentica nas clulas. As RNA polimerases realizam aproximadamente um erro em cada 104 nucletidos copiados no RNA, enquanto que a DNA polimerase comete um em cada 107 nucletidos. As consequncias de um erro na transcrio do RNA so muito menos significativas do que aquelas que ocorrem na replicao do DNA.
34
Captulo 4 Transcrio |
Biologia Molecular 1 Ano - FFUP
2006/2007
1.3. Tipos de RNA
A maioria dos genes transportados no DNA das clulas especifica a sequncia de aminocidos de protenas: RNA mensageiro (mRNA) molculas de RNA que so copiadas a partir desses genes de DNA que especificam a sequncia de aminocidos das protenas. RNA de transferncia (tRNA) formam os adaptadores que seleccionam aminocidos e os colocam no local adequado dos ribossomas para serem incorporados em protenas. RNA ribossmico (rRNA) formam o cerne dos ribossomas. RNA nuclear (snRNA) direccionam o splicing do pr-RNA para formar o mRNA. RNA nucleolar (snoRNA) utilizados para processar e modificar quimicamente os rRNAs. Outros RNA no codificantes actuam em diversos processos celulares, incluindo a sntese de telmeros, a inactivao do cromossoma X e o transporte de protenas para o retculo endoplasmtico.
1.4. Hibridizao
Quando uma soluo aquosa de DNA aquecida at 100C ou exposta a um pH muito alto (pH 13), a complementaridade de bases, que normalmente mantm as duas cadeias da dupla hlice unidas, rompida, e a dupla hlice dissocia-se rapidamente em duas cadeias simples. Este processo, designado de desnaturao de DNA, foi considerado irreversvel por vrios anos. Em 1961, entretanto, foi descoberto que cadeias simples complementares de DNA reconstituam prontamente duplas hlices atravs de um processo designado hibridizao ou renaturao de RNA, se fossem mantidas por um perodo prolongado de tempo a 65 C. Podem ocorrer reaces similares de hibridizao entre quaisquer duas fitas simples de cadeias de cidos nucleicos (DNA/DNA, RNA/RNA ou DNA/RNA), desde que tenham sequncias de nucletidos complementares. Hibridizao DNA-RNA cada transcrito de RNA complementar para uma nica cadeia da molcula parental de DNA. Cada uma das cadeias simples de DNA transcrita de forma assimtrica, ou seja, apenas uma cadeia transcrita num local em particular. Desta forma, a direco de transcrio diferente e oposta nas duas cadeias simples de DNA.
| Captulo 4 Transcrio
35
Biologia Molecular - 1 Ano - FFUP
2006/2007
Hibridizao RNA-RNA processo maioritariamente utilizado em mecansimos de terapia gnica. Depois de ocorrer a transcrio da molcula de DNA e se ter formado a molcula de mRNA subsequente, este colocado em conjunto com uma molcula de RNA anti-sense. Com isto, a molcula de RNA final com as duas cadeias sense e anti-sense no vai ser traduzida e deixa de originar protenas que realizam processos celulares normais.
2. Transcrio nos procariotas
2.1. RNA polimerase bacteriana
Nas clulas procariotas, a transcrio dos RNA celulares, quer sejam ribossomais (rRNA), mensageiros (mRNA), de transferncia (tRNA) ou outros, processa-se sob catlise de uma nica RNA polimerase. A RNA polimerase bacteriana apresenta uma estrutura oligomrica, em que cada uma das subunidades constituintes confere propriedades bioqumicas especficas, inerentes complexidade da funo desempenhada pela enzima. A holoenzima da E. Coli formada por cinco subunidades, constitudas por entidades proteicas diferentes, designadas , , e , que na enzima activa se apresentam organizadas num complexo constitudo por duas cadeias , uma cadeia , uma cadeia e uma cadeia . A core enzyme, de estrutura 2, tem a propriedade de catalizar a elongao das cadeias de RNA, mas depende da subunidade para poder iniciar a transcrio. A iniciao da transcrio condicionada pela ligao do complexo aos stios especficos do DNA, que correspondem ao incio de cada gene, inscrito no genoma celular sem solues de continuidade fsica. Na RNA polimerase da E. Coli: A cadeia responsvel pela fixao dos nuclesidos trifosfato da ribose, precursores do RNA, propriedade experimentalmente demonstrada mediante utilizao de anlogos radioactivos destes substratos. A cadeia apresenta carcter bsico acentuado, e determina a ligaao do complexo enzimtico ao DNA molde, que como se sabe tem carcter cido. O factor determina o reconhecimento da regio promotora de cada gene, sendo portanto essencial iniciao correcta da transcrio dos genes bacterianos. A ligao do factor modifica a conformao da core enzyme, conferindo-lhe a capacidade de reconhecer o DNA quele nvel especfico. O factor da RNA polimerase DNA dependente que actua normalmente na transcrio de todos os genes bacterianos designado por 70, devido sua massa molecular de 70 kDa. 36 Captulo 4 Transcrio |
Biologia Molecular 1 Ano - FFUP
2006/2007
A subunidade intervm tambm no reconhecimento dos promotores, como indica o facto de alteraes estruturais desta subunidade resultar na diminuio da afinidade da holoenzima para os elementos promotores do DNA.
2.2.
Regio promotora
Os elementos promotores da transcrio ou cis-acting elements de cada gene activo, que correspondem ao stio de ligao da RNA polimerase ao DNA genmico da clula, podem ser identificados pelo mtodo de footprinting. A anlise de um nmero elevado de genes procariticos, e a caracterizao dos respectivos promotores revelou a existncia de motivos estruturais comuns, localizados a montante do stio de iniciao da transcrio, que foram designados consensus, elementos cannicos ou elementos de consenso. Foram encontrados elementos constitudos por seis pares de bases, com predomnio dos resduos A e T, e por isso designados por TATA box. Estes elementos promotores, tambm conhecidos por sequncia de Pribnow, situam-se de modo geral na posio -10 dos genes bacterianos. Um outro elemento de consenso designado por sequncia de reconhecimento, situando-se na posio -35. Constata-se que os promotores fortes de E. Coli, mais eficazes por permitirem uma frequncia elevada da transcrio do gene adjacente, so constitudos por sequncias de estrutura muito prximas da dos elementos cannicos. Em contrapartida, os promotores fracos, que determinam a iniciao da transcrio de um gene a taxas muito inferiores, apresentam substituies de nucletidos a nvel das sequncias localizadas a -10 e a -35. Este facto demonstra-se por experincias de mutagnese dirigida, em que so introduzidas, in vitro, modificaes a nvel de uma ou de vrias bases dos elementos de consenso da regio promotora, quer na sequncia -10, quer na sequncia -35. A substituio de uma nica base pode produzir uma baixa importante, ou mesmo uma perda completa, da actividade promotora do elemento.
| Captulo 4 Transcrio
37
Biologia Molecular - 1 Ano - FFUP
2006/2007
2.3.
Footprinting
O ensaio de footprinting consiste em submeter o fragmento de DNA, que contm a regio promotora do gene, - previamente marcado a nvel de uma extremidade de uma das cadeias e ligado enzima , a uma hidrlise controlada. Da resulta o corte de todas as ligaes fosfodister acessveis, logo que exclui os segmentos de DNA que se encontram protegidos pela ligao RNA polimerase. A ausncia de cortes nesta regio traduz-se na ausncia de bandas correspondentes aos nucletidos do segmento no hidrolisado, na autorradiografia, definindo desta forma a marca da protena sobre o DNA. Os resultados dos ensaios de footprinting, a par da sequenciao dos segmentos de DNA, permitem determinar a posio e a estrutura nucleotdica do stio de fixao da protena ao DNA. Estes mtodos experimentais permitem tambm demonstrar as diferenas entre o papel de cada uma das duas cadeias de DNA no processo, em que apenas uma dela se encontra protegida pela polimerase, facto perfeitamente compatvel com a assimetria da transcrio do DNA. Atravs desta tcnica, foi possvel observar-se que o stio de fixao protena/DNA corresponde aproximadamente regio entre -50 e +20, facto ainda comprovado atravs de estudos de proteco do DNA, que delimita a regio promotora dos genes bacterianos, ao segmento delimitado pelas posies -44 a +20.
38
Captulo 4 Transcrio |
Biologia Molecular 1 Ano - FFUP
2006/2007
1 amostras incubadas separadamente, na presena e na ausncia de RNA polimerase. (ou outra protena de ligao ao DNA). 2 hidrlise pela DNase, em condies controladas, de modo a assegurar o corte sequencial da cadeia. O segmento de DNA que se encontra ligado protena protegido da degradao pela nuclease. 3 desnaturao dos complexos DNA/protena. 4 anlise dos fragmentos de DNA produzidos em 2, por electroforese seguida de autorradiografia. No gel de sequenciao, observa-se a ausncia do segmento de DNA protegido pela protena, na amostra (pista da esquerda). A respectiva sequncia nucleotdica lida na pista da direita.
2.4.
Iniciao da transcrio
A iniciao da transcrio implica a identificao da regio promotora do DNA molde de cadeia dupla, e a formao do complexo de pr-iniciao com a polimerase. o factor que modifica significativamente a afinidade da RNA polimerase para o DNA, reconhecendo especificamente os promotores dos genes potencialmente activos para a transcrio. Nas bactrias existe o factor major que designado vegetativo, de que so exemplo o 70 de E. Coli ou o 43 de B. Subtilis, a par de uma srie de outros factores especializados. As subunidades conferem holoenzima a propriedade de reconhecer os stios de ligao a nvel do incio dos genes, aos quais se fixam com maior ou menor afinidade, na dependncia da estrutura nucleotdica dos elementos promotores e da identidade do factor em presena. A fixao da RNA polimerase multimrica rpida, pois que a deteno dos promotores dos diferentes genes se d sem que seja necessria a abertura da dupla hlice do DNA. Subsequentemente, e devido desnaturao pontual induzida no DNA, a enzima ligada desloca-se ao longo do DNA molde, sem ter que se fixar e libertar a cada passo. O nucletido que marca o stio de iniciao da transcrio corresponde posio +1 do gene respectivo, designando-se os nucletidos situados imediatamente a montante (5) pelos nmeros -1, -2, etc., e os resduos a jusante (3) no DNA do locus, nucletidos +2, +3, etc.
| Captulo 4 Transcrio
39
Biologia Molecular - 1 Ano - FFUP
2006/2007
2.5.
Elongao da transcrio
O complexo binrio constitudo pela holoenzima ligada ao promotor do gene converte-se rapidamente aps formao da primeira ligao fosfodister, no complexo ternrio DNA-enzima-cadeia nascente de RNA. Seguidamente, o factor liberta-se do complexo, e a core enzyme que prossegue a polimerizao e a elongao da cadeia de RNA nascente. A libertao do factor faz diminuir a estabilidade do complexo de iniciao, ao mesmo tempo que permite o deslizamento da core enzyme sobre a cadeia molde do DNA, atravs de um mecanismo em que a regio aberta da dupla hlice acompanha esta deslocao. Durante este processo, a enzima ocupa um segmento de cerca de 70 pares de bases de DNA, mas apenas numa extenso de 17 pares de bases se d a desnaturao pontual da hlice. Por outro lado, o hbrido corresponde ao segmento da cadeia molde de DNA e ao RNA transcrito tem apenas existncia transitria, sendo limitado apenas a uma extenso de cerca de 12 pares de bases. Aps a passagem da enzima, a cadeia de RNA nascente destacada da sua ligao cadeia molde, dando lugar imediata reconstituio da dupla hlice do DNA, na sua forma nativa. O complexo constitudo pela RNA polimerase, pelo segmento de DNA contendo a pequena regio em que a dupla hlice se encontra desnaturada, e pela cadeia nascente de RNA designado transcription bubble. Em E. Coli, a transcrio um processo muito rpido, que para a sntese dos mRNA, a 37 C, ocorre velocidade calculada de 40 a 50 nucletidos polimerizados por segundo.
2.6.
Terminao da transcrio
2.6.1. Terminao dependente do factor
O factor constitudo por uma protena hexamrica. Quando a terminao dependente de si, ele liga-se sem que seja necessria a formao de quaisquer estruturas secundrias da cadeia nascente, e induz ruptura da ligao RNA/DNA molde complementar. O factor dotado de actividade ATPsica RNAdependente, pelo que se desloca de forma activa sobre a cadeia do RNA nascente, em direco extremidade 3, seguindo de perto o percurso da RNA polimerase. Quando, devido a uma pausa ou atraso da transcrio, a RNA polimerase alcanada pelo factor , o segmento de RNA ainda hibridado com o DNA molde dissocia-se, com libertao da RNA polimerase da sua anterior ligao ao DNA.
40
Captulo 4 Transcrio |
Biologia Molecular 1 Ano - FFUP
2006/2007
Contrariamente aos sinais de iniciao presentes no promotor dos genes bacterianos, os sinais de terminao se encontram no prprio RNA em vias de transcrio e no directamente no DNA molde, quer sejam do tipo dependente ou independente.
2.6.2. Terminao independente do factor
A terminao da sntese das molculas de RNA nos procariotas muitas vezes determinada pela presena de elementos de estrutura palindrnica no DNA. o caso de uma sequncia de nucletidos, rica em G-C, que se encontra numa das cadeias de DNA, e que aparece repetida em posio invertida na regio imediatamente adjacente da cadeia complementar. Foi observado que estes elementos palindrnicos so seguidos de uma regio de DNA constituda maioritariamente por A e T. A sequncia palindrmica vai dar origem a que a cadeia de RNA em formao adquira uma estrutura secundria em gancho, resultante do emparelhamento dos dois segmentos de estrutura complementar e antiparalela, perante a qual a RNA polimerase sofre atraso ou mesmo paragem. O hbrido DNA molde RNA nascente imediatamente a jusante deste elemento estrutural menos estvel por ser formado por emparelhamentos dA-rU, da resultando a ruptura da ligao do RNA nascente ao DNA molde. O imediato re-emparelhamento das duas cadeias complementares do DNA nativo leva libertao da core enzyme, at a ligado ao molde, e da molcula do RNA transcrito.
3. Transcrio nos eucariotas
3.1. RNA polimerases
Nos ncleos das clulas eucariotas, encontram-se trs tipos de RNA polimerases DNA dependentes: RNA polimerase I responsvel pela sntese de cerca de 80% da totalidade do RNA celular, localizase no nuclolo, transcrevendo os genes dos RNA ribossomais, que conduzem produo dos rRNA 18S; 5.8S e 28S. Esta polimerase insensvel inibio pela -amanitina.
| Captulo 4 Transcrio
41
Biologia Molecular - 1 Ano - FFUP
2006/2007
RNA polimerase II responsvel pela sntese de cerca de 2% do RNA celular, localiza-se no nucleoplasma, e catalisa a sntese dos produtos primrios precursores dos RNA mensageiros, que do origem ao hnRNA. A RNA polimerase II inibida pela -amanitina a baixa concentrao. RNA polimerase III responsvel pela sntese de cerca de 20% dos RNA celulares, est igualmente localizada no nucleoplasma, e catalisa a sntese dos RNA de transferncia tRNA e outros pequenos RNA, que incluem o rRNA 5S, o snRNA ou small nuclear RNA e snoRNA ou small nucleolar RNA. A RNA polimerase III animal sensvel inibio pela -amanitina a concentraes relativamente elevadas, mas a RNA polimerase III de levedura e a de insecto so insensveis a este inibidor.
3.2. Regio promotora
Os primeiros genes a serem sequenciados em transcries in vitro foram genes virais e genes codificantes de protenas que so activamente transcritos tanto em alturas particulares do ciclo celular como em tipos especficos de clulas diferenciadas. Em todos estes genes, uma sequncia altamente conservada designada TATA box encontrada aproximadamente 25 a 35 pares de bases acima do local de iniciao. Para alm da TATA box existem outras regies promotoras como a CAAT box, que contm uma citosina na posio -1 e uma timina na posio +1, com duas adeninas pelo meio, e que se encontra cerca de 75 a 80 pares de bases acima do local de iniciao. Por fim, existem outros genes que no contm TATA box nem CAAT box, apresentando uma sequncia rica em C e G com aproximadamente 40 nucletidos, situada 100 pares de bases acima do local de iniciao. O dinucletido CG estatisticamente menos representativo em DNA, sendo considerado como uma regio genmica de DNA que indica que existe uma regio de iniciao da transcrio.
42
Captulo 4 Transcrio |
Biologia Molecular 1 Ano - FFUP
2006/2007
3.3.
Enhancers
A transcrio de vrios genes eucariotas pode ser estimulada por elementos de controlo localizados vrios milhares de pares de bases acima do local de iniciao. Os mais encontrados so designados por enhancers e raramente so encontrados fora de genomas eucariotas. Os enhancers contm um conjunto de elementos proximamente unidos entre si que condicionam a ligao de diversos factores de transcrio que estejam ligados a alguns milhares de pares de bases de distncia. Desta forma, o DNA pode sofrer um rearranjo de tal forma que os factores de transcrio na regio do promotor e do enhancer interajam para formar um complexo proteico de grandes dimenses.
3.4.
Iniciao da transcrio 3.4.1. Complexo de iniciao com promotores contendo TATA box
A primeira enzima que se liga no vai ser a RNA polimerase, porque vai ser puxada para a regio promotora por outros factores de transcrio q uese ligam anteriormente. Em conjunto com uma das RNA polimerases I, II ou III, h uma protena que reconhece e que se liga TATA box, a TBP que tambm necessria para iniciar o complexo de transcrio. Em todos os casos, consoante a RNA polimerase em causa, ela vai estar presente para depois chamar outros factores adicionais, como o SL1 no caso da RNA polimerase I, TFIIB na RNA polimerase II e TFIIIB na RNA polimerase III.
| Captulo 4 Transcrio
43
Biologia Molecular - 1 Ano - FFUP
2006/2007
3.4.2. Complexo de iniciao da RNA polimerase I
Os elementos reguladores que comandam a iniciao com a RNA polimerase I esto localizados relativamente perto do local de iniciao da transcrio. Um core element estende-se sobre o local de iniciao desde -40 a +5, sendo essencial para a transcrio. Um elemento de avano na cadeia estende-se tambm desde -155 a -60, estimulando a transcrio in vitro pela RNA polimerase I. A formao de um complexo de iniciao totalmente activo com a RNA polimerase I comea com a ligao de um factor multimrico de activao (UAF). Duas das seis subunidades que compem o UAF so histonas que provavelmente participam na ligao do DNA. Seguidamente, um elemento trimrico liga-se ao elemento central com a TBP, que cria contacto tanto com o UAF como com o factor central. Finalmente, um complexo pr-formado com RNA polimerase I e Rrn3p associa-se com protenas de ligao, posicionando a RNA polimerase I junto aos locais de iniciao. Em clulas humanas, o TBP estavelmente ligado a outros trs polipptidos, formando o factor de iniciao SL1 que liga o promotor central e funcionalmente equivalente ao TBP.
3.4.3. Complexo de iniciao da RNA polimerase II
Para iniciar a transcrio, a RNA polimerase II necessita de vrios factores gerais de transcrio (denominados TFIIA, TFIIB, e assim por diante). O promotor contm uma sequncia de DNA denominada de TATA box, a qual est localizada a 25 nucletidos do stio no qual a transcrio iniciada. A TATA box reconhecida e ligada pelo factor de transcrio TFIID, o qual permite a ligao adjacente do TFIIB. Os restantes factores gerais de transcrio, assim como a prpria RNA polimerase, associam-se junto ao promotor. Seguidamente, o TFIIH usa ATP para separar a dupla hlice de DNA, no ponto inicial de transcrio, permitindo que a transcrio se inicie. O TFIIH tambm fosforila a RNA polimerase II, modificando a sua conformao, de tal modo que libertada dos factores gerais de transcrio e pode iniciar a fase de elongao da transcrio.
44
Captulo 4 Transcrio |
Biologia Molecular 1 Ano - FFUP
2006/2007
3.4.4. Complexo de iniciao da RNA polimerase III
Ao contrrio dos genes codificantes de protenas e genes de pr-mRNA, as regies promotoras do tRNA e do 5S-RNA contradizem inteiramente a sequncia transcrita. Dois elementos promotores internos, designados por A-box e B-box, esto presentes em todos os genes do tRNA. Estas sequncias altamente conservadas no s funcionam como promotores como tambm codificam duas pores invariantes de tRNA eucariota que so necessrias para a sntese proteica. Nos genes do 5S-RNA, apenas existe uma sequncia promotora interna, a C-box. Existem trs factores de transcrio que so necessrios para a RNA polimerase III iniciar a transcrio do tRNA e do 5S-RNA in vitro. Dois factores multimricos, TFIIIC e TFIIIB participam na iniciao do promotor do tRNA e do 5S-RNA; um terceiro factor, o TFIIIA necessrio para a iniciao dos promotores do 5S-RNA exclusivamente. Tal como acontecia nos complexos de iniciao da RNA polimerase
| Captulo 4 Transcrio
45
Biologia Molecular - 1 Ano - FFUP
2006/2007
I e II, os factores de transcrio da RNA polimerase III ligam-se ao promotor do DNA em sequncias especficas. A metade terminal-N de uma subunidade do TFIIIB, denominado BRF, semelhante sequencialmente ao TFIIB (um factor de transcrio da RNA polimerase II). Esta semelhana sugere que o BRF e o TFIIB apresentem tambm funo semelhante na iniciao, nomeadamente, para direccionar a polimerase para o local de iniciao correcto. Assim que o TFIIIB esteja ligado a um gene de tRNA ou 5SRNA, a RNA polimerase III pode ligar-se e iniciar o processo de transcrio na presena de trifosfatos ribonucleosdicos. A subunidade BRF do TFIIIB interage especificamente com uma das subunidades da polimerase, tendo em conta a iniciao para esta RNA polimerase especfica. Outra das trs subunidades que constituem o TFIIIB o TBP que surge aqui como componente principal dos factores de transcrio para as 3 RNA polimerases eucariticas.
3.5. Terminao da transcrio
Nos eucariotas, os mecanismos para a terminao da transcrio diferem para cada uma das trs RNA polimerases. A transcrio dos genes de pr-RNA pela RNA polimerase I terminada por um mecanismo que requer um factor de terminao especfico da polimerase. Esta protena de ligao do DNA liga-se a uma sequncia especfica no fim da unidade de transcrio. Uma terminao eficiente necessita que o factor de terminao se ligue ao DNA modelo na orientao correcta. A polimerase III pura termina depois de polimerizar uma srie de resduos de U. O hdrido desoxi(A)-ribo(U)-DNA-RNA, que resulta quando uma extenso de Us sintetizada, particularmente instvel comparado com todas as outras sequncias de pares de bases. A facilidade com que este hdrido pode ser fundido provavelmente contribui para o mecanismo de terminao da RNA polimerase III. Na maioria dos genes que codificam protenas de transcritos nos mamferos pela polimerase II, assim que a polimerase transcreve cerca de cinquenta bases, a elongao progressiva altamente susceptvel de ser processada e no termina at que uma sequncia que leve ao corte e segmentao do RNA na sequncia que forma o fim da 3 do mRNA codificado. A RNA polimerase II pode ento terminar num elevado nmero de locais localizados a mais de 0,5 2 kb alm deste local de adio poly(A). 46 Captulo 4 Transcrio |
Biologia Molecular 1 Ano - FFUP
2006/2007
Experincia bioqumicas e de imunoprecipitao de cromatina sugerem que o complexo proteico que corta e poliadenila o mRNA nascente transcreve em sequncias especficas associadas a domnios de terminais carboxlicos fosforilados (CTD) de RNA polimerase II a seguir iniciao. Este complexo de corte/poliadenilao pode suprimir a terminao pela RNA polimerase II at que a sequncia que assinala o corte e poliadenilao seja transcrita pela polimerase. Enquanto que alguma terminao da transcrio no regulada para a maior parte dos genes, para alguns especficos, uma escolha feita entre elongao e terminao ou pausa entre algumas dezenas de bases do local do incio da transcrio. Esta escolha pode ser regulada; contudo, a expresso da protena codificada controlada no s pela iniciao da transcrio, mas tambm pelo controlo antecipado na elongao da unidade de transcrio.
4. Inibidores da transcrio
Rifamicina holoenzima que actua na transcrio dos procariotas que inibe a iniciao. -Amanitina enzima que actua na transcrio dos eucariotas. Nunca inibe a RNA polimerase I, inibe a polimerase II e apenas inibe a RNA polimerase III em algumas espcies. Actinomicina D primariamente utilizada atravs da sua ligao ao DNA no complexo de iniciao da transcrio, inibindo a elongao atravs da RNA polimerase. Streptolidigina enzima central que actua sobre a transcrio dos procariotas, inibindo a sua elongao.
| Captulo 5 RNA e processamento
47
Biologia Molecular - 1 Ano - FFUP
2006/2007
Captulo 5 RNA e processamento
1. RNA mensageiro procariota
1.1. Caractersticas do mRNA
Policistrnico os mRNAs procariotas podem variar relativamente ao nmero de protenas que codificam. Alguns mRNAs representam apenas um nico gene, sendo monocistrnicos, enquanto que outros (a maioria) transportam sequncias codificantes para vrias protenas, ou seja, so policistrnicos. Nestes casos, uma molcula de mRNA transcrita a partir de um grupo de genes adjacentes. Colinear atraves da comparaao da sequencia nucleotidica de um gene com a sequencia de aminoacidos de uma proteina, pode determinar-se directamente se o gene e a proteina so colineares. Assim, isto acontece quando a sequencia de nucleotidos no gene corresponde exactamente sequncia de a.a na proteina. Ou seja, o rna formado a partir do dna igual. Cistro o teste de complementao usado pra determinar se duas mutaes existem no mesmo gene ou em genes diferentes. Consiste na comparao da localizao das duas mutaes, sendo que seelas existem no mesmo gene, ir existir uma diferena nos fentipos das configuraes cis e trans. Assim, a configurao trans mutante porque cada alelo transporta uma mutao, mas a configurao cis apresenta umalelo com duas mutaes e outro alello sem mutao. Desta forma, o mrna um cistro pois a unidade definida pelo teste de complementao. Polaridade o factor rho segue um percurso que estabelece uma relao entre a trancrio e a traduo, que explica um fenmeno de ligao entre os dois processos. Nalguns casos, uma mutao nonsense num gene de uma unidade de transcrio impede a expresso dos genes subsequentes nessa mesma unidade. A principal causadesta caracterstica a falta de mrna correspondente s partes mais distantes da unidade.
1.2. Componentes bsicos do gene procariota
Um gene procariota constituinte do mRNA designado por opero e contm as seguintes estruturas: Promotor actua como iniciador da transcrio para o gene ou genes fisicamente ligados a si no mesmo fragmento de DNA. Pode apresentar um ou mais locais reguladores sendo o mais comum a existncia de duas sequncias consenso. Repressor esta protena impede um determinado gene de ser expresso. Operador localiza-se junto do promotor actuando como alvo da protena repressora. Quando ocorre a ligao entre estes dois elementos, a RNA polimerase nao inicia a transcrio e a expresso gnica interrompida (no caso de operes de controlo negativo).
48
Captulo 5 RNA e processamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
Factor de transcrio factor necessrio para auxiliar a RNA polimerase na iniciao da transcrio junto do promotor. Genes estruturais quaisquer genes que codifiquem para protenas. Representam uma enorme variedade de estruturas e funes proteicas, incluindo protenas estruturais, enzimas com actividade cataltica e protenas reguladoras. Gene regulador descreve um gene estrutural que codifica para a protena envolvida na regulao da expresso de outros genes. Um gene regulador codifica para uma protena que controla a transcrio atravs da ligao a locais particulares do DNA.
1.3. Constituio do mRNA
Regio codificante consiste num conjunto de codes que representa a sequncia de aminocidos da protena iniciando, normalmente, com AUG e terminando com um codo de terminao. 5 leader regio extra presente na extremidade 5 que precede o incio da regio codificante, ou seja, o codo de iniciao (sequncia nao traduzida). 3 trailer sequncia adicional que existe depois do sinal de terminao, constituindo a extremidade 3 (sequncia no traduzida). Regio intercistrnica existe entre as vrias regies codificantes que podem variar em tamanho, podendo apresentar cerca de 30 nucletidos nos mRNAs de bactrias ou apenas 1 a 2 nucletidos separando o codo de terminao de uma protena do codo de iniciao da seguinte.
| Captulo 5 RNA e processamento
49
Biologia Molecular - 1 Ano - FFUP
2006/2007
Sequncia Shine-Dalgarno (5 ...UAAGGAGG...3) sequncia complementar de uma sequncia altamente conservada da extremidade 3 do 16S rRNA. Mutaes pontuais na sequncia de Shine-Dalgarno podem inibir a traduo do mRNA. Por outro lado, a introduo de mutaes na sequncia complementar do rRNA insignificante para a clula alterando o padro de sntese proteica. Uma mutao na sequncia de Shine Dalgarno de um mRNA pode ser eliminada por uma mutao no rRNA que restaura o emparelhamento de bases. Assim sendo, basicamente o papel desta sequncia ser o de colocar o DNA na posio correcta do ribossoma para que se possa iniciar a leitura.
1.3.1. Ligao da sequncia Shine-Dalgarno ao ribossoma
A sequncia existente na extremidade 3 do rRNA conservada entre os procariotas e os eucariotas excepto que nos eucariotas existe a eliminao da sequncia de cinco bases CCUCC que o principal complemento da sequncia de Shine-Dalgarno. Neste local, no ocorre emparelhamento de bases entre o mRNA eucariota e o 18S rRNA, que actua como uma grande diferena no mecanismo de iniciao. Nos procariotas, a subunidade 30S liga-se directamente ao local de ligao do ribossoma. Como consequncia, o complexo de iniciao forma uma sequncia em torno do codo de iniciao AUG. Quando o mRNA policistrnico, cada uma das regies codificantes inicia com um local de ligao ribossmico. A natureza da expresso dos genes bacterianos significa que a transcrio do mRNA bacteriano ocorre sequencialmente atravs dos seus cistres. A partir do momento em que os ribossomas se ligam primeira regio codificante, as regies codificantes seguintes ainda no forem transcritas.
50
Captulo 5 RNA e processamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
1.4. Sequncias de DNA que codificam mais do que uma protena
A maioria dos genes consiste numa sequncia de DNA que apresenta como principal funo a codificao de uma protena. Contudo, existem alguns casos em que a sequncia da cadeia simples de DNA codifica para mais de uma protena. Os overlapping genes existem em casos relativamente simples em que um gene parte do outro. A primeira metade de um gene usada independentemente para poder especificar uma protena que representa a primeira metade da protena especificada por todo o gene. O resultado final o mesmo sempre que ocorra uma clivagem parcial na protena resultante para dar origem a protenas parciais ou completas. Isto acontece na medida em que ao longo de uma cadeia de DNA podem existir vrios codes AUG que indicam o incio da traduo e que vo conduzir a RNA com diferentes tamanhos. Dois genes interagem de forma mais subtil quando a mesma sequncia de DNA partilhada por duas protenas nao homlogas. Esta situao afirma que a mesma sequncia de DNA traduzida em mais de uma sequncia de leitura. Nos genes celulares, a sequncia de DNA usualmente lida em apenas um dos trs possiveis mecanismos, mas em algns genes virais e mitocondriais, ocorre uma interaco entre dois genes adjacentes que so lidos por formas diferentes.
| Captulo 5 RNA e processamento
51
Biologia Molecular - 1 Ano - FFUP
2006/2007
2. RNA mensageiro nos eucariotas
2.1. Componentes bsicos de um gene eucariota
Um gene eucariota pode ser definido como um gene interrompido pois apresenta exes e intres na sequncia codificante. Estes genes, so lidos para formar o pr-RNA ou o hnRNA que vai sofrer modificaes de processamento, como o capping, o splicing e a poliadenilao. O RNA formado no ncleo depois de sofrer o processamento, apresentando na regio promotora vrias sequncias como a TATA box, a CAAT box e eventualmente a GC box junto ao local de iniciao. Existe, depois toda a sequncia codificante, terminada por um sinal de finalizao (codo de terminao) e um segmento que no vai ser traduzido onde existe o sinal de poliadenilao e a extremidade 3 da cpia da molcula de mRNA.
52
Captulo 5 RNA e processamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
2.2. Processamento do hnRNA
A expresso de genes interrompidos requer um passo adicional que no ocorre na expresso de genes no interrompidos. O DNA d origem a uma cpia de RNA (o transcrito) que representa exactamente a sequncia do genoma. No entanto, este RNA apenas um precursor no podendo ser usado para a sntese proteica. Em primeiro lugar, os intres tm de ser removidos da cadeia de RNA para originar um RNA mensageiro que consiste unicamente num conjunto de exes. Este processo designado por RNA splicing, que envolve uma eliminao precisa de um intro do primeiro transcrito, sendo que as extremidades do RNA se juntam para formar uma molcula covalentemente intacta.
2.3. Capping
A transcrio inicia-se com um nuclesido trifosfato. O primeiro nucletido retm o seu grupo 5trifosfato e estabelece a normal ligao fosfodister desde a sua posio 3 at posio 5 do nucletido seguinte. Quando o mRNA maduro tratado in vitro com enzimas que o deviam degradar para nucletidos individuais, a extremidade 5 no expe o nuclesido trifosfato. Desta forma, contm dois nucletidos ligados por uma ligao 5-5 trifosfato e tambm por grupos metilo agora adicionados. A base terminal sempre uma guanina que adicionada ao RNA original depois da transcrio. A adio do terminal G extremidade 5 catalizada por uma enzima nuclear, a guanidil transferase. Esta reaco ocorre to perto do incio da transcrio que se torna impossvel a deteco de algo mais para alm de quantidades do 5-trifosato do RNA nuclear. A reaco total pode ser representada como uma condensao entre um GTP e o terminal original 5-trifosfato do mRNA. Consequentemente, o novo resduo guanlico adicionado extremidade do RNA, apresenta orientao contrria de todos os outros nucletidos, sendo designado por cap que actua como substrato para vrios eventos de metilao.
| Captulo 5 RNA e processamento
53
Biologia Molecular - 1 Ano - FFUP
2006/2007
2.4. Modelos alternativos de splicing
Os modelos alternativos de splicing fazem com que ocorra a incluso de um exo em alguns mRNAs, enquanto vai sendo liberto dos restantes. Um nico tipo de transcrito produzido a partir do gene, mas tambm pode sofrer splicing por dois processos diferentes. No primeiro processo, dois intres so removidos, permitindo a juno de trs exes agora consecutivos. No segundo processo, o segundo exo no reconhecido. Como resultado, um intro de maiores dimenes (visto que inclui dois intres verdadeiros e um exo) removido, fazendo com que o exo em causa seja considerado como sequncia intrnica. Os dois processos originam protenas que so iguais nas extremidades, mas uma delas apresenta uma sequncia adicional num determinado local da cadeia e a outra no. Em certos casos, os dois processos operam simultaneamente, e uma determinada poro de RNA removida em cadsa um deles. Noutros casos, os processos ocorrem alternativamente, sendo expressos em condies diferentes: um num determinado tipo de clulas e outro noutro tipo de clulas.
2.5. Modelo de clivagem e poliadenilao do pr-RNA em mamferos
A formao de uma estrutura terminal na extremidade 3 adequada requer: Endonuclease que consiste nos componentes CFI e CFII, para cortar o RNA. Poli-A polimerase (PAP) que sintetiza a cauda poli-A. Componente de especificade (CPSF) que reconhece a sequncia AAUAAA e comanda as restantes actividades. Factor de estimulao (CstF) condiciona a ligao de que uma sequncia rica em nucletidos de G e U, que se encontra depois do local de clivagem. O factor de especificidade contm quatro subunidades, que quando se encontram juntas se ligam especificamente aoRNA que contm a sequncia AAUAAA. As subunidades individuais so protenas que apresentam vrias sequncias de ligao ao RNA, mas que no se ligam obrigatria e especificamente a essas molculas. As interaces proteicas que existem nas subunidades podem ser necessrias para gerar o local especfico de ligao da sequncia AAUAAA. O CPSF liga-se fortemente a essa sequncia, mas s quando o CstF se encontra presente para se ligar sequncia rica em G-U. O factor de especificidade requerido tanto para a reaco de clivagem como para a reaco de poliadenilao. Geralmente, existe num complexo com uma endonuclease e com uma poli-A polimerase, que comanda a clivagem seguida de poliadenilao.
54
Captulo 5 RNA e processamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
Os dois componentes, o CFI e o CFII (cleavage factors I e II), quando se encontram juntos com o factor de especificidade, so necessrios e suficientes para a clivagem endonucletica. A poli-A polimerase apresenta uma actividade cataltica no especfica. Quando se encontra combinada com outros componentes, a reaco de sntese torna-se especfica para o RNA que contm a sequncia AAUAAA. A reaco de poliadenilao ocorre em duas fases. Em primeiro lugar, uma sequncia oligossacardica relativamente pequena adicionada extremidade 3, numa reaco absolutamente dependente da sequncia AAUAAA, sendo que a poli-A polimerase a completa, no sentido do factor de especificidade. Na segunda fase, a cauda oligo-A aumentada em cerca de 200 resduos de comprimento. Esta reaco requer outro factor de estimulao que reconhece a cauda oligo-A e direcciona a poli-A polimerase especificamente para a extenso da extremidade 3. A poli-A polimerase adiciona resduos adenlicos individualmente extremidade 3. O modo intrnseco de actuao desta enzima distributivo, porque se dissocia aps a adio de cada nucletido. No entanto, na presena do CPSF e da PABP (poly-A binding protein), a enzima actua processivamente para prolongar uma cadeia poli-A individual. O comprimento da cauda poli-A controlado pela PABP, que em certo ponto, limita a aco da poli-A polimerase adio de 200 resduos adenlicos, sendo que este limite pode representar uma acumulao crtica de PABPs na cadeia poli-A. Desta forma, a PABP liga-se ao factor de iniciao da transcrio eIF4G, e consequentemente gera uma terminao da cadeia, onde o complexo proteico contm as duas extremidades 5 e 3 do mRNA.
2.6. Processamento do RNA mitocondrial
O RNA mitocondrial apresenta um processamento caracterstico, devido existncia de uma unidade de informao especfica. Numa fase inicial, ocorre um processo de autocatlise, em que o prprio RNA vai promover o corte de apenas um dos seus dois intres. Consequentemente, os dois exes agora consecutivos vo juntar-se, continuando na mesma com um segundo intro na constituio da molcula. Como existe um sinal de iniciao e outro de terminao logo no processo de remoo dos intres, ele codifica uma protena que depois de sintetizada que vai remover o segundo intro, sendo essa protena denominada de maturase. Seguidamente, existe na mesma um corte inicial do primeiro intro, mas em vez de ser por aco de uma enzima, o proprio RNA que catalisa esse corte, e que faz com que o segundo corte j passe pela aco de uma protena codificada por esse prprio RNA.
| Captulo 5 RNA e processamento
55
Biologia Molecular - 1 Ano - FFUP
2006/2007
2.7. Processamento do mRNA
O processamento do mRNA nos eucariotas requer a formao de uma estrutura especfica denominada spliceossoma e requer tambm o comprimento de determinadas regras que existem e que se caracterizam na sequencia do intro. Existe um intro que tem de ser removido e para que essa remoao tenha sucesso, na regio intrnica da extremidade 5 tem de existir GU e na extremidae 3 tem de existir AG. Alm disso, existe uma regio a 500 pares de bases da extremidade 3 que rica em pirimidinas e tambm um ponto de ramificao. Se houver modificaes que interferem nas duas bases que delimitam o intro, no se consegue remov-lo. Tendo por base as regras de splicing que tm de existir, o processamento no ocorre sem a presena de outros RNA, como os snRNAs sintetizados pela RNA polimerase II.
Os locais de splicing das extremidades 5 e 3 e a sequncia entre elas so reconhecidos pelos componentes do aparelho de splicing que se forma para originar um complexo de grandes dimenses. Este complexo junta os locais de splicing das duas extremidades antes da ocorrncia de qualquer reaco. O complexo rene-se sequencialmente com o pr-mRNA, e vrios intermedirios podem ser reconhecidos 56 Captulo 5 RNA e processamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
por complexos fraccionados de diferentes dimenses. Desta forma, o fenmeno de splicing ocorre aps todos os componentes estarem reunidos e agregados. O aparelho de splicing contm protenas e RNAs, para alm do pr-mRNA. Os RNAs adquirem a forma de pequenas molculas que existem como protenas ribonucleoproteicas. Tanto o ncleo como o citoplasma das clulas eucariticas contm vrias espcies discretas de pequenos RNAs. Eles variam no seu tamanho, podendo apresentar entre 100 e 300 bases nos eucariotas superiores e cerca de 1000 bases nos eucariotas inferiores. Os RNAs restritos ao ncleo so designados small nuclear RNAs (snRNA) e os encontrados no citoplasma so denominados de small cytoplasmic RNAs (scRNA). No seu estado natural, existem ambos como partculas ribonucleoproteicas (snRNP e scRNP). Coloquialmente, podem ser designados por snurps e scyrps. Existe ainda uma pequena classe de RNAs encontrada no nuclolo small nucleolar RNAs (snoRNA), que est envolvida no processamento do rRNA. Os snurps ou scyrps (snRNAs ou scRNAs) so nucleares ou citoplasmticos, conforme o caso, e no spliceossoma so designados por U1, U2, U3, U4, U5 e U6. As snRNPs envolvidas no splicing, juntas com vrias protenas adicionais, foram um complexo de grandes dimenses designado spliceossoma. Isolado dos sistemas de splicing in vitro, compreende uma partcula ribossmica aproximadamente 50-60S e geralmente pode ser formado em vrias fases, medida que as snRNPs se vo juntando, formando complexos de pr-splicing.
2.7.1. Splicing e o spliceossoma
O processo de splicing pode ser amplamente dividido em duas fases: Em primeiro lugar, as sequncias consenso no local de splicing da extremidade 5, a sequncia central e a pirimidina adjacente so reconhecidos, ocorrendo a formaao de um complexo que contm todos os componentes necessrios ao splicing. De seguida, a clivagem e as reaces de ligao alteram a estrutura do RNA substrato. Desta forma, os componentes do complexo so libertados e/ou reorganizados medida que se vo processando as reaces de todo o processo de splicing. O reconhecimento das sequncias consenso envolve tanto os RNAs (snRNAs) como as protenas (snRNPs). Certas molculas de snRNAs apresentam sequncias que so complementares s sequncias consenso, sendo que o emparelhamento de bases entre o snRNA e o pr-mRNA ou entre dois snRNAs desempenha um papel de grande importncia no processo final de splicing. A snRNP U1 contm 8 protenas, tal como o snRNA correspondente. A ligao desta protena ao local de splicing da extremidade 5 o primeiro passo no processo de spliicng, sendo que a participao desta protena envolve uma interaco entre uma das suas subunidades e a protena ASF/SF2 que surge como factor geral de splicing, que pertence classe SR.
| Captulo 5 RNA e processamento
57
Biologia Molecular - 1 Ano - FFUP
2006/2007
O primeiro complexo formado durante o splicing designa-se complexo E (early presplicing complex) e contm a snRNP U1, o factor de splicing U2AF e alguns membros de uma famlia de protenas SR, que compreendem um grupo importante de factores e regulados do splicing na sua totalidade. No complexo E, o U2AF est ligado regio entre a sequncia central e o local de splicing da extremidade 3. Mais tarde, o complexo E transformado no complexo A quando a snRNP U2 se liga sequncia central, requerendo tanto a snRNP U1 como o U2AF para o processamento da ligao. Isto acontece porque o snRNA U2 inclui sequncias complementares sequncia central. Para alm disso, uma sequncia perto da extremidade 5 do snRNA emparelha com a sequncia central intrnica. Seguidamente, vrias protenas da snRNP U2 esto ligadas ao RNA substrato imediatamente acima da sequncia central. Assim, a adio da snRNP U2 ao complexo E conduz formao do complexo A (advanced presplicing complex), tendo em conta que esta ligao requer a hidrlise de molculas de ATP e condiciona uma molcula de pr-mRNA para o percurso de splicing. Depois da formao dos complexos E e A, outras snRNPs e outros factores de splicing associamse ao ltimo complexo numa ordem j pr-estabelecida. Forma-se o complexo B1 quando um trmero contendo as snRNPs U5 e U4/U6 se ligam ao complexo A que j continha as snRNPs U1 e U2. Este complexo identificado como um spliceossoma, a partir do momento que contm os componentes necessrios para ocorrer todo o restante processo de splicing. Quando existem os elementos necessrios para conduzir justaposio da snRNP U6 ao local de splicing da extremidade 5, inicia-se a formao do complexo B2, sendo que para tal, tambm preciso que ocorra a libertao da snRNP U1 e que ocorram vrias alteraes na U5. Esta sofre modificaes na sua posio, pois inicialmente encontrava-se junto s sequncias exnicas do local de splicing da extremidade 5 e agora, devido U6, passa a encontrar-se nas proximidades das sequncias intrnicas. A reaco cataltica iniciada com a libertao da snRNP U4, requerindo a hidrlise de uma molcula de ATP. Simultaneamente, a U5 mantm-se ligada ao local de splicing da extremidade 3 e a U2/U6 catalisa a reaco de transesterificao, conduzindo formao do complexo C1. Por fim, as snRNPs U5 e U2/U6 mantm-se ligadas no complexo C2, mas com a clivagem da extremidade 3 e com a ligao de todas as sequncias exnicas entre si, ocorre a sua libertao e a consequente formao da molcula de RNA que apenas contm exes.
58
Captulo 5 RNA e processamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
2.8. RNA editing
O RNA editing um processo em que a informao muda de nvel no mRNA. encontrado em situaes em que a sequncia codificante no RNA difere da sequncia de DNA de onde foi transcrita. O RNA editing ocorre em duas situaes distintas, com causas distintas. Em clulas eucariticas, existem casos em que a substituio ocorre numa base individual do mRNA, provocando uma mudana da sequncia da protena que codificada. O genoma contm um gene individual e interrompido, cuja sequncia idntica em todos os tecidos, e que apresenta uma sequncia codificante de 4563 codes. Este gene transcrito numa molcula de mRNA que traduzida numa protena de 512 kD, representando a sequncia codificante completa, que sintetizada no fgado. Uma forma mais curta da protena, produzida no intestino, com aproximadamente 250 kD, que consiste na metade do terminal-N da protena completa. transcrita para mRNA, cuja sequncia idntica interior excepto num codo. Esta substituio ocorre pois nenhum gene ou exo alternativo est disponvel no genoma para codificar para a nova sequncia, e alm disso no h alterao no padro de splicing, permitindo concluir que a modificao foi devida a uma alterao directa na sequncia do transcrito. Este tipo de adio raro, mas a apolipoprotena-B (apo-lipo-B) no nica. Os eventos de RNA editing na apo-B causam a modificao da C2153 para um uracilo e nos receptores de glutamato, a adenina passa a inosina. Estes eventos so designados de desaminaes, pois o grupo amino do anel nucleotdico removido. Estas alteraes so catalisados por enzimas designadas por citidina e adenosina desaminases, respectivamente. A especificidade destas reaces de RNA editing devida, maioritariamente, s enzimas intervenientes. Desta forma, as enzimas que desaminam os nucletidos apresentam grande particularidade de actuao, como o caso da adenosina desaminase que actua em qualquer resduo de adenina, numa regio de emparelhamento de RNA. As enzimas de RNA editing esto relacionadas com as desaminases gerais, mas tambm apresentam outras subunidades que controlam a sua especificidade no interior desta classe. No caso do editing feito pela apo-B, a subunidade cataltca de um complexo de editing est relacionado com a citidina desaminase das bactrias, mas tambm apresenta uma regio adicional para a ligao do RNA que ajuda a reconhecer o local especfico para o editing. Neste caso, uma adenosina desaminase de caractersticas especiais que reconhece esses locais no receptor de glutamato do RNA.
| Captulo 5 RNA e processamento
59
Biologia Molecular - 1 Ano - FFUP
2006/2007
Por fim, no caso da GluR-B RNA, uma regio j com bases emparelhadas, que necessria para o reconhecimento do local alvo de ligao, formada entre a regio editada no exo e a sequncia complementar no intro correspondente. Desta forma, um padro de falta de emparelhamento criado, pois necessrio na regio entre os dois intres complementares, sendo que diferentes sistemas de RNA editing podem apresentar diferentes tipos de sequncias especficas necessrias para o seu substrato.
3. RNA de transferncia
Numa molcula de mRNA, os codes no reconhecem directamente os aminocidos que determinam, por exemplo, o grupo de trs nucletidos no se liga directamente ao aminocido. Mais exactamente, a traduo do mRNA em protena depende de molculas adaptadoras que podem reconhecer e se liguem ao codo, e noutra regio da sua superfcie, ao aminocido. Estes adaptadores consistem de um conjunto de pequenas molculas de RNA conhecido como RNA de transferncia (tRNA), cada um com tamanho de aproximadamente 80 nucletidos. As molculas de RNA podem dobrar-se em estruturas tridimensionais estabelecidas com alta preciso, e as molculas de tRNA fornecem um extraordinrio exemplo disso. Quatro pequenos segmentos do tRNA dobrado formam a dupla hlice, produzindo uma molcula que se assemelha a uma folha de trevo quando desenhada esquematicamente. Por exemplo, uma sequncia 5-GCUC-3 numa regio da cadeia polinucleotdica pode formar uma associao relativmente forte com uma sequncia 5-GAGC-3 noutra regio da mesma molcula. A folha de trevo submetida ainda a outras dobras para formar uma estrutura compacta em forma de L que mantida unida por meio de pontes de hidrognio adicionais entre diferentes regies da molcula.
60
Captulo 5 RNA e processamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
Todas as molculas de tRNA apresentam estruturas secundrias e tercirias. A estrutura secundria pode ser expressa na forma de folha de trevo, em que as bases complementares emparelham, formando loops, designados braos do tRNA. As suas sequncias incluem bases pouco usuais que geralmente so obtidas por modificao de quatro bases normais depois da sntese da cadeia polinucleotdica. Os principais quatro braos que existem na estrutura do tRNA so: Brao aceitador consiste num conjunto de bases emparelhadas que termina numa sequncia no emparelhada, em que o 2-OH ou o 3-OH livre pode ser ligado ao aminocido. Braco TC designado desta forma devido presena deste tripleto em que o representa a pseudouridina base modificada. Braco anticodo contm sempre o tripleto de anticodo no centro do loop. Brao D designado desta forma porque contm a base dihidrouridina base modificada. Brao extra contm entre 3 e 21 bases e est entre os braos TC e anticodo.
3.1. Processamento do pr-tRNA
Ao longo do processamento deste tipo de RNA existem trs principais etapas correspondentes a trs molculas diferentes: um transcrito primrio, um intermedirio e um maduro. O transcrito primrio, por sua vez, sofre trs modificaes sucessivas de bases, sendo duas clivagens e uma adio. Inicialmente, a RNase P remove a extremidade 5 leader da cadeia, depois a RNase D remove a extremidade 3 trailer, e por fim adicionada uma sequncia CCA na extremidade 3. A molcula obtida vai ser designada por transcrito intermedirio. Esta molcula vai ser alvo do processo de splicing, sendo que o objectivo a remoo de sequncias intrnicas que existem num dos loops. Por fim, a molcula final obtida vai ser o transcrito maduro de pr-tRNA que vai seguir o processo de processamento.
| Captulo 5 RNA e processamento
61
Biologia Molecular - 1 Ano - FFUP
2006/2007
3.2. Processamento do tRNA
Os produtos da clivagem do tRNA so um intro de estrutura linear e duas molculas que consistem em metade da molcula de tRNA, todas elas com extremidades caractersticas. Cada extremidade 5 termina num grupo OH e cada extremidade 3 termina num 2,3-fosfato. As duas metades de tRNA emparelham para adquirirem uma estrutura semelhante do tRNA e quando a molcula de ATP adicionada, ocorre uma segunda reaco, em que as duas extremidades caractersticas criadas pela endonuclease tm de ser alteradas. O grupo fosfato de estrutura cclica aberto para originar um terminal 2-fosfato, sendo que esta reaco requer a aco de uma fosfodiesterase cclica, e vai originar um grupo 2-fosfato e um grupo 3-OH. O grupo 5-OH criado pela nuclase tem de ser fosforilado para originar um 5-fosfato, conduzindo formao de uma maior proximidade entre o 3-OH e o 5-fosfato. Desta forma, pode concluir-se que os principais intervenientes neste processamento o ATP e o GTP para a juno dos exes decorrentes do processamento, a endonuclease que permite a remoo dos intres e a fosfatase ou fosfodiesterase que remove o grupo fosfato em 2.
4. RNA ribossmico
4.1. Ribossomas procariotas e eucariotas
Cada uma das subunidades ribossmicas contm uma molcula principal de rRNA e um variado nmero de pequenas protenas. No caso dos seres procariticas, a subunidade pequena (30S) consiste no 16S rRNA e em 21 protenas-r. A subunidade grande (50S) consiste no 23S rRNA, no 5S RNA e em 31 protenas-r. Com a excepo de uma protena presente em quatro cpias por ribossoma, todas as outras s se encontram numa nica cpia. As molculas maiores de rRNA constituem a grande parte da massa do ribossoma procariota. A sua presena extremamente importante, e propriamente a maior parte das protenas ribossmicas contactam, actualmente, com o rRNA. Desta forma, estas partculas foram o que geralmente 62 Captulo 5 RNA e processamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
designado por esqueleto de cada subunidade, que constitui uma linha contnua que determina a posio das protenas ribossmicas. No caso dos ribossomas eucariticas, estes apresentam maiores dimenses que os procariotas, pois o contedo total de rRNA e protenas superior, as molculas principais de rRNA so maiores e existem mais protenas-r. Desta forma, a subunidade maior (60S) contm o 28S, o 5.8S e o 5S rRNAs mais 49 protenas e a subunidade menor (40S) contm o 18S rRNA e 33 protenas.
4.2. Genes do rRNA procariota
No rRNA procariota, as sequncias esto intervaladas por tRNA, tendo de sofrer processamento e originando as vrias partculas necessrias para constituir a partcula ribossmica. Esse processamento passa normalmente por cortes feitos por genes do mRNA eucariota, que tem a capacidade de cortar a cadeia de RNA. No caso dos ribossomas procariotas, o rRNA precursor contm 5S rRNA e uma ou duas sequncias de tRNA. Existem 7 operes rrn que esto dispersos ao longo do genoma, quatro loci rrn que contm dois genes de tRNA entre o 16S e o 23S rRNA e outro loci rrn que contm dois genes de tRNA nesta regio. Outros genes adicionais de tRNA podem ou no estar presentes entre a sequncia 5S e a extremidade 3. Desta forma, o precursor com os subunidades 16S, 23S e 5S vai sofrer metilao e induzir a actuao de vrias RNases no passo seguinte clivagem. Aqui, forma-se um intermedirio 17S, por aco da RNase III, um tRNA por aco da RNase P, um 25S pela RNase III e um 5S pela RNase E. Estes intermedirios sofrem a interveno de nucleases que removem as sequncias intrnicas e originam os rRNAs maduros.
| Captulo 5 RNA e processamento
63
Biologia Molecular - 1 Ano - FFUP
2006/2007
4.3. Genes do rRNA eucariota
No rRNA eucariota, existem espaadores externos (ETSs) que ligam as vrias unidades que codificam para o rRNA e dentro dele, existem espaadores internos (ITSs) que vo separar as diferentes unidades e que tm de ser removidos, atravs de um processamento de corte. Neste rRNA, excepo da subunidade 5S, as outras so todas sintetizadas simultaneamente aps o processamento.
4.4. Estrutura do ribossoma em E. coli
O ribossoma em E. Coli usualmente denominado por ribossoma 70S, desde que esta unidade utilizara como medida de quo rpido os sedimentos deste ribossoma sedimentam quando centrifugados. Este ribossoma, contm duas subunidades, sendo a maior a 50S e a menor a 30S que por sua vez se podem subdividir em pequenas molculas de rRNA e em protenas ribossmicas. A maior contm o 23S e o 5S rRNA juntamente com 34 protenas-r e a menor contm o 16S rRNA juntamente com 21 protenas-r. Estes ribossomas podem ser reconstitudos atravs de vrios processos. Isso possvel atravs de modificaes, no s a nivel de temperatura mas tambm atravs de modificaes adicionando compostos que permitem a associaao de partculas das subunidades ribossmicas e a associao das partculas para 64 Captulo 5 RNA e processamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
obter uma partcula ribossmica completa. Embora o ribossoma seja constitudo por duas subunidades, elas no so sempre constante, logo no fim de uma reaco em que se verifica sntese proteica, pode ou no haver reaco entre as diferentes subunidades.
4.5. Processamento do rRNA
O processamento do rRNA um processamento que pode ocorrer por duas principais vias: splicing tipo I e splicing tipo II. De um modo geral, ocorre num processo autocataltico, em que o rRNA funciona como ribozima, tendo capacidade de reconhecer os seus intres e cort-los. Desta forma, consoante o tipo de splicing, o processamento vai conduzir sada do intro numa estrutura linear no caso do splicing tipo I ou numa estrutura cclica no splicing tipo II. Para alm disso, no processamento do rRNA, existe uma reaco de transesterificao em que a base G vai promover o corte do intro na extremidade 5 devido sua actividade de ribozima, vai promover o corte na outra extremidade e permitir a ligao entre os dois. No caso particular do splicing tipo II, em vez de uma guanina, uma adenina que vai catalisar a reaco de transesterificao, fazendo com que o intro depois de ser cortado, saia na forma cclica. Neste processamento, importante referir que por vezes podem intervir alguns mRNAs, sendo necessria a interveno da partcula U3.
| Captulo 5 RNA e processamento
65
Biologia Molecular - 1 Ano - FFUP
2006/2007
5. Pseudogenes
Os pseudogenes () so definidos devido ao seu contedo em sequncias que esto relacionadas com os genes funcionais, mas que no podem ser traduzidos numa protena funcional. Alguns pseudognes apresentam a mesma estrutura enquanto genes funcionais, com sequncias correspondentes a exes e a intres nos locais usuais. Por vezes, podem tornar-se inactivos atravs de mutaes que inibem uma ou vrias fases da expresso gnica: Abolio de sinais de iniciao; Remoo de intres e das junes exo-intro; Presena de sinais de terminao prematuros; Por transcrio reversa de RNA em DNA de cadeia dupla que depois se insere em qualquer parte do genoma. Os pseudogenes processados so aquelas sequncias genmicas inactivas que reconhecem o transcrito de RNA e geralmente so originados atravs da sua insero num local aleatrio de um produto derivado do RNA, seguida de uma reaco de retrotransposio. A eliminao de um par de bases no codo 20 do pseudogene 2 causou uma mutao frameshift que poderia conduzir terminao logo de seguida. Vrios pontos mutantes produziram alteraes nos codes que representavam aminocidos que so considerados altamente conservados nas globinas . Nenhum dos dois intres vai possuir extremidades reconhecveis com os exes, fazendo com que provavelmente os intres possam no sofrer splicing quando o gene transcrito. No entanto, verifica-se que no existem quaisquer transcritos correspondentes ao gene, possivelmente devido a alteraes na regio de corte da extremidade 5. Se o pseudogene se tornou inactivo assim que foi produzido pela duplicao da 1, pode esperar-se que o local de substituio e de manuteno apresentem caractersticas semelhantes. No entanto, actualmente existe um menor nmero de locais de substituio do que de manuteno. Isto sugere que no incio (quando o gene era expresso) existiam seleces em comparao s substituies, permitindo distinguir o tempo de inactivao do gene activo, devido a mutaes. Desta forma, o gene da 1 globina foi activo h cerca de 55 milhes de anos, mas o pseudogene da mesma globina, foi inactivo h 35 milhes de anos.
66
Captulo 5 RNA e processamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
Captulo 6 Traduo
Basicamente, a traduo o processo em que se realiza a produo de uma protena a partir do mRNA. associando-se s protenas, O rRNA inicialmente armazenado nos nuclolos, passa para o citoplasma e associando forma os ribossomas (organelos nos s quais ocorre a sntese proteica). O mRNA move-se se para o citoplasma e liga-se aos ribossomas. Ele formado mado por uma sequncia de trades de nucleotdeos (trs nucleotd nucleotdeos), e cada trade corresponde a um aminocido. As trades so denominadas codes e a combinao dos diferentes codes determina o tipo, o nmero e a posio dos aminocidos na cadeia polipe polipeptdica. O tRNA desloca-se se para o citoplasma, onde se liga ao aminocido do anticodo correspondente, levando-o levando at o ribossoma. Os tRNA transportam aminocidos especficos, salvo raras excepes. exce O desenho abaixo esquematiza como ocorre a sntese proteica nos ribossomas.
| Captulo 6 Traduo
67
Biologia Molecular - 1 Ano - FFUP
2006/2007
A traduo envolve 3 fases. So elas:
A iniciao envolve as reaes que precedem a formao da ligao do peptdeo entre os primeiros dois a.a. da protena. Requer a ligao do ribossoma ao mRNA, dando forma a um complexo da iniciao que contenha o primeiro aminoacil-tRNA. Esta uma etapa relativamente lenta na sntese da protena, e determina geralmente a taxa em que um mRNA traduzido.
A elongao inclui todas as reaes da sntese da primeira ligao do peptdeo adio do a.a.. Os a.a. so adicionados cadeia, umde cada vez; a adio de um a.a. a etapa mais rpida na sntese da protena.
A terminao abrange as etapas que so precisas para libertar a corrente terminada do polipeptdeo; ao mesmo tempo, o ribossoma dissocia-se do mRNA.
Erros na transcrio
Os erros em transcrever o mRNA so raros, tendo, provavelmente, uma quantidade inferior a 10-6. Este um estgio importante a controlar, porque uma nica molcula de mRNA traduzida em muitas cpias da protena, no existindo ainda grandes conhecimentos acerca dos mecanismos que intervm nestes processos.
Geralmente, o ribossoma pode fazer dois tipos de erros na sntese da protena. Pode causar um frameshift saltando uma base quando l o mRNA (ou no sentido inverso lendo uma base duas vezes, uma vez que a ltima base de um codo e outra vez com a primeira base do codo seguinte). Estes erros so raros. No segundo erro, pode permitir que um aminoacil-tRNA incorrecto seja emparelhado com um codo, de modo que o a.a. errado seja incorporado. Este provavelmente o erro mais comum na sntese da protena e controlado pela estrutura e pela velocidade do ribossoma.
Uma tRNA sintetase pode fazer dois tipos de erro. Pode colocar o aminocido errado no seu tRNA; ou pode carregar o a.a. com o tRNA errado. A incorporao do a.a. errado mais comum, provavelmente porque o tRNA oferece uma superfcie maior como a superfcie em que a enzima pode fazer muitos mais contactos para assegurar a especificidade.
68
Captulo 6 Traduo |
Biologia Molecular 1 Ano - FFUP
2006/2007
1. Iniciao
1.1. Incio da cadeia polipeptdica
A sntese de qualquer protena comea com o mesmo aminocido, a metionina. O sinal para iniciar a sntese de um polipptido um codo especial de iniciao que marca o incio da sequncia de leitura. Geralmente, o codo de iniciao o tripleto AUG, mas nas bactrias os codes GUG ou UUG tambm podem ser utilizados. O codo AUG representa a metionina, e dois tipos de tRNA podem transportar este aminocido consigo. Um deles usado para a iniciao enquanto que o outro tem a capacidade de reconhecer os codes AUG durante a elongao. Nas bactrias e nas organelas eucariticas, o tRNA iniciador transporta um resduo de metionina que tinha sido formilado no seu grupo amino, formando uma molcula de N-formil-metionil-tRNA, tambm conhecido por tRNAfMet. O nome do aminoacil-tRNA usualmente abreviado para fMet-tRNAf. O tRNA iniciador adquire o seu aminocido modificado num processo de duas fases. Em primeiro lugar, ele colocado em reaco com o aminocido para gerar o Met-tRNAf e de seguida, ocorre a reaco de formilao representada na figura ao lado, que bloqueia o grupo NH2 livre at ao momento. No entanto, ainda que este grupo esteja bloqueado e pudesse prevenir um determinado iniciador de participar na elongao da cadeia, ele no interfere com a capacidade de iniciar uma outra protena. Este RNA , ento, usado apenas para a iniciao. Isto acontece porque ele reconhece os codes AUG ou GUG (ou, por vezes, UUG). Os codes no so reconhecidos de forma igual, porque a extenso da fase de iniciao rejeita cerca de metade dos codes quando o codo AUG substitudo pelo codo GUG e rejeita cerca de outra metade quando o UUG utilizado. As espcies responsveis pelo reconhecimento dos codes AUG em localizaes internas so os tRNAmMet. Este tRNA responde unicamente aos codes internos AUG, logo a sua metionina no pode ser formilada. Existe um conjunto de diferenas entre o fMet-tRNAf iniciador e o Met-tRNAm elongador, sendo que algumas delas so necessrias para impedir que o iniciador seja utilizado como elongador e outras so necessrias para intervir no processo de iniciao: A formilao no estritamente necessria pois o MettRNAf no formilado pode funcionar como iniciador, mas
| Captulo 6 Traduo
69
Biologia Molecular - 1 Ano - FFUP
2006/2007
aumenta a eficincia com a qual o Met-tRNAf utilizado, na medida em que uma das funes reconhecidas pelo factor IF-2 que se liga ao tRNA iniciador. As bases que se encontram frente a frente na ltima posio da cadeia, s quais o aminocido se liga, esto emparelhadas em todos os tRNAs excepto no tRNAfMet. Algumas mutaes podem criar um emparelhamento de bases nesta posio do tRNAfMet e permitem que ele actue como elongador. A falta deste par , no entanto, importante para impedir que o tRNAfMet seja utilizado na elongao e para o desenrolar da reaco de formilao. Um conjunto de trs pares G-C, no final da cadeia, que precede o anel contendo o anticodo nico para o tRNAfMet. Estes pares de bases so necessrios para permitir que o fMet-tRNAf seja inserido directamente no local P do ribossoma.
1.2. Utilizao do fMet-tRNAf pelo IF-2
O significado dos codes AUG e GUG depende do seu contexto na molcula. Quando o codo AUG utilizado na iniciao, ele lido como uma formilmetionina, mas quando usado na regio de codificao j representa a metionina. Por sua vez, o significado do codo GUG ainda mais dependente da sua localizao. Quando se encontra no primeiro codo, lido atravs da reaco de iniciao como uma formil-metionina, mas quando se encontra presente no interior de um gene ligado como um Val-tRNA, um dos membros regulares do conjunto de tRNA, apresentando a valina como necessria para o cdigo gentico. Num complexo de iniciao, uma pequena subunidade individual est ligada ao mRNA. O codo de iniciao permanece no interior da parte do local P transportando por essa subunidade. O nico aminoaciltRNA que se pode tornar elemento do complexo de iniciao o iniciador que apresenta a propriedade particular de ser capaz de entrar directamente para o local P para reconhecer o seu codo. Quando a subunidade maior entra para o complexo, os locais parciais de ligao do tRNA so convertidos para os locais P e A intactos. O fMet-tRNAf iniciador ocupa ento o local P e o local A permanece disponvel para a entrar de um aminoacil-tRNA complementar para o segundo codo do gene. Assim, a primeira ligao peptdica forma-se entre o iniciador e o aminoacil-tRNA seguinte. A iniciao comea totalmente quando um codo AUG (ou GUG) se encontra num local de ligao ribossmico, pois apenas o tRNA iniciador pode entrar para o local P criado quando a subunidade 30S se liga novamente ao mRNA. A leitura interna inicia-se subsequentemente quando os codes so enfrentados por um ribossoma que se encontra a traduzir continuamente o mRNA, por apenas os aminoacil-tRNAs regulares poderem estar no local A (completo). 70 Captulo 6 Traduo |
Biologia Molecular 1 Ano - FFUP
2006/2007
1.3. Factores de iniciao nos procariotas
Nos procariotas verifica-se que o primeiro aminocido a ser incorporado a forma formilada da metionina (f-metionina) que transportada por um tRNA diferente do tRNA que transportada por um tRNA diferente do tRNA que transporta, posteriormente, os resduos de metionina. A seleco do cdigo AUG que inicia a traduo feita pela presena de caractersticas adicionais na regio 5 do mRNA. Na E. Coli, Shine e Dalgarno mostraram, em 1974, que os mRNA apresentavam uma regio rica em bases pricas constituda por 3 a 10 nucletidos com uma sequncia presente no rRNA 16S. Esta sequncia especfica ficava localizada a montante do codo de iniciao. O emparelhamento de bases entre estas sequncias do mRNA e do rRNA 16S permite ao ribossoma encontrar o codo AUG responsvel pelo incio da traduo. Desta forma, nos procariotas ocorre a iniciao da traduo na presena de vrios factores de iniciao (IFs). Inicialmente, ocorre a ligao do IF1 e do IF3 subunidade maior livre 70S do ribossoma que se vai dissociar nas duas subunidades 30S e 50S. De seguida, a regio 5 do mRNA liga-se subunidade ribossomal 30S livre, permitindo a libertao do IF3. Com isto, o iniciador fMet-tRNAfMet transportado pelo IF2-GTP liga-se ao local P da mesma subunidade ribossmica 30S. Por fim, a ligao da subunidade ribossmica 50S ocorre e os factores IF1 e IF2-GDP libertam-se, juntamente com um Pi.
1.4. Factores de iniciao nos eucariotas
A traduo inicia-se quando h um aminoacil tRNA que se liga ao local P da subunidade pequena do ribossoma, em vez de se ligar ao local A. Assim, com esta ligao, este aminoacil tRNA tem um codo complementar de iniciao AUG. A ligao do aminoacil tRNA ao local P, bem como a ligao da subunidade pequena ao mRNA, deve-se participao de factores proteicos ou factores de iniciao (eIF, eIF1, eIF2 e eIF3). O eIF2 vai, ento, ligar-se molcula de tRNA, fazendo com que esta molcula se possa ligar ao local P do ribossoma. No entanto, esta molcula pode sofrer alteraes e deixa de se poder ligar ao tRNA, sendo necessrios novos mecanismos de regulao.
| Captulo 6 Traduo
71
Biologia Molecular - 1 Ano - FFUP
2006/2007
O eIF2 na forma inactiva tem ligado a si GDP e na forma activa, troca para GTP. Para isso, necessrio que o eIF2 sofra fosforilao atravs de cinases. As duas subunidades do ribossoma esto separadas no citosol. A pequena est ligada ao eIF3 e a grande est ligada ao eIF6, sendo que quando se encontram neste forma, perdem a capacidade de se unir e impedem o decorrer da traduo. Por sua vez, o eIF2 que permanece ligado ao GTP, pode associar-se ao aminoacil tRNA e para se conseguir ligar ao local P do ribossoma, tem de estar aqui associada a subunidade menor, ligando-se ao eIF1A, e formando um complexo de pr-iniciao (tem esta designao porque ainda no comeou a sntese). Depois de a molcula ter o eIF4, o complexo de iniciao liga-se, iniciando a sntese da molcula de RNA. No entanto, se o tRNA est ligado a outras moleculas, caso haja um novo aminoacil tRNA no vai ter capacidade de se ligar. Quando se forma o complexo de iniciao, liga-se molcula de RNA na extremidade 5 e segue at encontrar o codo AUG. Quando o encontra, d-se o estabelecimento de pontes de hidrognio entre o codo e o anticodo presente a nvel do RNA. Quando h pontes de hidrognio, a subunidade maior pode ento ligar-se, mas inicialmente o GTP hidrolisa-se e o eIF2 deixa de ter capacidade de ligao e todos os factores ligados at ao momento, desligam-se tambm. A partir deste momento, a subunidade maior do ribossoma j tem a capacidade de se associar menor. No entanto, como tem ligado a si o eIF6, impede esta conexo, necessitando de um novo factor activo com uma molcula de GTP que permita a ligao do eIF6 subunidade menor, o IF5. Assim que a ligao ocorre, os factores eIF5 e eIF libertam-se, juntamente com a GTP, permitindo que finalmente se inicie a sntese do peptdeo.
2. Elongao
A elongao corresponde etapa da formao da cadeia peptdica, ou seja, a sntese da ligao peptdica entre aminocidos ordenados segundo a informao transmitida pelo mRNA. Este processo ocorre nos ribossomas em 3 fases de um ciclo que se repete continuamente. Este ciclo, que permite a ligao de cerca de 40 resduos por segundo, envolve a participao de vrios factores proteicos designados por factores de elongao (EFs). Com o terminar da etapa de iniciao, as subunidades ribossomais voltam a estar ligadas, o que permite ocorrer o emparelhamento de um outro tRNA transportando o aminocido correspondente ao segundo 72 Captulo 6 Traduo |
Biologia Molecular 1 Ano - FFUP
2006/2007
codo de leitura. Subsequentemente, pode-se formar a primeira ligao peptdica, ou seja, comea a elongao. Um aminoacil-tRNA endereado para o centro A num complexo que envolve uma molcula de GTP associada ao factor EF-1 (EF-Tu nas bactrias) e, subsequentemente, d-se o emparelhamento entre o anticodo do tRNA e o respectivo codo do mRNA no centro A. Este emparelhamento de bases est associado com a activao do domnio GTPase do factor de elongao e, com hidrlise do GTP, dissociado o aminoacil-tRNA do complexo que ocupa o centro A. O GDP dissociado do factor EF1A/EF-Tu substitudo posteriormente por GTP; A ligao peptdica sintetizada, na segunda fase do ciclo, por deslocamento nucleoflico do tRNA do centro P pelo grupo amino do aminoacil-tRNA do centro A. Esta fase designada por transpeptidao catalisada pela enzima peptidil-transferase que estabelece a ligao peptdica entre o grupo amino do aminocido do centro A e o grupo carboxilo do outro aminocido; Posteriormente, ocorre a transferncia do peptidil-tRNA do centro A para o centro P e a sada do tRNA do centro P. Esta deslocao concomitante com o posicionamento do tRNA que perde o aminocido para o centro E (exit). Esta etapa envolve a participao do factor de elongao EF2 (EF-G nas bactrias) que se liga ao ribossoma juntamente com GTP, dissociando-se do ribossoma com hidrlise de GTP, permitindo deste modo o reincio de um novo ciclo de elongao.
3. Terminao
A terminao requer um s factor de dissociao RF, que se liga ao ribossoma juntamente com GTP. na forma associada ao GTP que este factor reconhece o codo de terminao e induz a hidrlise da ligao aminoacil. A ligao de um factor RF ao codo de terminao induz a hidrlise da ligao do polipptido ao tRNA. Este tRNA dissocia-se do ribossoma com hidrlise de GTP e, subsequentemente, o mRNA juntamente com os factores RF-1 e RF-3 dissocia-se do ribossoma, resultando um ribossoma 70S inactivo e pronto a participar novamente na sntese de protenas.
| Captulo 6 Traduo
73
Biologia Molecular - 1 Ano - FFUP
2006/2007
Na E. Coli, os codes de terminao, para os quais no existem tRNA com os correspondentes anticodes, so reconhecidos pelos factores de terminao/dissociao, designados por RF1, RF2 e RF3. O factor RF1 reconhece os codes UAA e UAG e o factor RF2 reconhece UAA e UGA. O factor RF3 liga-se ao GTP e estimula a ligao do RF1 e RF2 ao ribossoma. Por sua vez, o GTP intervm nas alteraes conformacionais e na capacidade de verificao.
4. Polissoma
A distribuio espacial dos ribossomas no citosol ou no RER no completamente aleatria. Muito frequentemente, a anlise por TEM revela a existcnia de agrupamentos ribossomais formando uma espcie de rosrio. Tais estruturas representam vrios ribossomas no processo de traduo de uma mesma molcula de mRNA e designam-se por poliribossomas ou polissomas. A distncia mnima que separa cada um dos ribossomas nestas estruturas cerca de 80 nucletidos. Apesar da complexidade ultrastutural e molecular do ribossoma, foi possvel constatar a presena de vrias zonas especficas de ligao para as diferentes molculas de RNA que com ele interactuam localizadas nas subunidades ribossomais a que foram dados o nome de centros. Um centro para o mRNA, centro este por onde o mRNA desliza medida que vai sendo traduzido. Dois centros recebem tRNA e localizam-se em zonas adjacentes no ribossoma. Um deles o chamado centro P ou centro peptidil que recebe a molcula do tRNA covalentemente ligada extremidade da cadeia polipeptdica em crescimento. O outro, chamado centro A ou centro aminoacil recebe a molcula de tRNA que contm o aminocido que vai ser incorporado na cadeia polipeptdica.
74
Captulo 6 Traduo |
Biologia Molecular 1 Ano - FFUP
2006/2007
5. Inibidores da sntese proteica
Muitos dos mais eficientes antibiticos utilizados na Medicina moderna so compostos produzidos por fungos que inibem a sntese de protena bacteriana. Algumas dessas drogas exploram as diferenas estruturais e funcionais entre os ribossomas bacterianos e eucariticos de forma a interferir preferencialmente com o funcionamento dos ribossomas bacterianos. Tendo em vista que bloqueiam etapas especficas nos processos que levam do DNA protena, muitos dos compostos inibidores so utilizados para estudos de biologia celular. Entre as drogas mais comumente utilizadas em estudos experimentais esto o cloranfenicol, a cicloexamida e a puromicina, todos inibindo especificamente a sntese proteica.
Inibidor Efeito especfico
Com aco apenas em bactrias Tetraciclina Streptomicina Bloqueia a ligao do aminoacil-tRNA ao local A do ribossoma. Evita a transio do complexo de iniciao para um ribossoma capaz de extender a cadeia, podendo tambm causar erros de descodificao. Bloqueia a reaco de peptidil-transferase nos ribossomas. Bloqueia a reaco de translocao nos ribossomas. Bloqueia a iniciao das cadeias de RNA por meio da ligao RNA polimerase, evitando a sntese de RNA.
Cloranfenicol Eritromicina Rifamicina
| Captulo 6 Traduo
75
Biologia Molecular - 1 Ano - FFUP
2006/2007
Com aco em bactrias e em eucariotas Puromicina Causa a libertao prematura das cadeias polipeptdicas em formao por meio de sua adio extremidade da cadeia em crescimento. Liga-se ao DNA e bloqueia o movimento da RNA polimerase, evitando a sntese de RNA.
Actinomicina D
Com aco apenas em eucariotas Cicloexamida Anisomicina -Amanitina Bloqueia a reaco de translocao nos ribossomas. Bloqueia a reaco de peptidil-transferase nos ribossomas. Bloqueia a sntese de mRNA por meio da sua ligao preferencial RNA polimerase II.
5.1. Sentido da sntese proteica
A sntese polipeptdica ocorre com adio sucessiva de aminocidos formando-se a protena no sentido do N-terminal para o C-terminal. Esta ordem na formao da cadeia polipeptdica corresponde a uma leitura do mRNA na direco de 5 para 3, ou seja o primeiro codo a ter correspondncia ao aminocido no N-terminal situa-se prximo da extremidade 5 do respectivo mRNA.
5.2. Dissociao das subunidades ribossmicas
Durante a sntese proteica, existem duas classes de ribossomas que diferem relativamente ao seu peso molecular: os leves e os pesados. Alguns dos ribossomas mais pesados na presena de um excesso de ribossomas leves so adicionados a um tubo de ensaio que contm todos os componentes necessrios para a sntese proteica. Comomentaneamente, num tubo semelhante, so adicionados os mesmos elementos e componentes mas em conjunto com a esparsomicina que actua como produto qumico inibidor da sntese proteica. 76 Captulo 6 Traduo |
Biologia Molecular 1 Ano - FFUP
2006/2007
Como resultado, depois de um perodo curto de incubao, os ribossomas so isolados. Desta forma, quando se comparam os ribossomas de cada um dos tubos, obtm-se resultados distintos para cada um deles. No caso do primeiro tubo, quando a sntese proteica ocorre, so isolados numerosos ribossomas hbridos devido troca de subunidades no interior de cada ribossoma, como por exemplo a formao de ribossomas 30S pesados, 50S leves, etc.. No caso do segundo tubo, quando a sntese proteica bloquada, no pode ocorrer qualquer troca das subunidades ribossmicas e no so isolados nenhuns ribossomas hbridos.
6. Localizao e funo das protenas sintetizadas
Procariotas: Membrana interna transporte e metabolismo de nutrientes Periplasma enzimas hidrolticas e associadas ao transporte de nutrientes Membrana externa transporte de nutrientes e receptores para bacterifagos Eucariotas: Protenas solveis citosol e actuam como locais catalticos; Complexos macromoleculares centrolos; Protenas nucleares cromatina, matriz e lminas nucleares; Organelos citoplasmticos retculo endoplasmtico, complexo de Golgi e lisossomas; Protenas secretadas sequncia lider, sequncia adicional e SRP (sequncia reconhecedora do sinal).
| Captulo 6 Traduo
77
Biologia Molecular - 1 Ano - FFUP
2006/2007
6.1. Sequncia sinal ou leader
As mitocndrias e os cloroplastos sintetizam apenas algumas das suas protenas. As mitocndrias sintetizam apenas cerca de 10 protenas enquanto que os cloroplastos sintetizam aproximadamente 50. A maioria das protenas de organelos so sintetizadas no citosol, pela mesma classe de ribossomas que sintetiza as protenas citoslicas, para serem depois importadas para o interior do organelo. Vrias protenas que entram nas mitocndrias ou nos cloroplastos atravs de processos ps-traduo apresentam sequncias leader que so responsveis pelo reconhecimento primrio da membrana externa do organelo. Assim, esta sequncia inicia um mecanismo de interaco entre o precursor e a membrana do organelo. A protena atravessa a membrana e a sequncia leader clivada por uma protease no lado do organelo.
6.2. Partcula reconhecedora de sinal - SRP
A interaco existente entre a SRP e o receptor para a SRP o evento-chave na traduo eucaritica em transferir um ribossoma que transporta uma protena nascente para a membrana. No caso das bactrias, existe um sistema anlogo, mas o papel desta interaco mais restrito. A SRP um complexo ribonucleoproteico 11S, que contm 6 protenas (de massa total 240 kD) e um peqeuno 7S RNA (cerca de 100 kD, com 305 bases). O 7S RNA da partcula SRP encontra-se dividido em duas partes. As 100 bases na extremidade 5 e 45 bases da extremidade 3 esto aproximadamente relacionadas com a sequncia do AluRNA, uma pequena sequncia de RNA comum nos mamferos, sendo esta poro designada por domnio Alu. A parte restante de RNA compreende o domnio S. As diferentes partes da SRP representadas na figura tm funes diferentes nos processos de endereamento proteico. A SRP54 pode ser atravessada por uma sequncia sinal da protena nascente e directamente responsvel pelo reconhecimento da protena-substrato. O dmero SRP68-SRP72 liga-se regio central do RNA e necessrio para o reconhecimento do receptor da SRP. O dmer SRP9-SRP14 liga-se outra extremidade da molcula, sendo responsvel pelo desenrolar da elongao. 78 Captulo 6 Traduo |
Biologia Molecular 1 Ano - FFUP
2006/2007
6.3. Sequncia adicional ou anchor
As protenas com um domnio transmembranar individual so divididas em duas classes. As protenas do grupo I em que o N-terminal se encontra virado para o espao extracelular so mais comum que as protenas do grupo II em que a orientao foi invertida, de tal modo que o N-terminal esteja virado para o citoplasma. Em ambos os casos, a orientao determinada durante a insero da protena no retculo endoplasmtico.
No caso de as protenas apresentarem domnio transmembranar mltiplo, existem tambm dois casos. Quando a protena atravessa a membrana num nmero mpar de vezes, significa que ambos os terminais da protena esto virados para lados opostos da membrana, enquanto que se atravessar a membrana um nmero par de vezes, significa que os terminais esto virados para o mesmo lado. A extenso dos domnios expostos para um para os dois lados da membrana determinada pelas localizaes dos domnios transmembranares. Assim, os domnios localizados em cada um dos terminais podem ser expostos e as sequncias internas entre os domnios criam um loop externo para o interior do espao extracelular ou do citoplasma.
6.4.
Protenas sintetizadas no retculo endoplasmtico
A ligao dos ribossomas s membranas do retculo endoplasmtico requer uma SRP que neste caso apresenta duas principais capacidades: Pode ligar-se sequncia sinal da protena nascente; Pode ligar-se a uma protena (receptor SRP) localizado na membrana. A SRP e o seu receptor funcionam cataliticamente para transferir um ribossoma que transporta consigo uma protena nascente para a membrana. O primeiro passo o reconhecimento da sequncia sinal pela SRP que, de seguida, se vai ligar ao receptor e induzir a ligao do ribossoma membrana. O papel da SRP na translocao proteica transitrio. Quando a SRP se liga sequncia sinal, permite o decorrer da traduo, sendo que isto geralmente acontece quando cerca de 70 aminocidos foram incorporados na cadeia polipeptdica. Mais tarde, quando a SRP se liga ao seu receptor, liberta a sequncia sinal. O ribossoma passa a estar ligado a um componente da membrana do retculo, permitindo a continuao da
| Captulo 6 Traduo
79
Biologia Molecular - 1 Ano - FFUP
2006/2007
traduo. Quando o ribossoma se encontra anexado membrana, significa que o papel da SRP e do seu receptor j foi desempenhado, podendo agora ser reciclados e livres para auxiliarem um outro polipptido nascente na membrana. O pptido sinal clivado da protena translocativa por um complexo de 5 protenas designado peptidase sinal que est localizado na face do lmen da membrana do retculo endoplasmtico, implicando que a sequncia sinal completa consiga atravessar a membrana antes da clivagem ocorrer. Por fim, a protena final secretada atravs da membrana do retculo endoplasmtico e as subunidades ribossmicas so libertadas da molcula de mRNA.
6.5. Transporte de protenas
As protenas entram no percurso que conduz secreo de acordo com vrias transferncias realizadas com a traduo, atravs da membrana do retculo endoplasmtico. Seguidamente, so transferidas para o complexo do Golgi onde so distribudas para os seus destinos finais, ou para a membrana plasmtica. Existe um conjunto de sinais especficos que induz as protenas a regressar do complexo de Golgi ao retculo endoplasmtico, outros para ficarem retidas no complexo de Golgi, para ficarem retidas na membrana plasmtica ou para serem transportadas para endossomas ou lisossomas.
6.6.
Exocitose e endocitose
As protenas secretadas e as protenas transmembranares iniciam o seu percurso de transpote quando esto translocadas para o interior do retculo endoplasmtico durante a sua sntese. O transporte desde o retculo endoplasmtico, atravs do complexo do Golgi e at membrana plasmtica ocorre em vesculas. Uma protena incorporada numa vescula numa determinada superfcie membranar e libertada da vescula na superfcie seguinte. O processo continua a partir do momento em que a protena altera o seu estado de glicosilao, medida que atravessa o complexo de Golgi, desde os compartimentos cis para os compartimentos trans. 80 Captulo 6 Traduo |
Biologia Molecular 1 Ano - FFUP
2006/2007
As vesculas so utilizadas para transportar protenas tanto para dentro como para fora da clula. A secreo de protenas designa-se exocitose, enquanto que a interiorizao de protenas se designa endocitose. Os percursos para o movimento vesicular so diferentes consoante os mecanismos utilizados e apesar do ciclo de transporte para cada vescula ser similar, estes variam consoante se exportam ou importam protenas.
| Captulo 6 Traduo
81
Biologia Molecular - 1 Ano - FFUP
2006/2007
Captulo 7 Recombinao
A evoluo depende da recombinao gentica. Alterando a posio dos genes, a recombinao permite mutaes favorveis ou desfavorveis, sendo testadas como unidades individuais em novas variedades/combinaes. Isto fornece o significado para a sobrevivncia e propagao dos alelos favorveis e a eliminao dos alelos desfavorveis, sem que os outros genes a que esse alelo est associado sejam prejudicados isto a base para a seleco natural. A recombinao permite: Reparao do DNA; Manipulao de genes; Eliminao de genes mutados; Expresso de protenas diferentes; Determinao da distncia e posio relativa entre genes.
A recombinao, para ocorrer, necessita de sequncias correspondentes do mesmo DNA ou de DNAs diferentes, de DNA de cadeia simples ou de cadeia dupla cortado. Trs tipos de recombinao que partilham o processo que envolve trocas fsicas de material entre cadeias de DNA (ou de RNA que d origem a DNA posteriormente): Recombinao homloga/crossing-over recombinao que envolve a reaco entre sequncias homlogas de DNA. Nos eucariotas, ocorre durante a meiose na altura em que se formam os cromossomas bivalentes envolvendo apenas duas das quatro cadeias (cromatdeos). Recombinao local especfica ocorre entre pares especficos de sequncias. responsvel pela integrao do genoma fgico no cromossoma bacteriano; as sequncias do DNA dador e do DNA receptor tm que ter uma certa homologia. Transposio insero de DNA (originrio de uma cadeia de DNA ou RNA) numa outra cadeia de DNA, sem necessidade de haver homologias nas sequncias. Justifica a movimentao de elementos de um local do cromossoma para outro local.
1. Crossing-over - processos propostos
Ocorre entre duas cadeias duplas de DNA. A principal caracterstica que as enzimas responsveis pela troca, podem usar qualquer sequncia homloga como substrato, embora haja sequncias que so mais favorveis que outras. A frequncia de recombinao no constante durante todo o genoma, mas influenciada por efeitos globais e locais. A frequncia depende tambm da estrutura do cromossoma por exemplo, o crossing-over suprimido nas regies condensadas e inactivas da heterocromatina.
82
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
A recombinao ocorre durante a profase I da meiose.
O incio da meiose marcado pelo ponto em que cada cromossoma se torna visvel. Cada um destes cromossomas foi replicado previamente, constitudo por duas cromossomas, cada um com uma dupla cadeia de DNA. Os cromossomas homlogos aproximam-se e comeam a emparelhar numa ou mais regies formando bivalentes. O emparelhamento extende-se at todo o comprimento de cada cromossoma estar oposto ao seu homlogo emparelhamento de cromossomas. No final, os cromossomas esto todos associados lateralmente na forma de um complexo sinaptonmico que consiste numa estrutura proteica que permite o alinhamento correcto dos cromossomas homlogos que constituem o bivalente (cada gene colocado diante do seu alelo do cromossoma homlogo) e que fornece o suporte necessrio recombinao no estando envolvido no processo. A recombinao entre cromossomas homlogos envolve a troca fsica entre algumas partes, usualmente representadas por quebra e reunio, em que dois cromatdeos no irmos (contendo cada um uma cadeia dupla de DNA) so quebrados e depois se ligam um com o outro. Quando os cromossomas comeam a separar-se, os pontos de quiasmata tornam-se evidentes, sendo que o quiasmata o local onde ocorre a recombinao.
| Captulo 7 Recombinao
83
Biologia Molecular - 1 Ano - FFUP
2006/2007
Este fenmeno ocorre apenas entre sequncias homlogas, sendo possvel ocorrer entre qualquer par, dessas sequncias correspondentes, das duas molculas, de uma maneira muito especfica que permite que o material seja trocado com preciso ao nvel de cada par de bases. Nenhuma sequncia de nucletidos alterada no local de troca; normalmente ocorre alguma replicao de DNA, mas os eventos de clivagem e religao acontecem de modo to preciso que nenhum nucletido perdido ou adicionado. O local de ligao das duas molculas de DNA forma DNA heteroduplex que consiste de uma parte de cada cadeia simples de cada DNA duplo (hbrido). O processo tem como base a complementaridade entre as bases das cadeias simples. A chave para a recombinao entre 2 cadeias diplas de DNA a troca entre as cadeias simples. Inicialmente uma cadeia simples de cada uma das duas duplas cadeias clivada por uma exonuclease, em locais homlogos. Depois uma das cadeias simples desloca a correspondente no outro duplex, criando uma estrutura ramificada, ento as duas duplas cadeias de DNA estaro ligadas especificamente em sequncias correspondentes. Seguidamente podem acontecer dois casos: - os segundos cortes ocorrem nas mesmas cadeias e ento no se obter uma recombinao genmica, mas as duplas cadeias contero regies com heteroduplex; - os segundos cortes ocorrem nas cadeias que ainda esto intactas e segue-se o processo descrito acima, em que 1 cadeia desloca a cadeia simples do duplex homlogo. No final, os cortes so selados por interveno de enzimas e obtm-se genomas recombinantes recprocos, (ou seja, cada cadeia deixa a sua complementar para se ligar complementar do outro duplex)
1.1. Corte da dupla cadeia
Conceitos gerais: - A recombinao iniciada efectuando uma quebra na dupla cadeia numa dupla hlice de DNA; - Aco da exonuclease gera uma cadeia simples com terminal 3, permitindo a invaso na outra dupla cadeia de DNA.
84
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
- A nova sntese de DNA substitui o material que foi degradado. - Isto gera uma juno da molcula recombinante juno de Holliday - em que as duas duplas cadeias de DNA esto conectadas pelo DNA heteroduplex.
Ambas as cadeias do DNA devem ser quebradas para ocorrer uma troca gentica. Anteriormente abordou-se um modelo em que as quebras das cadeias individuais ocorriam sucessivamente. Contudo, a troca gentica iniciada pela quebra da dupla cadeia, como ilustra a figura. Uma endonuclease cliva uma das duas duplas cadeias de DNA (o receptor), o corte ampliado para uma abertura por aco de uma exonuclease, formando em cada cadeia simples um terminal 3. Um desses terminais livres invade uma regio homloga na outra dupla cadeia de DNA (o doador) este passo designa-se por invaso da cadeia simples. A formao do heteroduplex forma um D-loop, onde uma das cadeias da cadeia dupla (doadora) deslocada (ou seja, a cadeia simples invasora desloca a sua homloga). O D-loop prolongado pela sntese de reparo de DNA, usando o terminal 3 livre como local para produzir o PRIMER para regenerar a dupla cadeia de DNA. Eventualmente o D-loop torna-se to amplo de modo a corresponder ao tamanho da abertura provocado no primeiro DNA duplo (o receptor). Quando a cadeia deslocada alcana o lado distante da abertura, as cadeias complementares ligam-se. Neste momento, h DNA heteroduplex nos dois lados da abertura. A integridade das duplas cadeias no local de abertura pode ser restaurada pelo reparo de sntese usando o terminal 3 livre do lado esquerdo da abertura como Primer. A migrao de braos converte esta estrutura numa molcula com duas junes recombinantes que devem ser resolvidas atravs de corte.
| Captulo 7 Recombinao
85
Biologia Molecular - 1 Ano - FFUP
2006/2007
Diferenas entre o modelo de cadeia simples e de cadeia dupla:
Segundo a quebra da dupla cadeia, DNA heteroduplex foi formado em cada extremidade da regio envolvida na troca. Entre os dois segmentos de heteroduplex, a regio que corresponde abertura, a qual agora tem a sequncia do DNA doador (nas duas molculas). Ento o arranjo do DNA heteroduplex assimtrico, e parte de uma molcula que foi convertida na sequncia do outro. Seguindo o modelo da troca de cadeias simples recproco, cada cadeia dupla de DNA tem um heteroduplex que cobre a regio desde o stio inicial da troca at ramificao migrada. Nas variantes do modelo da troca de cadeia simples, em que o DNA degradado e ressintetizado, o cromatdeo iniciante o doador de informao (ao contrrio da quebra dupla de cadeia) No modelo da troca de cadeia simples no h perda de informao quando o corte efectudo, ao contrrio d modelo de dupla cadeia que esta associada perda de informao. Outro processo proposto para o crossing-over, alm daquele j descrito quebra e reunio h tmabm o processo escolha de cpia, contudo este processo no possvel, uma vez que no segue a replicao segundo o modelo semi-conservativo. Para admitir o processo de copy choice teria de se admitir o modelo conservativo (coisa que no se pode fazer) e consistiria no seguinte: A sntese dos cromatdeos-irmos de cada um ds cromossomas homlogos; Troca da sntese copiando o outro cromatdeo (do cromossoma homlogo), reciprocamente.
NOTA: copy choice usado pelos retrovrus polimerase troca de um lado para o outro enquanto est a sintetizar o RNA, dando origem a uma molcula com uma sequncia de informao de vrias partes.
Ento, durante a meiose, temos dois casos possveis: 1 - ocorrncia de crossing-over: d origem a metade dos gmetas com cromossomas no modificados e a outra metade dos gmetas tem cromossomas modificados/recombinantes. 2 - no ocorrncia de crossing-over: todos os gmetas tm o mesmo genoma.
86
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
2. Estudo da recombinao com istopos pesados
Uma bactria, que cresce num meio leve, infectada por dois tipos de stock de fagos lambda, um stock foi preparado por um crescimento do fago lambda ABC em clulas contendo no meio istopos pesados. O outro stock de fagos lambda abc cresceu num meio com istopos leves. Portanto a bactria infectada por material gentico pesado (com istopos pesados) e material gentico leve. Durante o crescimento, h replicao do material genmico fgico e como o meio em que a bactria se encontra leve as cadeias pesadas passam a ser hbridas. Depois da infeco isola-se os progeny e faz-se uma centrifugao por densidade em meio CsCl. Observa-se que existem duas bandas: uma bem pronunciada e a outra menos pronunciada. A mais superior pertence aos constitudos por cadeias leves. As bandas intermdias pertencem aos fagos que sofreram recombinao. Observou-se tambm que os fagos que continham o gentipo abC encontravam-se mais prximos da banda dos fagos com cadeia leve e os que tinham um gentipo ABc encontravam-se mais prximos da zona de maior densidade, por conterem uma maior quantidade de istopos pesados. Deste modo, conclui-se que a recombinao entre dois fagos originais envolve a quebra e a reunio de ambas as cadeias de DNA.
3. Independncia entre recombinao e replicao
Uma bactria infectada por dois tipos de fagos que cresceram em diferentes meios. Um stock cresceu num meio com istopos pesados e tem genoma AB; o outro stock cresceu num meio leve e tem genoma ab. Durante a infeco da bactria, que se encontra num meio leve, a maior parte das cadeias pesadas passam a ser hbridos devido duplicao do DNA. Contudo, muito raramente, h cadeias que no replicam e permanecem intactas. Quando os novos fagos so formados, estas formas no-replicadas so includas nas novas cpsulas proteicas, incapacitando-nos de concluir se so sempre recombinantes. Ao fazer a centrifugao por densidade destas novas partculas fgicas, verificam-se 3 zonas/bandas. - uma mais superior que corresponde aos fagos com cadeias leves. - uma zona intermdia que correpsonde aos fagos com cadeias que contm alguns tomos pesados. - uma banda mais baixa que representa os fagos que contm as raras cadeias que no replicaram, mas que contudo recombinaram sendo se dois tipos Ab e aB.
| Captulo 7 Recombinao
87
Biologia Molecular - 1 Ano - FFUP
2006/2007
Deste modo conclui-se que crossing-over ocorre em cadeias intactas e que a sntese extensiva de DNA no est envolvida no crossing-over.
4. Recombinao em E. Coli
Esta recombinao ocorre entre regies homlogas do DNA, ou seja, muito semelhantes (mx. Aceitvel de diferena de 10%)
88
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
4.1. Protenas intervenientes
Protena RecA: Promove o emparelhamento de bases entre o DNA de cadeia simples e o complementar de cadeia dupla. Tem actividade protesica na resposta SOS e requer ATP. Ambas as actividades so activadas por cadeia simples de DNA na presena de ATP. Aco: liga-se fortemente e em extensos grupos cooperativos a segmentos de DNA de cadeia simples, formando um filamento nucleoproteico. Como cada monmero de RecA pode possuir mais que um stio de ligao ao DNA, um filamento de RecA pode interagir com uma cadeia simples e com uma dupla hlice, mantendo-as juntas. Protenas RecBCD: Promovem o desenrolamento do DNA com uma extremidade livre na dupla cadeia; Reconhecem a sequncia Chi (5-GCTGGTGG-3); Tm actividade nuclesica (nuclease e helicase); Formam estruturas designadas por orelhas de coelho. As bactrias no costumam trocar grandes quantidades de DNA duplo, mas a recombinao pode ocorrer de diferentes maneiras. Em alguns casos, o DNA pode estar disponvel na forma de cadeia simples com terminal 3; pode ser fornecido na forma de pequenos fragmentos de cadeia simples, resultantes do efeito da radiao; terminaes em cadeia simples tambm podem ser originadas pelos genomas fgicos submetidos replicao por crculos rolantes. Contudo, h circunstncias que envolvem DNA duplo (como p.ex. na recombinao durante a meiose nos eucariontes), sendo necessrio criar regies de cadeia simples e terminais 3. O mecanismo de gerar terminais de cadeia adequados s molculas, para que possa ocorrer recombinao, a existncia de certos locais que estimulam a recombinao. Estes locais partilham uma sequncia constante no simtrica de 8bp ( 5-GCTGGTGG-3) e denomina-se por Chi. (3-CGACCACC-5)
Uma sequncia Chi estimula a recombinao numa vizinhana de mais 10 kb do local da sequncia inicial. Este local Chi pode ser activado por uma quebra (da dupla cadeia) feita vrias kb de distncia mas do lado direito da sequncia em questo. Esta dependncia de orientao sugere que o instrumento de recombinao deve-se associar ao DNA no local de quebra e que depois consegue remover-se ao longo da cadeia dupla num s sentido. De facto o que acontece como se vai verificar a seguir: Os locais Chi so alvos para a aco enzimtica do complexo enzimtico codificado pelos genes RecBCD.
| Captulo 7 Recombinao
89
Biologia Molecular - 1 Ano - FFUP
2006/2007
Este complexo enzimtico tem actividade de nuclease (degrada sequencias de nucletidos) de helicase (abre a dupla cadeia) e de ATPase. O seu papel na recombinao providenciar uma regio de cadeia simples com terminal 3 livre. 1 - RecBCD liga-se ao DNA no lado direito do local Chi e move-se ao longo da dupla cadeia desenrolada e degrada DNA da cadeia 3 (aco nuclesica). Quando chega ao local Chi, pra e cliva uma cadeia de DNA na posio 4 e 6 bases do lado direito do Chi. Esse local direita da sequncia Chi reconhecido como uma forma de cadeia simples. O reconhecimento da sequncia Chi leva dissociao da subunidade ou inactivao RecD, perdendo a funo da nuclease, contudo continuam a abrir a cadeia com a funo de helicase.
4.1.1. Aco da RecA
A actividade manipuladora de DNA da RecA permite uma cadeia simples deslocar a sua homloga numa cadeia dupla numa reaco denominada assimilao de cadeia simples, contudo esta reaco tem trs condies gerais: 1 - uma das cadeias de DNA tem de ter uma regio de cadeia simples 2 - uma das cadeias tem de ter terminal da cadeia 3 livre 90 Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
3 - a regio de cadeia simples e o terminal 3 devem ser situados dentro de uma regio que seja complementar entre as molculas. A RecA activa sobre o substrato gerado prlo RecBCD, ou seja, sobre a cadeia simples com terminal 3, usando-a para reagir com a sua homloga do DNA duplo, criando assim uma molcula comum (incio da formao de DNA hteroduplex). H seis monmeros da RecA por cada volta do filamento, o qual tem a forma de estrutura de hlice com um grande sulco que contm. Cada monmero liga-se a trs nucletidos. Quando a dupla cadeia de DNA ligada, ela contacta com a RecA pelo menor sulco. Depois h um intermedirio de 3 cadeias onde isto permite que a RecA catalise a reaco de assimilao de cadeia simples e de sinapse de DNA de vrias etapas entre uma dupla hlice e uma regio de cadeia simples de DNA homologa. A regio de homologia identificada antes da dupla-hlice ter sido clivada, por meio de um intermedirio de 3 cadeias, no qual a cadeia simples de DNA forma um emparelhamento temporrio com bases que se projectam do sulco maior da hlice da molcula de cadeia dupla. A interaco entre as duas molculas de DNA ocorre dentro desses filamentos. A RecA aumenta a sua actividade usando hidrlise de ATP. O ATP liga-se num centro alostrico desta protena induzindo alteraes na sua conformao. Quando ligada ao ATP, o local de ligao de DNA tem uma grande afinidade para esse, sendo condio requerida para o DNA se ligar RecA e para a reaco de complementaridade entre as cadeias. A hidrlise de ATP converte o local de ligao para ter pouca afinidade, requisito para libertar o DNA heteroduplex. Pode-se dividir a reaco de catlise mediada pela RecA, entre a cadeia simples de DNA e o DNA duplo em 3 fases: 1- RecA polimeriza no DNA de cadeia simples; 2- Rpida reaco de pareamento entre o DNA de cadeia simples e a sua homloga de cadeia dupla, produzindo um DNA heteroduplex; 3- Deslocamento lento de uma cadeia do duplex (produzindo uma longa regio para formar um heteroduplex.)
Uma vez que a sinapse tenha ocorrido, a pequena regio de heteroduplex onde as cadeias de das diferentes molculas de DNA iniciaram a ligao aumentada por um processo denominado por migrao de ramificao. A protena RecA catalisa a migrao unidireccional, produzindo prontamente uma regio de heteroduplex de DNA com milhares de pares de bases. Esta catlise depende de uma outra propriedade desta protena que a de ser uma ATPase DNA-dependente, tendo portanto alm de dois stios para a ligao s cadeias de DNA, um stio adiconal para a ligao e hidrlise de ATP.
| Captulo 7 Recombinao
91
Biologia Molecular - 1 Ano - FFUP
2006/2007
NOTA: Quando uma molcula de DNA de cadeia simples reage com uma dupla, esta torna-se desenrolada no local da recombinao. A regio inicial de DNA heteroduplex consiste em 2 cadeias associadas lado a lado, sendo denominada por paranemic joint, sendo instvel, que com o progresso da reaco requer a sua converso na forma de dupla hlice. A imagem apresentada demonstra a importncia da extremidade 3 da regio de cadeia simples, ela que se vai ligar, iniciando a reaco. A assimilao inicia-se no final da cadeia linear (extremidade 3) onde a cadeia simples invade a dupla deslocando a sua homloga no duplex. Mas quando a reaco chega regio que h duplex nasduas molculas a molcula complementar original molcula invasora, reage com a molcula deslocada do outro duplex, obtendo-se no final molculas duplas recombinadas.
4.1.2. Troca de cadeias promovida pela RecA in vitro
Esta situao envolve 3 cadeias de DNA. O filamento de RecA est ligado ao DNA de cadeia simples circular, o qual, vai deslocar a cadeia homloga da molcula de DNA duplo linear, homlogo. A migrao ramificada (branch migration), faz com que se obtenha (aps a sada do filamento de RecA) um DNA circular recombinado mais uma cadeia simples (o que foi deslocado do duplex original)
92
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
Por sua vez, este caso envolve 4 cadeias de DNA, ou seja, duas cadeias duplas de DNA circular e outra linear. A circular contmuma regio de cadeia simples de modo a iniciar a reaco. O filamento de RecA liga-se ao DNA circular (o que contm o ssDNA single stranded DNA). Ao ligar-se com o DNA duplo linear homlogo, ocorre o deslocamento da cadeia homloga e esta emparelha coma sua complementar do DNA circular, formando uma estrutura de Holliday. Ocorre a migrao ramificada. No final (aps a sada das protinas RecA), obtm-se um DNA duplo circular recombinado e um DNA duplo linear recombinado.
Fase I formao do filamento da RecA Fase II alinhamento das regies homlogas Fase III formao e extenso do DNA heterocuplex Fase IV hidrlise de ATP (sada de RecA)
5. Estrutura de Holliday e heteroduplexes
5.1. Estrutura de Holliday
A estrutura de Holliday uma estrutura intermediria que se forma na recombinao de dois DNAs de cadeia dupla e que formada por uma cadeia de cada molcula de DNA. Na juno de Holiday, as duas hlices de DNA homlogos que foram inicialmente alinhados so mantidos unidos pela permuta recproca de duas das quatro cadeias presentes no evento, uma de cada uma das hlices, ou seja, contm um par de cadeias cruzadas e um par de cadeias no cruzadas. Essa estrutura pode sofrer isomerizao, por meio de uma srie de movimentos rotacinais catalisados por enzimas especializadas. Para regenerar as duas hlices de DNA separadas e, ento terminar o processo de permuta, as cadeias que unem as duas hlices na estrutura de Holliday devem ser clivadas num processo denominado resoluo. Existem duas maneiras de resolver a estrutura de Holliday: 1. o par original de cadeias cruzadas clivado as duas hlices de DNA originais so separadas entre si de modo quase inalterado, trocando apenas a cadeia simples formada na heteroduplex. 2. o par original de cadeias no cruzadas clivado o resultado mais significativo: 2 cromossomas recombinantes so formados com grandes segmentos de Dna de cadeia dupla permutados de modo recproco entre si, por meio de crossing-over.
| Captulo 7 Recombinao
93
Biologia Molecular - 1 Ano - FFUP
2006/2007
Quando a recombinao ocorre entre duas cadeias duplas de DNA, cada uma das cadeias simples desloca a sua homloga na outra cadeia e liga-se sua complementar, ao ocorrer isto, a troca recproca, h a criao de uma conexo entre os dois DNA duplos, sendo designada por: joint molecule (juno da molcula). O ponto a que cada cadeia individual de DNA atravessa da sua molcula para o outro duplex, designa-se por juno recombinante.
5.2. Heteroduplexes
Em cada local de recombinao, cada duplex tem uma regio que consiste numa porode cadeia de uma das molculas de DNA originais denomina-se por heteroduplex DNA. (DNA de cadeia dupla, em geral como produto das recombinantes, formado por cadeias complementares derivadas de 2 molculas de DNA com sequncias semelhantes.)
94
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
5.2.1. Heteroduplexes e migrao de braos
Migrao de braos - capacidade de uma cadeia de DNA, DNA, emparelhada com a sua complementar num duplex, em estender e deslocar a cadeia residente da qual homloga. A pequena regio de heteroduplex onde as cadeias das diferentes molculas de DNA iniciaram a reaco pode ser aumentada por um processo denomina denominado do por migrao deramificao de braos. A migrao pode ocorrer em qualquer local das cadeias de DNA com sequncias homlogas que esto a tentar estabelecer ligao com a cadeia complementar, nesta reaco, uma regio que est ligada/ pareada de uma das cadeias adeias simples desloca uma regio que est ligada da outra cadeia,movendo o ponto de ramificao / o local onde ocorre crossing crossing-over, over, sem alterar o nmero total de pares de bases do DNA. Esta migrao pode ocorrer espontaneamente, porm, como procede igualmente igualmente para ambas as direces (esquerda e direita), o seu deslocamento pequeno e incapaz de completar a recombinao eficientemente. Tambm a migrao pod ser catalizada pelas protinas RecA, sendo portanto a migrao unidireccional (para a direita), produzindo oduzindo prontamente uma regio de heteroduplex de DNA com milhares de pares de bases.
A migrao de braos importante por razes tericas e prticas. Como princpio, ela confere dinamismo nas estruturas de recombinao. Como caractestica prtica, a sua sua existncia significa que o local de ramificao (no incio da recombinao) no pode ser estabelecido por examinao de uma molcula in vitro, pois a ramificao pode ter migrado desde que a molcula foi isolada.
| Captulo 7 Recombinao
95
Biologia Molecular - 1 Ano - FFUP
2006/2007
5.3. Funes dos genes ruvA, ruvB e ruvC
O complexo ruv actua nas junes recombinantes e nas estruturas de Holliday. Um dos pontos mais crticos na recombinao a resoluo da estrutura de Holliday, a qual determina se se d uma recombinao recproca ouse se origina uma dupla cadeia com uma pequena regia de heteroduplex. A migrao de braos determina o comprimento da regio de DNA hbrida (com ou sem recombinao). As protenas envolvidas na estabilizao e reoluo da estrutura de Holliday foram identificadas como produtos dos genes Ruv na E.Coli.
RuvA aumenta a formao de heteroduplexes reconhece a estrutura de Holliday liga-se s 4 cadeias de DNA no ponto de crossing-over, formando dois tetrmeros de cada lado. RuvB aumenta a formao de heteroduplexes tem actividade helicase + ATPase (que catalisa a migrao de braos) hexamrica liga-se volta de cada hlice de DNA acima do ponto de recombinao. O complexo RuvAB usa a ramificao para migrar 10 a 20 bp/seg. Desloca a RecA do DNA durante a sua aco. RuvC tem actividade de endonuclease resolve a juno da estrutura de Holliday
Uma sequncia tetranucletida comum promove o local para a ao da RuvC resolvendo a estrutura de Holliday. O tetranucleotdeo (ATTG) assimtrico e poe assim dirigir a resoluo tendo em condiderao qual ser o par de cadeias a ser cortado. Isto determina se o resultado final patch recombination no ocorre recombinao clivagem das cadeias cruzadas ou slice recombination ocorre recombinao das cadeias no cruzadas.
96
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
6. Recombinao durante a meiose
A meiose o processo que d origem aos gmetas, ou seja, o processo onde h resuo para metade do caritipo, passa de diplide para haplide, havendo separao dos cromossomas matenos e paternos, que so homlogos. No incio da meiose, quando o material gentico j est duplicao, os cromossomas homlogos alinhamse e ligam-se ao complexo sinaptonmico, local onde as permutas do material ocorrem, ou seja, onde ocorre recombinao geral crossing-over.
Mais detalhadamente, existem as seguintes fases: Profase I Leptteno Inicia-se a condensao dos cromossomas Visualizao dos crommeros e dos nuclolos Formao das placas de adeso Zigteno Emparelhamento ou sinapse dos cromossomas homlogos Formao do complexo sinaptonmico Formao de bivalentes (2 cromossomas homlogos juntos) ou ttradas (4 cromatdeos)
| Captulo 7 Recombinao
97
Biologia Molecular - 1 Ano - FFUP
2006/2007
Paquteno Sinapse total dos cromossomas homlogos Formao de ndulas recombinantes ao longo do complexo sinaptonmico Ocorre crossing-over Diplteno Cromossomas homlogos comeam a repetir-se, levando dissoluo do complexo sinaptonmico Os cromossomas homlogos permanecem unidos por quiasmas (locais onde ocorreu crossingover) Diacinese Condensao total dos cromossomas Desagregao da membrana nuclear e dos nuclolos Paragem de sntese de RNA Telomerizao movimentao dos quiasmas para as extremidades dos cromossomas (telmeros) Seguidamente, na metafase I, h formao do fuso acromtico e os bivalentes alinham-se no equador da clula, formando a placa equatorial. Nesta fase, j as trocas de material gentico esto concretizadas. Seguidamente, d-se a anafase I, que separa os bivalentes, indo a linha paternal para um lado e a maternal para o outro. Depois, d-se a telofase I, dando origem a duas clulas, cada uma com um cromossoma, constitudo por dois cromatdeos. Posteriormente, do-se as fases da segunda diviso meitica, obtendo-se quatro clulas haplides, podendo conter ou no cromossomas recombinantes, isto pois primeiramente quando a recombinao ocorre, ocorre apenas num dos cromatdeos do cromossoma, logo duas das clulas contero o cromatdeo que no recombinou ou ento ocorreram dois crossing-over entre dois genes, produzindo cromossomas no-recombinados. Por exemplo, tomando dois cromossomas homlogos identificando dois genes para estudo:
98
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
Quando ocorre um duplo crossing-over entre dois genes obter-se-o gentipos no recombinantes nos gmetas, pois o segundo crossing-over inutilizou o que o primeiro realizou. Para exemplificar a ocorrncia de cromossomas recombinantes admitindo trs genes:
6.1. Converso gentica
A converso gentica um processo no recproco, que pode ocorrer na recombinao, em que um alelo convertido noutro, por emparelhamento incorrecto. Ocorre associada a eventos de recombinao homloga na meiose (e mais raramente na mitose) e acredita-se que seja uma consequncia directa do mecanismo de recombinao geral e de reparo de DNA. Quando a recombinao entre genes alelos foi descoberta, assumiu-se que ocorreria o mesmo processo de recombinao recproca. Isto aplica-se a loci distantes e quer dizer que um evento individual de quebra e reunio dentro do locus ocorre para gerar um par recproco de cromossomas recombinados. Contudo, nos quartos mais prximos de um nico gene, a formao por si s de um heteroduplex usualmente responsvel pelo evento de recombinao. A meiose num heterozigtico devia gerar quatro cpias de cada alelo (dois de cada progenitor). Tomando em considerao, um estudo sobre a formao de esporos de uma espcie de fungos, conclui-se que esse fenmeno ocorre, a maior parte das vezes, obtendo-se uma relao de 2:2. Contudo, h
| Captulo 7 Recombinao
99
Biologia Molecular - 1 Ano - FFUP
2006/2007
excepes, em que a relao no equitativa, mas sim de 3:1, sendo explicada pela ocorrncia de formao e correco de DNA heteroduplex na regio em que os alelos diferem converso gentica. No processo de converso gentica, a informao da sequncia de DNA transferida de uma hlice de DNA que permanece inalterada, para outra hlice de DNA, cuja sequncia alterada. Este facto envolve dois processos principais: Como indica o modelo da quebra de dupla cadeia, uma cadeia dupla de DNA actua como doadora da informao gentica que substitui directamente as sequncias correspondentes no outro duplex por um processo de gerao de espao corte troca de cadeia e preenchimento isto proporcionado por um trabalho de enzimas, entre as quais as que catalisam a reparao, nomeadamente de bases no complementares entre os heteroduplexes, removendo-as e promovendo uma cpia extra da sequncia de DNA da cadeia oposta. Como parte do processo de troca, o DNA heteroduplex gerado quando uma cadeia simples de um DNA duplo complementa com a sua complementar do outro DNA duplo. O sistema de reparao de bases no-complementares no DNA heteroduplex e procede exciso e substituio de uma das cadeias por complementaridade, convertendo um alelo noutro uma quantidade limitada de sntese localizada de DNA.
A converso de genes no depende do crossing-over mas est relacionada com esse fenmeno.
7. Mecanismos resultantes da recombinao
A recombinao entre dois stios de uma molcula de DNA pode ter consequncias possveis, dependendo de como os stios envolvidos esto orientados. A recombinao pode remover segmentos intermedirios (deleco) ou invert-los (inverso). As clulas, em determinadas situaes, utilizam a inverso recombinacional para escolher entre dois arranjos alternativos de DNA, que permitem que diferentes protenas ou grupos de protenas sejam expressos.
7.1. Inverses e deleces
Se os genes recombinantes esto na forma invertida em vez da orientao normal, h uma inverso em vez de uma deleco (ocorre em cpias de repetio directa). Ento, ocorre uma quebra da dupla cadeia. Haver uma poro de DNA intermedirio, entre as pores a sofrer recombinao. Os terminais do fragmento denominam-se por terminais de sinal. Os terminais clivados das partes a recombinar so designados por unidades codificantes. As duas unidades ligam-se covalentemente, formando uma juno molecular, se os terminais de sinal tambm se conectarem, o fragmento excisado formar uma molcula circular.
100
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
Na inverso, ocorre na mesma uma quebra e juno molecular, mas o fragmento intermedirio em vez de ser excisado invertido. Portanto, a recombinao entre dois stios de uma determinada molcula de DNA pode ter duas consequncias possveis, dependendo de como os stios envolvidos esto orientados. A recombinao pode remover segmentos intermedirios ou invert-los. As clulas, em alguns momentos, utilizam a inverso recombinacional para escolher entre dois arranjos alternativos de DNA. Um exemplo bem estudado envolve a expresso alternada de duas protenas flagelares da bactria Salmonella, chamadas H1 e H2, que nunca so expressas simultaneamente. A regio do DNA com repeties (26 pb) orientadas em direces opostas ao redor do promotor do gene H2 fica frente deste gene, permitindo a sua transcrio, juntamente com outro gene que codifica uma protena que reprime a expresso do gene da protena H1, ou ento, fica escondido antes do loci Hin, permitindo a expresso do gene H1. O segmento que invertido codifica a enzima Hin, que catalisa a inverso.
Ao haver a inverso do segmento correspondente ao promotor, a RNA polimerase sintetiza um mRNA com informaes diferentes e ocorre que dados genes deixem de ser expressos. No caso da deleco, a expresso dos genes removidos fica comprometida.
| Captulo 7 Recombinao
101
Biologia Molecular - 1 Ano - FFUP
2006/2007
7.2. Duplicaes e deleces
Geralmente, estes mecanismos so resultantes de crossing-over ilegtimo, que ocorre durante a replicao do DNA, entre seces homlogas do DNA de cromossomas-filhos. Por outro lado, o crossing-over no equivalente ocorre entre zonas de tal forma que um cromossoma perde carga gentica e o outro ganha.
8. Recombinao local-especfica
Esta recombinao envolve a reaco entre dois stios especficos, sendo mediada por enzimas de exciso e integrao que reconhecem pequenas sequncias nucleotdicas especficas as enzimas que catalisam este tipo de recombinao so as recombinases (que pertencem famlia das integrases). Os stios-alvo da recombinao local-especfica so pequenos (14 50 pb), sendo que em alguns casos, os dois stios tm a mesma sequncia e noutros casos, so homlogos.
8.1. Recombinao entre o fago e a E.Coli
A converso do DNA entre as suas duas formas de vida envolve dois tipos de eventos. A sua condio fsica diferente do estado lisognico e ltico: No estado ltico DNA lambda existe numa forma circular independente na bactria. No estado lisognico DNA fgico () uma parte integrante do cromossoma bacteriano (designa-se por pr-fago)
A transcrio entre estes estados envolve recombinao local especfica: Para entrar na condio lisognica, o DNA fgico independente tem de ser inserido no DNA hospedeiro integrao. Para sair do estado lisognico para o ltico, o DNA pro-fgico tem de ser libertado do cromossoma bacteriano exciso.
A integrao e a exciso ocorrem por recombinao em loci especficos no DNA bacteriano e DNA fgico chamados por locais attachment att. Para descrever as reaces de integrao/exciso o local attachment bacteriano chamado por attB, consistindo por sequncia BOB. O local attachment do fago attP - consiste de sequncia POP. Os stios attB e attP so dissimilares mas contm uma regio central idntica, a regio 0 (15bp) e o evento de recombinao ocorre dentro dela. A recombinao e responsvel por tornar o DNA fgico circular numa sequncia 102 Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
linear como o cromossoma bacteriano. O profago limitado por dois locais novos do att (produtos da recombinao) chamados attL e attR (com sequncias BOP e POB respectivamente). Uma importante consequncia da constituio dos locais att que as reaces de integrao e exciso no envolvem o mesmo par de sequncia. A integrao requer o reconhecimento entre attP e attB e s ocorre porque o genoma fgico sintetiza a integrase e a E.Coli o factor IHF (integration host factor). A exciso requer o reconhecimento entre attL e attR, ou seja, ocorre por recombinao recproca, do factor Xis excisionase e integrase sintetizadas pelo lambda e do factor HIF sintetizado pela E.Coli. A exciso permite que saia apenas o genoma fgico. O carcter direccional da recombinao local especfica controlado pela identidade dos locais recombinantes. Embora a recombinao seja reversvel, diferentes condies prevalecem em cada direco da reaco. Isto uma caracterstica importante para o ciclo fgico, pois oferece um seguro de que um evento de integrao no imediatamente reversvel por uma exciso e vice-versa.
A recombinao entre attP e attB ocorre pelo corte da dupla cadeia. As cadeias correspondentes de cada duplex so cortadas na mesma posio, o terminal 3 livre troca entre os duplexes. A ramificao migra numa distncia de 7 bp ao longo da regio de homologia e depois a estrutura resolvida cortando o outro par das cadeias correspondentes.
8.2. Recombinao entre fagos mutantes
Dois fagos podem infectar simultaneamente uma bactria se forem da mesma espcie. Ao infectar uma bactria com dois fagos que apresentam mutaes em alelos diferentes verifica-se que por recombinao se obtm:
| Captulo 7 Recombinao
103
Biologia Molecular - 1 Ano - FFUP
2006/2007
- um com 2 alelos mutantes no cresce. - um sem qualquer alelo mutante (= selvagem) cresce
Note-se que os dois fagos em separado, devido sua mutao no crescem.
8.3. Recombinao por transposies
A recombinao por transposio pode ser simples ou replicativa. Consiste na insero de um elemento transposicional no DNA-alvo com sequncias no homlogas. Transposes - movimentam-se atravs de um intermedirio de DNA. O transposo geralmente codifica a protena- enzima especfica transposase, que actua sobre uma sequncia especfica do DNA presente em cada uma das extremidades do transposo inicialmente desconectando-a do DNA adjacente e depois inserindo-a num novo DNA alvo, no havendo necessidade de homologia entre as extremidades do elemento e o local de insero, contudo ele insere-se numa sequncia qual tenha maior afinidade.
O elemento de transposio termina com pequenas sequncias repetidas invertidas, e so capazes de gerar pequenas sequncias directas no local de insero.
104
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
Retrotransposes so derivados do retrovrus. So capazes de inserir cpias de DNA (proviroses) a partir de RNA logo, a sua mobilidade mediada por um intermedirio de RNA. A poro de RNA pela aco da transcriptase reversa e DNA polimerase toma a forma de DNA, podendo agora ser inserida no DNA-alvo.
A integrase retroviral corta a sequncia LTR do DNA retrovrico em locais especficos e complementares do DNA-alvo (onde tambm a sequncia clivada). Ocorre uma ligao posterior ao DNA-alvo e duplicao das sequncias do DNA- alvo para os quais tem mais afinidade de retrotransposio.
8.3.1. Transposio simples
Ocorre corte total do transposo a inserir no DNA-alvo. Posterior ligao das extremidades livres do elemento de transposio com as sequncias do DNA-alvo para as quais tem maior afinidade. Sntese dos fragmentos de DNA do DNA-alvo.
8.3.2. Transposio replicativa
Ocorre o corte de duas extremidades do elemento de transposio originando a extremidade 3OH para se ligar ao DNAalvo em locais especficos. Posterior ligao ao DNA-alvo do elemento de transposio. A parte do DNA que tem o elemento a inserir tambm ficava ligada ao DNA-alvo por intermdio do elemento de insero. A replicao de cada cadeia do elemento de transposio. As cpias obtidas ficam integradas entre si. A enzima resolvase liga-se a sequncias especficas (local res) de cada cpia do elemento de transposio. Faz a recombinao entre as cpias e o elemento de transposio dissocia a estrutura co-integrada.
| Captulo 7 Recombinao
105
Biologia Molecular - 1 Ano - FFUP
2006/2007
9. Complementao
No envolve de gentipos! Corresponde capacidade dos genes independentes fornecerem produtos difusveis. Estes produtos restauram o fentipo selvagem quando dois mutantes so testados na configurao trans.
Processo: infectar bactria com dois fagos da mesma espcie mas com mutaes diferentes e em locais diferentes. Dado terem mutaes estes fagos crescem se se complementarem entre si, h duas hipteses:
1. Se os produtos que cada um forma (apesar da mutao) complementa os produtos que o outro forma h crescimento dos fagos. 2. Se os produtos no se complementam no h crescimento dos mesmos.
Esta anlise dos produtos difusveis permite verificar se: - determinadas mutaes esto no mesmo gene ou em genes diferentes; - uma regio do cromossoma tem um ou mais genes, em funo de um ou mais produtos produzidos.
9.1. Complementao e recombinao
Ento, como j foi referido: - na complementao NO h troca de gentipos. - na recombinao H troca de gentipos.
106
Captulo 7 Recombinao |
Biologia Molecular 1 Ano - FFUP
2006/2007
Teste cis-trans:
|/
107
Biologia Molecular - 1 Ano - FFUP
2006/2007
Captulo 8 Mapeamento
O mapeamento dos genes refere-se localizao especfica de genes nos cromossomas. um importante passo na compreenso das doenas genticas. Existem dois tipos de mapeamento de genes: mapeamento gentico e mapeamento fsico.
1. Mapa gentico
Mapeamento gentico utiliza a anlise das ligaes a anlise das ligaes envolvidas na molcula, para determinar a posio relativa entre dois determinados genes num cromossoma. Mapa gentico corresponde sequncia de genes num cromossoma; representa a localizao/posio relativa dos genes e/ou marcadores genticos dentro de um fragmento de DNA. Permite compreender as consequncias fenotpicas das alteraes que ocorrem nos cromossomas, uma vez que atravs deste se podem determinar quais os genes que podem vir juntos com uma certa caracterstica que est a ser seleccionada. E compara o contedo da informao gentica de um cromossoma em espcies diferentes.
108
Captulo 8 Mapeamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
O principal objectivo do mapeamento gentico a clonagem de genes, sobretudo sobretudo genes de doenas. Uma vez clonado o gene, podemos determinar a sua sequncia de DNA e estudar o produto da sua protena.
1.1. Anlise de ligaes
O mapeamento gentico baseado na ligao entre loci (locais dos genes). Se dois loci so herdados juntos, diz-se se que esto ligados. Dois loci em diferentes cromossomas no esto ligados, porque geralmente so separados devido sua disposio independente. Na metfase I da meiose, dois pares de cromatdeos irmos homlogos alinham lado a lado. Um par (o homlogo materno) vindo do ovo e outro par (o homlogo paterno) vindo do espermatozide. Subsequentemente, os dois pares de homlogos so separados em duas clulas filhas diferentes. Uma vez que eles so alinhados aleatoriamente, a escolha do homlogo que que vai para cada clula filha aleatria. Como resultado, cada clula filha contm alguns homlogos maternos e alguns paternos. Para a clula humana, h no total 8.4 milhes (223) de combinaes possveis.
Figura: Ilustrao da disposio independente. Clulas do espermatozide (1n) vindas da diviso meitica dos espermatcitos primrios (2n). Nos 23 cromossomas, alguns (rosa) ) so herdados do homlogo materno e outros (azul) ) do homlogo paterno. H no total 8.4 milhes (= 223) de combinaes possveis possveis, mas s 4 dessas so mostradas aqui.
2. Mapas fsicos
Mapeamento fsico utiliza todas as tcnicas (exs: Somatic cell hybridization e Fluorescent In Situ Hybridization (FISH)) ou informaes para determinar a posio absoluta de um gene num cromossoma. Corresponde rresponde ao mapa obtido aps a aco de uma enzima de restrio, portanto, sequncia de genes num cromossoma. O mapa fsico d-nos nos a posio e a distncia de um loci de um gene num determinado cromossoma, enquanto que o mapa gentico nos d a construo desse mesmo gene. H uma identidade entre mapa fsico e gentico no cromossoma.
| Captulo 8 Mapeamento
109
Biologia Molecular - 1 Ano - FFUP
2006/2007
Um locus (singular de loci) a posio ocupada por um gene no mapa gentico, representa uma caracterstica, pode ter diferentes sequncias, os alelos e estes tm a mesma posio posio nos cromossomas individuais. Considerando-se dois loci A e B, cada um com dois alelos (um da me outro do pai). A1 eA2 so dois alelos do locus A; B1 e B2 so dois alelos do locus B. inicialmente, A1 e B1 esto localizados no mesmo cromossoma. A2 e B2 esto localizados num cromossoma diferente.
Figura: Ilustrao da recombinao entre dois loci A e B. (a) Dois pares de cromatdeos irmos alinham durante a meiose. A1 e B1 esto localizados no mesmo cromossoma. A2 e B2 esto localizados num cromossoma diferente. (b) DNA crossover origina recombinao se o quiasma se localiza entre os dois loci. (c) DNA crossover no origina recombinao se o quiasma no se localiza entre dois loci.
O crossover do DNA pode originar a recombinao dos loci A e B. A saber, A1 e B2 (ou A2 e B1) esto localizados no mesmo cromossoma. A frequncia de recombinao depende da distncia entre dois loci e a posio de crossover (o quiasma). Quanto mais juntos estiverem, menor a probabilidade de ocorrer recombinao, porque orque a recombinao ocorre somente quando o quiasma se localiza entre dois loci. Para aplicar este princpio bsico para um mapa dum gene da doena, precisamos analisar o pedigree e estimar a frequncia de recombinao.
Unidade mapa ou centiMorgan representado representado pela sigla cM, a unidade de distncia entre dois genes ligados, equivalente a 1% de recombinantes. Esta unidade utilizada em mapeamento cromossmico feito pela anlise da percentagem de recombinantes entre dois locus gnicos quaisquer, localiz localizados num mesmo cromossoma. Por exemplo, quando na descendncia de um cruzamento em que so analisados dois locus, 15% dos indivduos so recombinantes, a distncia entre estes locus de 15 cm.
110
Captulo 8 Mapeamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
2.1. Elaborao de mapas fsicos, genticos (restrio), de cromossomas e de localizao de genes
| Captulo 8 Mapeamento
111
Biologia Molecular - 1 Ano - FFUP
2006/2007
3. Clculo da distncia entre dois genes
Frequncia de recombinao corresponde proporo de clulas haplides que representam uma recombinao entre dois locus.
A distncia entre dois genes dada pelo nmero total de recombinantes resultante do cruzamento entre um duplo heterozigoto e um duplo homozigoto recessivo.
Ex: Numa populao de 100 indivduos os fentipos obtidos foram: - 41 indivduos AB e 41 indivduos ab - 9 indivduos Ab e 9 indivduos a B A frequncia de recombinantes/crossing-over de 18%(9+9) A distncia entre o locus A e o locus B de 18 unidades mapa.
4. Clculo da posio relativa entre trs genes
Esta dada pelo nmero total de recombinantes simples entre cada dois locus + frequncia de duplos rescombinantes obtidos aps o cruzamento entre um triplo heterozigoto e um triplo homozigoto recessivo.
112
Captulo 8 Mapeamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
Ex: Numa populao de 1000 indivduos os fentipos obtidos foram: - 430 indivduos ABC e 452 indivduos abc - 45 indivduos aBC e 38 indivduos Abc - 16 indivduos abC e 17 indivduos ABc - 1 indivduo aBc e 1 indivduo AbC A distncia entre os locus A e B dada pela soma de: - recombinantes simples: Abc + aBc (38+45=83) - recombinantes duplos: aBc + AbC (1+1=2)
A e B distam de 8,5 unidades de mapa (83+2=85=85%)
A distncia entre os locus B e C dada pela soma de: - recombinantes simples: ABc + abC (17+16=33) - recombinantes duplos: aBc + AbC (1+1=2)
| Captulo 8 Mapeamento
113
Biologia Molecular - 1 Ano - FFUP
2006/2007
B e C distam de 3,5 unidades de mapa (33+2=35=3,5%)
As duas classes mais frequentes ou no recombinantes (ABC e abc) indicam que os genes esto em cis (localizam-se na mesma poro de DNA) Os duplos recombinantes (aBc e AbC) indicam que o gene do meio o B.
114
Captulo 8 Mapeamento |
Biologia Molecular 1 Ano - FFUP
2006/2007
Captulo 9 Gentica de bactrias
As bactrias possuem um nico cromossoma, circular e no ramificado. Cerca de 90% dos seus genes so de uma nica cpia, exceptuando os genes que codificam o rRNA que so de cpias mltiplas (num conjunto de sete genes).
O cromossoma bacteriano j foi sequenciado, todos os genes j foram localizados. Estes encontram-se agrupados por funes relacionadas (biossntese ou degradao), em operes com localizao e direcco diferentes no cromossoma (cerca de 260 genes em 75 operes).
O cromossoma apresenta tambm sequncias IS, sequncias de insero, j estudadas, que permitem saltos ao longo do genoma.
1. Opero
Opero um conjunto de genes nos procariontes que se encontram funcionalmente relacionados, contguos e controlados coordenadamente, sendo todos expressos num mRNA policistrnico. Assim sendo, constitudo por genes reguladores (promotor e operador) e pelos os genes estruturais.
| Captulo 9 Gentica de bactrias
115
Biologia Molecular - 1 Ano - FFUP
2006/2007
2. Utilidade dos estudos em bactrias
As bactrias so imensamente utilizadas em vrios estudos genticos devido enorme capacidade de adaptao dos seus sistemas genticos s diferentes condies ambientais em que se encontram, permitindo-nos realizar o estudo dos genes, da sua regulao e do modo como o cdigo gentico actua. tambm mais fcil a deteco e identificao de mutaes espontneas e induzidas por replica plating, que conduzem ao aparecimento de diferentes fentipos. As bactrias podem ser prototrficas (bactrias sem mutaes, que crescem num meio mnimo de glucose, gua e sais minerais, produzem os nutrientes e ATP para as actividades metablicas) ou auxotrficas (requerem a adio do nutriente que deixaram de ter capacidade de sintetizar, possuem um gene inactivado atravs de uma qualquer mutao, por exemplo conjugao, transposio, etc.). A anlise de flutuao permite verificar agentes mutagnicos. Ou seja, atravs dos resultados, podemos concluir se as bactrias j teriam sofrido mutaes, que podero implicar uma resistncia aco de antibiticos ou de fagos por exemplo.
116
Captulo 9 Gentica de bactrias |
Biologia Molecular 1 Ano - FFUP
2006/2007
3. Deteco de bactrias auxotrficas
A deteco de bactrias auxotrficas pode ser feita atravs da tcnica replica plating:
. No meio rico, todas as bactrias que sobreviveram ao agente mutagnico, cresceram. No meio mnimo, todas as bactrias cresceram excepo das que auxotrficas. Estas vo ser identificadas no meio mnimo com o suplemento de arginina (nutriente que queremos identificar na bactria auxotrfica), pelo que as bactrias que apareceram a mais so as auxotrficas.
| Captulo 9 Gentica de bactrias
117
Biologia Molecular - 1 Ano - FFUP
2006/2007
4. Enriquecimento de mutantes
Seleco directa: aplicao de condies de crescimento que favorecem o isolamento de mutante (agentes qumicos: antibiticos; fsicos U.V., biolgicos: fagos)
Seleco fsica: tamanho, mobilidade, temperatura e diferente metabolizao de nutrientes;
Contra seleco: utilizao de agentes que matam clulas em crescimento (penincilina)
O genoma do cromossoma bacteriano mantm-se praticamente inalterado, no entanto mecanismos como por exemplo: recombinao gentica, transposio, transduo, transformao, conjugao e crossing over ilegtimo, podem altera-lo.
118
Biologia Molecular 1 Ano - FFUP
2006/2007
5. Recombinao gentica
A recombinao gentica verifica-se entre duas bactrias auxotrficas e permite a formao de bactrias prototrficas.
A recombinao nem sempre depende de regies homlogas e da RecA, apesar deste ser o mecanismo mais frequente. Existem outros tipos de recombinao, que geralmente dependem de alguma enzima que reconhea sequncias de DNA (transposio).
5.1. Transposio
Resulta da capacidade que os elementos de transposio (ou transposes) tm de se movimentarem de um local do genoma para outro. Os transposes tm nas suas extremidades, sequncias invertidas repetitivas e quando reconhecem o seu target site, saltam e a podem promover a duplicao de um segmento de bases do DNA alvo. Por outro lado, podem sair apenas com a sequncia de insero ou levar qualquer tipo de DNA adjacente.
119
Biologia Molecular - 1 Ano - FFUP
2006/2007
H dois tipos de transposio: Transposio simples: os transposes saltam de uma molcula para a outra, em que a inicial degradada; Transposio replicativa: reconhecem a sequncia alvo, e formam uma estrutura co-integrada em que ambos os DNAs ficam com um transposo.
5.2. Transformao
A transformao um mecanismo de transferncia gentica entre bactrias no qual o DNA dador DNA livre no ambiente que circunda a bactria receptora (ex: Plasmdeos). Ou seja, a bactria absorve DNA podendo ou no integr-lo no seu genoma, com consequente alterao do gentipo e fentipo.
Para introduzirmos o DNA numa bactria, temos de trat-la de modo a torn-la competente, ou seja, susceptvel de receber o plasmdeo. O tratamento feito com cloreto de clcio, ligeiramente hipotnica, a 0C (para promover a inibio das nucleases). Ao adicionarmos DNA plasmdico s clulas competentes, h formao de precipitados de Ca2+-DNA. Por fim, as clulas competentes so incubadas com o vector, neste perodo realizado o tratamento com choque trmico. Agora a bactria tem a capacidade de integrar o DNA plasmdico.
120
Biologia Molecular 1 Ano - FFUP
2006/2007
Quando integrados no genoma, os plasmdeos tm a designao de epissoma.
5.3. Transduo
Outro processo pelo qual podemos alterar o genoma bacteriano. um fenmeno raro, mediado por um fago de transduo, que transfere um gene bacteriano de uma bactria para outra. O fago infecta a bacctria, introduzindo o seu material gentico no genoma da bactria, pode ocorrer o ciclo ltico ou o ciclo lisognico. Existem portanto dois mecanismos de transduo: generalizada, em que qualquer gene pode ser transmitido, e restrita, que se limita a alguns genes especficos. Na transduo generalizada, nucleases virais quebram o genoma do hospedeiro. Alguns fragmentos so depois encapsulados, formando-se uma partcula transductora. Quando esta infecta outra bactria, o DNA pode integrar-se no seu genoma.(ciclo ltico) A transduo restrita est ligada ao ciclo lisognico dos fagos. Neste caso, o genoma viral insere-se num local especfico do genoma bacteriano e transmitido descendncia. Quando o ciclo ltico desencadeado, o genoma viral excisado por protenas virais e, por vezes, fica agarrado a algum DNA bacteriano e encapsulado, tal como na transduo generalizada.
121
Biologia Molecular - 1 Ano - FFUP
2006/2007
5.4. Conjugao
Processo unidireccional de transferncias de informao gentica entre bactrias, que ocorre de uma bactria dadora, F+ (masculina que possui o factor F ou factor de fertilidade, que funciona como plasmdeo) para uma bactria receptora (feminina que no possui o factor F) pelo que designada F-.
Caractersticas do factor F: Cadeia dupla de Dna Cerca de 60 genes Tem sequencias Is Local de origem de replicao (oriV) Regio de transferncia (OriT)
Esta regio (Ori T) sintetiza a fmbria sexual (pili F), protena especifica que permite a transferncia do factor F para as formas F-, pois pe em contacto a clula F+ com a clula F-. Esta regio contem cerca de 33 genes, designados genes tra: gene traA (codifica para a pilina, protena da pili F), o gene traD (faz parte do canal por onde passa o Dna), e os genes traS e traT (codificam para protenas de superfcie de excluso que impedem o contacto entre de duas clulas F+).
6. Transferncia do factor de fertilidade
A conjugao (transferncia do factor F) feita atravs da fuso entre as paredes de duas bactrias, mas que numa primeira fase contactam atravs da pili F. De seguida d-se a clivagem de uma das cadeias do factor F, e a replicao comea (por crculos rolantes). Uma das cadeias introduzida na bactria feminina, sendo conduzida por uma pequena fraco de DNA. Quando a cadeia do Factor f fica completa, as clulas separam-se, e originam duas clulas masculinas. 122
|
Biologia Molecular 1 Ano - FFUP
2006/2007
Quando todas as clulas so masculinas, o contacto entre duas clulas masculinas impedido pelos genes traS e traT.
O factor F, tambm se pode integrar no cromossoma da bactria, em que a clula resultante passa a ter a designao de Hfr. O local de incio da transferncia varia conforme as estirpes. Quando integrado no genoma, a transferncia do factor F mais difcil. A integrao do factor F feita aleatria ao longo do genoma e faz-se num nico crossing-over originando um Hfr. As clulas Hfr permitem a identificao de genes no cromossoma bacteriano, assim como demonstrar que os genes nas bactrias existem numa nica cpia.
|
123
Biologia Molecular - 1 Ano - FFUP
2006/2007
As regies onde ocorre esta recombinao so sequncias de insero (IS), que consistem em sequncias de DNA homlogas no plasmdeo conjugativo e no cromossoma bacteriano. Quando a conjugao ocorre entre uma clula Hfr e uma clula F-, inicia-se a transferncia de uma das cadeias do cromossoma Hfr para a clula receptora. Nesta verifica-se a recombinao gentica com troca de DNA (crossing over) nos pontos de homologia. transferida apenas uma parte do cromossoma, pelo que a clula receptora permanece F-. Para alm da pili sexual, o contacto entre as bactrias pode ser conseguido atravs de aglutininas (promovem a formao de agregados celulares) ou de feromonas (substancias qumicas produzidas por um indivduo que alteram os outros individuos da mesma espcie)
Mas e o que so plasmdeos? Os plasmdeos so elementos genticos autnomos, distintos do genoma principal bacteriano. A sua replicao muitas vezes independente da replicao cromossomica da bactria. Podem existir vrios plasmdeos numa bactria, quer fora do cromossoma, quer integrado (epissoma). Podem codificar para funes no essenciais, mas tambm podem conferir caractersticas nicas bactria, nomeadamente resistncia a antibiticos. Algumas funes atribudas aos plasmdeos so:
124
Biologia Molecular 1 Ano - FFUP
2006/2007
7. Recombinao em E. coli
125
Biologia Molecular - 1 Ano - FFUP
2006/2007
Captulo 10 Gentica de bacterifagos
Bacterifagos so vrus que parasitam e matam bactrias. Estes replicam-se no interior das clulas bacterianas e so libertados para o exterior quando a clula lisa. Os fagos so muito importantes na Biologia Molecular sendo utilizados como vectores de clonagem para inserir DNA nas bactrias e constituem uma importante ferramenta no controlo de infeces bacterianas pelo facto de serem incuos, ou seja, terem elevada especificidade contra o microrganismo hospedeiro e serem capazes de infectar bactrias que desenvolveram resistncia a antibiticos, sendo por isso uma alternativa a estes. Os fagos vo permitir estudos genticos da bactria. Dois fagos com gentipos diferentes podem cruzar-se para avaliao da recombinao e do mapa gentico. Por exemplo, se se infectar uma bactria com dois fagos mutantes e se houver recombinao isso verificado pela observao de placas fgicas, pois cada gene tem um fentipo diferente.
Forma:
Filamentosa - DNA fica embebido ao longo de todo o fago. Material genmico envolvido por protenas. Os capsdios so cilindros ocos, com estrutura helicoidal. Um exemplo de um vrus filamentoso o vrus do mosaico do tabaco (TMV)
Esfrica ou icosadrica uma estrutura icosadrica devido s 20 faces triangulares. Estrutura com uma cabea, pescoo, colar, revestimento e placa basal com fibras caudais. A cabea contm material genmico (DNA).
1. Infeco por bacterifago
A infeco feita por bacterifagos engloba vrias etapas como a coliso, contraco do tubo, injeco do material gentico e expresso de genes. Na primeira etapa, tambm chamada por adsoro, ocorre a participao de receptores especficos da superfcie da clula hospedeira e das macromolculas do fago. Compreende duas fases, a primeira uma adsoro preliminar por ligaes inicas, facilmente reversveis por alterao do pH ou concentrao salina do meio, e uma segunda fase em que ocorre uma ligao mais firme e irreversvel entre o fago e os 126 Captulo 10 Gentica de bacterifagos |
Biologia Molecular 1 Ano - FFUP
2006/2007
receptores. Virtualmente, qualquer componente existente na superfcie usado pelo fago como stio especfico de adeso, nem que tenha de haver, em certos casos, um rearranjo fsico da partcula fgica. No caso especfico da interaco do fago T4 com a superfcie de clulas de E.coli, primeiro, h uma interaco relativamente fraca, entre as pontas das fibras caudais e os resduos lipopolissacridos existentes na superfcie da membrana externa da clula. A maltose ser importante pois ir aumentar a expresso da protena Ompc (porina proteica) levando assim a uma maior interaco entre o fago e a superfcie da bactria. Esta interaco desencadeia uma segunda interaco mais forte e irreversvel. Neste passo, os pinos da cauda interactuam com estruturas existentes na membrana externa da clula requerendo uma alterao na conformao das fibras da cauda. O magnsio ser tambm um elemento importante pois facilitar, igualmente, a adeso do fago.
Aps a coliso ocorre a compresso da cauda contrctil e injeco do material gentico para o interior da clula hospedeira atravs da penetrao da parede celular pelo tubo da cauda do fago. Fora da clula fica a cpsula proteica vazia. O material gentico do fago introduzido pode ser: DNA de cadeia dupla (), DNA de cadeia simples (M13), RNA de cadeia simples (MS2) ou RNA de cadeia dupla (retrovirus). No caso de fagos com cauda, como o caso do fago T4, apenas o DNA fgico e certas protenas acessrias entram na clula. No entanto existem outros casos (fagos sem cauda), em que a partcula fgica entra inteiramente na clula passando para o citoplasma. Depois da insero do material genmico podero ocorrer vrias respostas, como a resposta ltica ou resposta lisognica. Mais tarde ocorrer a expresso dos genes iniciais (preparao da clula para a produo de macromolculas), genes mdios (asseguram o processo de replicao do fago e sintetizam vria protenas necessrias para a posterior replicao do cido nucleico) e dos genes tardios (sintetizam protenas necessrias para formar a protena vrica completa). Todos eles sero responsveis pela formao de novas partculas fgicas. A lisosima, uma das protenas sintetizadas pelos genes ser, entre outras, responsvel pela lise celular das bactrias fazendo com que ocorra libertao das partculas virais e outros componentes celulares para o meio extracelular. A lise celular a etapa final do processo de infeco, no entanto no como a todos os processos de infeco fgica.
| Captulo 10 Gentica de bacterifagos
127
Biologia Molecular - 1 Ano - FFUP
2006/2007
2. Ciclo ltico
Existem vrios tipos de ciclos fgicos. No caso do ciclo ltico, o vrus transforma a clula em fbrica de fagos (j que este no capaz de se reproduzir sozinho), fazendo depois com que ocorra a lise desta e os fagos sejam liberados podendo infectar, assim, outras clulas bacterianas. A estes tipos de fagos que infectam a bactria e iniciam imediatamente o seu processo de reproduo provocando depois sua lise chama-se fagos virulentos.
O ciclo ltico pode ser divido em vrias partes: - Infeco o fago ataca a bactria e o seu material gentico injectado na bactria. - Infeco pr-inicial o fago apodera-se da maquinaria de sntese proteica da clula hospedeira e prepara a clula para a produo de macromolculas. Outras funes pr-iniciais asseguram que apenas o cido nucleico do fago replicado e que nenhum outro fago estranho interfira no processo. Um dos aspectos da replicao fgica mais fascinantes e menos compreendida o facto de uma pequena poro de cido nucleico, aps a entrada na clula, conseguir ser responsvel pelo metabolismo e capacidade sinttica da clula para a converso de novos fagos. - Infeco inicial ocorre a sntese de enzimas e substratos necessrios para a posterior replicao do cido nucleico. Inicia-se a sntese/replicao de DNA. - Infeco tardia so sintetizadas protenas estruturais virais e enzimas necessrias para a morfognese da cpsula e, tambm, para o empacotamento do cido nucleico. Ou seja, so sintetizadas todas as partes que constituem o fago, como a cabea e a cauda, e o DNA introduzido nas cabeas. Neste processo ocorre, ento a montagem das partculas que constituem o fago. No final deste processo obtm-se vrios viries, fase mais madura do fago. - Lise das bactrias fase final do ciclo ltico e caracteriza-se pela ruptura da clula bacteriana, e os novos bacterifagos so libertados para o meio extracelular, podendo infectar outras bactrias e iniciar outro ciclo. A lisozima uma das protenas que causa o rompimento da parede celular.
128
Captulo 10 Gentica de bacterifagos |
Biologia Molecular 1 Ano - FFUP
2006/2007
3. Formao de virio
A partcula vrica completa/madura conhecida pela denominao virio. O virio composto de cido nucleico, sede de sua infectividade, circundado por uma capa proteica, chamada capsdio, responsvel pela especificidade viral. Como j vimos o vrus pode ter DNA ou RNA, mas nunca so encontrado os dois juntos no mesmo virio, o que estabelece um contraste com todas as formas celulares de vida, as quais, sem excepo, contm os dois tipos de cidos nuclicos. Os viries abandonam a fase extracelular quando se introduzem numa nova clula hospedeira, por um processo chamado infeco. Durante o processo de penetrao na clula ou j no interior da clula infectada, os viries perdem o revestimento proteico e o genoma viral codifica a sntese de novas partculas virais completas, utilizando e dependendo da maquinaria metablica da clula infectada ocorrendo, deste modo multiplicao viral. semelhana do que se passa com os plasmdeos, se uma clula infectada se reproduz, as clulas filhas podem tambm conter o genoma viral. Contudo, o processo de reproduo dos vrus (formao de novos viries) implica, em muitos casos a destruio da clula infectada, situao que se encontra relacionada com a capacidade patognica de muitos vrus. Dado que a replicao viral depende em absoluto da maquinaria e funes metablicas da clula hospedeira, necessria uma grande complementaridade entre a estrutura da partcula viral e a da clula infectada, para que ocorra a replicao viral. Por esta razo, embora exista uma grande variedade de vrus e de clulas susceptveis de serem infectadas, a relao virio/clula hospedeira altamente especfica. Na maior parte dos casos, um determinado vrus apenas pode infectar um tipo muito especfico de espcies celulares. Esta especificidade comea logo na necessidade da existncia de estruturas complementares do virio na superfcie das clulas, que sirvam de receptores especficos para a adeso e penetrao intracelulares. Existe uma sequncia definida para a formao do fago maduro (virio). Cada um dos seus constituintes sintetizado separadamente (cabea, pescoo, colar, core, revestimento, cauda, placa basal e fibras caudais) e depois estes unem-se, h uma montagem destes, formando-se assim o virio. Este um modelo morfogentico ou self assembly.
4. Ciclo lisognico
Como j vimos, alguns fagos so virulentos, significando que uma vez que a clula tenha sido invadida, eles imediatamente iniciam seu processo de reproduo, e em pouco tempo "lisam" a clula. Estes seguem portanto apenas o ciclo ltico. Porm, h situaes em que os vrus, designados por fagos temperados, entram na clula num estado relativamente inofensivo, no dando lugar formao de viries infecciosos durante alguns ciclos de multiplicao celular. Diz-se ento que estes actuam na forma de provirus ou profagos, pois integram o seu DNA no cromossoma da bactria hospedeira. Estas bactrias so chamadas de lisognicas, pois o genoma viral vai ser copiado a cada diviso celular junto com o DNA da bactria, transmitindo assim o DNA viral, sem se manifestar. No entanto, em determinadas condies, naturais ou artificias (como radiaes ultravioleta, raios X ou certos agentes qumicos), o DNA do fago separa-se do
| Captulo 10 Gentica de bacterifagos
129
Biologia Molecular - 1 Ano - FFUP
2006/2007
DNA bacteriano e inicia-se o ciclo ltico, dando assim lugar a formao de viries, ou seja, os fagos tornamse activos iniciando o seu ciclo reprodutivo, resultando assim na lise de clula hospedeira. Ao ciclo seguido pelo fago temperado chama-se ento ciclo lisognico. Quando no cromossoma da bactria est integrado o DNA do fago, esta fica imune infeco por parte de novas partculas vricas. Um exemplo de fago temperado que segue este tipo de ciclo o vrus da herpes labial, pois pode viver vrios dias em via lisognica e a qualquer momento devido a vrios factores pode entrar em via ltica.
5. Infeco do fago
O fago lambda () um fago temperado. O seu genoma tem 48,5kb, e sequenciado. Este codifica para 46 genes. Nas extremidades do genoma linear existem duas regies em cadeia simples (12pb) que so complementares e actuam como extremidades coesivas (cos). Na extremidade esquerda esto localizados os genes que codificam as protenas da cabea e cauda, e os genes que codificam protenas envolvidas no ciclo ltico mapeiam na extremidade direita, podendo assim a regio central do genoma ser removida ou substituda por DNA exgeno. Aps infeco estas extremidades complementares permitem ao DNA adquirir o estado circular, devido ao emparelhamento das bases com ajuda da DNA ligase. Como acontece com outros fagos temperados, a maioria das infeces com fago resultam em ciclo ltico e lise. 130 Captulo 10 Gentica de bacterifagos |
Biologia Molecular 1 Ano - FFUP
2006/2007
Depois do fago seguir a via ltica, a sua replicao dividida em duas fases: - Fase inicial a replicao bidireccional. Esta primeira fase estende-se de 5 a 15 minutos aps a infeco. Depois comea uma nova fase. - Fase tardia a replicao feita em crculos rolantes. Aps a replicao tardia o fago encapsulado. O DNA que ser encapsulado contem concatmeros, que so conjuntos de genomas do fago uns a seguir aos outros que esto separados pelas extremidades cos. Estas extremidades servem para reconhecer e cortar as extremidades de lambda. As extremidades so cortadas por uma protena. O DNA do fago vai entrando na cabea at que esta fique cheia, quando isto acontece a cauda adicionada a cabea e tem-se o fago maduro que aps a lise estar pronto para infectar outras bactrias.
6. Recombinao entre fagos
A recombinao gentica um processo de manipulao artificial de genes que conduz ao aparecimento de novas caractersticas nos microrganismos. Nos fagos a recombinao acontece por via dos processos de transformao, transduco e conjugao. Se se infectar uma bactria com dois fagos possvel ver que ocorreu recombinao entre estes atravs da visualizao de placas fgicas, pois cada uma das bactrias infectadas ter um fentipo diferente. Assim, se se cruzar dois fagos, de gentipos h- r+ x h+r-, quatro fentipos podem ser facilmente diferenciados nas placas, dois fentipos parentais e dois recombinantes. O processo de recombinao vai permitir ento que partes do DNA de dois fagos mutantes que infectem uma bactria, se possam combinar formando uma nova molcula de DNA duplamente mutante ou uma molcula de DNA que no contenha o DNA de nenhum dos fagos (wild type). Ento o que ocorre o seguinte, dois fagos mutantes obtidos de duas culturas diferentes so introduzidos num meio com E.coli do tipo B. O cruzamento ocorre quando o DNA de cada um dos fagos mutantes injectado num nico bacilo. Aps ocorrer a replicao, o DNA ou de um tipo ou de outro, mas ocasionalmente a recombinao ir produzir ou um DNA duplamente mutante ou um DNA que no contem nenhuma das mutaes.
| Captulo 10 Gentica de bacterifagos
131
Biologia Molecular - 1 Ano - FFUP
2006/2007
7. Recombinao entre o fago e a bactria
Os fagos , so de grande utilidade na Tecnologia do DNA recombinante. Como j vimos, o fago lambda possui genes essenciais que so indispensveis reproduo, e tambm genes no essenciais, cuja presena dispensvel a multiplicao viral. Os genes essenciais produzem as protenas da cpsula e as enzimas que actuam na duplicao do DNA viral. Esses genes esto localizados nas extremidades do cromossoma do vrus. Entretanto, os genes no essenciais esto relacionados aos processos de recombinao entre molculas de DNA, e localizam-se na regio mediana do cromossoma viral, sem interferir na reproduo do vrus, sendo portanto dispensveis. A regio mediana do cromossoma, onde se localizam os genes dispensveis, pode ento ser retirada e substituda por um pedao de DNA de outro organismo. Sendo assim, quando o cromossoma viral se multiplica, o DNA estranho incorporado a ele tambm ser multiplicado. A recombinao entre o fago e o cromossoma de E.coli ocorre atravs de um local de ligao especfico do DNA circular e uma regio especfica entre os genes gal e bio do cromossoma da bactria.
8. Formao de um fago de transduo
A transduo especializada a transduo em que apenas certos genes dadores podem ser transferidos para a molcula receptora. Diferentes fagos podem transferir diferentes genes mas um fago individual apenas pode transferir alguns deles. Assim, esta transduo medida por fagos temperados ou lisognicos e os genes que vo ser transferidos vo depender de onde o pr-fago insere o gene na sequncia do cromossoma. Assim, inicialmente ocorre a produo de uma bactria lisognica atravs de crossing-over numa determinada regio especializada. A cultura das bactrias pode produzir o fago ou, em casos mais raros, uma partcula anormal, dgal, que vai actuar como partcula transdutora. Ao longo do mecanismo, podem ser produzidos transductantes de gal+ atravs de vrias incorporaes de dgal e de um fago helper ou, mais raramente, atravs de trocas no interior do gene gal.
132
Captulo 10 Gentica de bacterifagos |
Biologia Molecular 1 Ano - FFUP
2006/2007
Captulo 11 Vrus
As bactrias, os fungos e os parasitas eucariticos so clulsa propriamente ditas. Mesmo quando so parasitas obrigatrios, eles usam a sua prpria maquinaria de replicao de DNA, de transcrio e de traduo, provendo eles prprios os seus recursos de energia metablica. Os vrus, em contraste, so os perfeitos passeadores, carregando pouca informao sob a forma de cidos nucleicos. A informao totalmente replicada, empacotada e preservada pelas clulas hospedeiras. Basicamente, o genoma vrico contm molculas de DNA de cadeia simples ou dupla (3 a 200 Kb) e RNA de cadeia simples ou dupla (7 a 20 Kb). Geralmente, podem ter forma circular ou linear, sendo que no caso doo DNA, este no existe na forma segmentada, excepto nas plantas, e no caso do RNA, no existe na forma circular, excepto no caso dos procariotas. Quanto origem vrica, os mais comuns derivam de fragmentos de DNA nucleide, de cromossomas derivados de clulas vivas e de cpias de RNAs destes fragmentos anteriores. Relativamente diversidade vrica existente, pode afirmar-se que existe uma grande variedade, tendo em conta a forma, a estrutura interna e a funo que cada vrus apresenta.
1. Estrutura
Os vrus podem apresentar diferentes estruturas, de acordo com o organismo hospedeiro e com a sua origem. Fora de uma clula-hospedeira, os vrus no podem transportar as funes que desempenham, nem a sua reproduo consequente. No podem sintetizar protenas porque no contm ribossomas, necessitando assim dos ribossomas das clulas-hospedeiras para traduzir o mRNA viral para protenas virais. Geralmente, no podem gerar nem depositar energia na forma de ATP mastm de expr a sua energia e todos os restantes processos metablicos a partir da clula-hospedeira. Os vrus so geralmente classificados de acordo com os organismos que infectam, isto , animais, plantas ou bactrias. Desde que os vrus no conseguem penetrar atravs das paredes celulares, tm a capacidade de transmitir a sua informao gentica atravs de insectos ou outros organismos que se alimentam de plantas.
| Captulo 11 Vrus
133
Biologia Molecular - 1 Ano - FFUP
2006/2007
Os vrus so classificados em famlias, relativamente s suas estruturas, tendo em conta os seguintes factores: Tamanho e tipo de cido nucleico que integram Tamanho e forma da cpside Existncia ou ausncia de um envelope lipdico em torno da nucleocpside Relativamente forma, existem duas principais categorias: esfricas/icosadricas ou filamentosas. No caso dos esfricos, o principal exemplo do vrus HIV, que apresenta a forma de um polgono com 20 lados (icosaedro) e contm um envelope proteico (cpside), RNA como cido nucleico e enzimas. No que diz respeito forma de filamento, baseia-se no conceito da estrutura dos bacterifagos. Estes vrus apresentam uma cauda, que est anexada superfcie bacteriana. Esta cauda contacta com a cabea do vrus (onde se encontra o DNA) atravs de um prolongamento de protenas internas, e na parte inferior apresenta pequenas fibras.
2. Diferentes coberturas dos vrus
Em virologia, o genoma de um vrus de RNA pode ser (+) ou (-) consoante o sentido da polaridade da molcula de cido nucleico. No caso dos vrus de RNA (+), uma sequncia viral de RNA pode ser directamente transcrita para a protena viral desejada. Consequentemente, nestes vrus, o RNA viral muitas considerado como um mRNA e pode ser imediatamente traduzido para a clula-hospedeira. Os principais vrus desta cobertura so o togavrus, o retrovrus e o coronavrus. Contrariamente, os vrus de RNA (-) so complementares ao mRNA viral e, como consequncia, tm de ser convertidos para RNA (+) pela aco de uma RNA polimerase antes da traduo. Estes vrus apresentam no RNA (-) uma sequncia nucleotdica complementar ao mRNA que codifica, fazendo com que tal como o DNA, este RNA no possa ser traduzido directamente para a protena, tendo inicialmente de ser transcrito para um RNA (+) que actua como mRNA possvel de ser traduzido. Os principais vrus desta categoria so o paramixovirus, o rabdovirus e o ortomixovirus. 134 Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
Por fim, no caso dos vrus com dsDNA, os principais so o vrus do herpes e o poxvirus.
3. Efeitos de vrus na causa de doenas
As clulas do corpo humano, quando se encontram infectadas por vrus, produzem interfero. Os interferes so protenas naturais produzidas em resposta ao incio da actuao dos vrus, das bactrias, dos parasitas e das clulas tumorais e fazem parte de uma grande classe de glicoprotenas, as citoquinas. As principais classes que interferem nos vrus so os interferes nos linfcitos, os interferes nos fibroblastos e os interferes nos mitognios. Tendo em conta que podem ser usados na terapia gnica, o principal interveniente neste processo o gene da CTFR que quando se encontra recombinado com o adenovrus utilizado no tratamento da fibrose cstica.
4. Entrada e sada de vrus
Os vrus quando em contacto com determinadas clulas, atravessam um conjunto de mecanismos de entrada para desenvolverem o processo de infeco. Inicialmente, a partcula vrica interage com a clula-hospedeira, que apresenta receptores especficos, maioritariamente glicoprotenas. Seguidamente, para o virio ser incorporado na clula, tm de existir mecanismos de fuso membranar e de endocitose, sendo que nos de fuso membranar, produz-se uma protena fusognica (protena F) e nos de endocitose, formam-se vesculas revestidas por clatrina, endossomas e coated pits. Depois da incorporao para o interior da clula, a vescula vrica encapsulada funde-se com um endossoma e origina a libertao de uma cpside individual. Por sua vez, este elemento dissocia-se, originando uma sequncia de RNA que sofre simultaneamente replicao e transcrio originando mais
| Captulo 11 Vrus
135
Biologia Molecular - 1 Ano - FFUP
2006/2007
uma cadeia de RNA igual anterior e uma de mRNA. Este mRNA vai voltar a origina uma ncleocpside com RNA viral que vai interagir com protenas transmembranares vricas. Daqui, resulta a formao do envelope viral no processo de budding, fazendo com que a sua constituio varie em funo do local da clula onde se forma.
4.1. Formao do envelope viral
O capsdeo que envolve o genoma viral constitudo por uma ou vrias protenas, organizadas em camadas e padres regularmente repetidos. Em vrus envelopados, o capsdeo , por sua vez, envolvido numa membrana de bicamada lipdica que adquirida durante o processo de budding a partir da membrana plasmtica da clula-hospedeira. Os vrus no envelopados geralmente saem de uma clula infectada por meio de lise da mesma, ao passo que os vrus envelopados deixam a clula por budding, sem o rompimento da membrana plasmtica e, consequentemente, sem provocar a morte da mesma. Estes vrus podem, por vezes, causar infeces crnicas, e alguns deles podem auxiliar na transformao de uma clula infectada numa clula cancerosa.
136
Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
5. Classificao dos vrus
A principal classificao dos vrus feita com base na composio dos genomas e na via pela qual se forma o seu mRNA. Existem duas grandes divises dos vrus, que separam os vrus com DNA dos vrus com RNA. O mRNA viral designado como sendo uma cadeia extra, que sintetizada a partir de uma cadeia negativa de DNA ou RNA.
5.1. Classes de vrus
Existem sete classes principais de vrus, denominadas de acordo com a numerao romana: Classe I DNA de cadeia dupla com protenas ligadas covalentemente nas extremidades (Adenovrus) DNA de cadeia dupla circular (SV40 SimianVirus 40) DNA de cadeia dupla (Herpes e T4) DNA de cadeia dupla e extremidades seladas (Poxvirus) Classe II DNA de cadeia simples (Parvovirus) DNA de cadeia simples circular (M13 e 174) Classe III RNA de cadeia dupla (Reovirus) Classe IV IVa RNA de cadeia (+) que origina um mRNA (Poliovirus) IVb RNA de cadeia (+) que origina dois mRNAs (Sindbis virus) Classe V Va RNA de cadeia (-) no segmentada (VSV Vesicular stomatitis virus) Vb RNA de cadeia (-) segmentada (Influenza) Classe VI RNA de cadeia (+) com intermedirio de DNA (Retrovirus)
| Captulo 11 Vrus
137
Biologia Molecular - 1 Ano - FFUP
2006/2007
Classe VII DNA com intermedirio de RNA (Hepatitis DNA virus)
5.2. Tamanho dos vrus
138
Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
6. Estratgias de replicao
A replicao viral ocorre, geralmente, no ncleo da clula-hospedeira, exceptuando no caso do poxvirus (DNA) e do poliovirus (RNA) que se replicam no citoplasma. As trs principais enzimas que intervm neste processo de replicao so: RNA replicase existe nas protenas da cpside e sintetiza RNA(-) a partir de RNA (+) Transcriptase sintetiza mRNA a partir da RNA (-) Transcriptase reversa sintetiza DNA de cadeia dupla a partr de RNA
6.1. Estratgias de replicao consoante o tipo de vrus
Vrus de DNA de cadeia dupla Usam a RNA polimerase celular para a sntese do mRNA, excepto os poxvirus; Codificam para protenas da cpside e protenas necesssrias para a replicao do seu genoma. Vrus de DNA de cadeia simples (+) ou (-) Codificam para protenas da cpside; Usam sistemas de replicao e de transcrio da clula-hospedeira. Vrus de RNA (+) O RNA (+) funciona imediatamente aps a infeco com mRNA; O RNA (+) tem cauda poly-A a 3 e modificaes a 5 semelhantes s observadas no mRNA dos eucariotas; Codificam para enzimas que podem replicar o RNA atravs de intermedirios da cadeia (-). Vrus de RNA (-) O RNA (-) serve como molde na sntese de mRNA por aco da transcriptase vrica do vrus, logo aps a infeco. Vrus de RNA de cadeia dupla Codificam para enzimas que se encontram no virio, capazes de sintetizar mRNA a partir de um tipo de molde. Retrovrus A cadeia de RNA (+) serve primeiro de molde para a sntese de DNA de cadeia dupla atravs da aco da transcriptase reversa; Este DNA integra-se no genoma da clula-hospedeira e serve como molde para a transcrio do mRNA.
| Captulo 11 Vrus
139
Biologia Molecular - 1 Ano - FFUP
2006/2007
Vrus da hepatite B Possui genomas com uma cadeia de DNA (-) completa e uma cadeia de DNA (+) incompleta; Aps a infeco, a DNA poimerase completa a cadeia (+), forma-se uma cadeia de DNA dupla e ocorre a transcrio do mRNA. RNAs vricos Clivagem proteoltica sntse de protenas e posterior digesto com protease em mltiplas protenas; Genomas segmentados vrios genomas de RNA em que cada uma das molculas codifica para uma ou mais proteinas; Nested mRNAs mRNAs com extremidades 3 iguais e 5 diferentes, que codificam para protenas especficas; Sntese sequencial sntese de uma unidade longa de RNA (+) que depois separada em mRNAs para cada protena, por clivagem ou terminao. DNAs vricos Codificam para mRNAs monocistrnicos; Promotores de transcrio para todos os genes; Requerem a formao de um primer; Splicing alternativo (mRNAs sempre com a extremidade 5).
6.2. Replicao na classe I
Poxvirus A replicao ocorre no citoplasma, sendo o nico vrus que codifica a sua prpria RNA polimerase. Geralmnte, composto por dsDNA linear com 130-375 Kb, covalentemente selado nas extremidades, apresentando estruturas em hair pin em cada extremidade e com sequncias repetidas (10 Kb) associadas replicao. Codifica para 150 a 300 protenas, tendo regies codificantes bastante espaadas, mas sem intres e nas duas cadeias do genoma.
140
Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
A replicao do poxvirus inclui vrias fases. A primeira fase que o vrus atravessa a ligao a um receptor na superfcie da clula-hospedeira e a entrada do vrus para o interior da clula, onde vai perder a sua cpsula. Esta pode dividir-se em dois processos: inicialmente, a membrana externa removida medida que a partcula vrica entra para a clula, e de seguida, o envelope viral perdido para o citoplama. De seguida, os genes do poxvirus so expressos, tambm em duas fases: os genes novos so expressos em primeiro lugar, codificando uma protena noestrutural, incluindo as protenas necessrias para a replicao do genoma viral, sendo por isso expressas antes do mecanismo total de replicao. Os genes mais tardios so expressos depois da replicao do genoma e codificam protenas estruturais para a formao da partcula vrica. O reconhecimento do vrus ocorre sempre no citosqueleto da clula, conduzindo iniciao de um processo de replicao bastante complexo. Inicialmente, depois da remoo do core referido anteriormente, ocorre a sntese de mRNAs iniciais, juntamente com procesos de proliferao celular e supresso imune local. De seguida, tem local a sntese de DNA para empacotamento e molde para expresso de genes intermedirios que depois especificam factores de expresso gnica tardia. Por fim, verifica-se a transcrio e a traduo de protenas estruturais vricas, partculas que se renem no complexo de Golgi e a libertao do virio nas clulas lisadas ou a infeco de clulas adjacentes. SV40 A sua expresso e a replicao do seu genoma circular feita no ncleo, atravs da utilizao de polimerases celulares. A constituio do seu genoma apresenta, basicamente, trs principais regies: inicial, tardia e de regulao. Na regio inicial, existem protenas T (20 e 90 kDa), geralmente requeridas para o incio da replicao. Na regio tardia, existem sequncias que codificam para as protenas da cpside (VP1, VP2 e VP3) que se formam por splicing alternativo de RNAs sobrepostos. Por fim, na regio de regulao existe um local de origem de replicao, promotores equivalentes TATA box, e reguladores de expresso.
| Captulo 11 Vrus
141
Biologia Molecular - 1 Ano - FFUP
2006/2007
Adenovirus A sua replicao feita no ncleo, atravs do splicing alternativo, genes sobrepostos, e duas cadeias de DNA para maximizar a utilizao do seu genoma. Aqui, a variedade obtida atravs da escolha do local de poliadenilao e do local de splicing da extremidade 3.
6.3. Replicao na classe II
Parvovirus Os parvovirus podem possuir cadeias (+) ou (-), mas geralmente encontram-se em viries separados. Apresentam um genoma muito pequeno (4500 a 5000) e codificam para as protenas da cpside por traduo de mRNAs derivados do splicing alternativo, multiplicando-se na presena de vrus mais complexos, como o adenovirus, ou de clulas com multiplicao rpida (clulas sanguneas e tumorais). So definidos por serem vrus muito facilmente transmissveis e responsveis por infeces do tracto respiratrio inferior. Fagos M13 e 174 Estes fagos apresentam DNA de cadeia simples circular, tendo mecanismos de replicao que decorrem atravs de vrias formas replicativas.
6.4. Replicao na classe III
Reovirus Os reovirus apresentam RNA de cadeia dupla que contm 10 segmentos adjacentes. Normalmente, codificam para a transcriptase e para a polimerase, permitindo a obteno de mRNA a partir da cadeia (-). Contm um pre-core que uma estrutura que contm as cadeias de RNA (+) e que inclui a sntese da cadeia dupla de RNA atravs da aco da polimerase.
142
Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
6.5. Replicao na classe IV
Poliovirus Os poliovirus contm um genoma de RNA (+) que funciona como mRNA na fase inicial da infeco. Estes vrus no modificam os mRNAs celulares, mas afectam a traduo por bloqueio na fase de iniciao da sntese proteica, fazendo com que a replicao seja iniciada atravs a traduo do poli-RNA (+) numa poliprotena precursora designada por NCVP (240 kDA). Esta protena clivada nos locais nascent cleavages e forma as protenas vricas VPO (VP4 e VP2), VP3 e VP1 que vo formar a estrutura pr-vrica. A sua regio carboxlica contm as protenas necessrias para a replicao: VPg associada formao do primer Replicase RNA polimerase-RNA-dependente Proteases clivam resduos de glutamina-glicina da poliprotena O poliovirus apresenta uma patognese bastante caracterstica, baseada nos seguintes factos: Entrada pela boca Replicao na faringe, tracto gastrointestinal e sistema nervoso Atinge vasos linfticos, fibras nervosas e sistema nervoso central Destruio dos neurnios motores Paralisias e asfixia Relativamente ao processo de replicao, feito por intermedirios replicativos, em que as cadeias (-) servem de molde para a sntese de novas cadeias (+). Por sua vez, estas novas cadeias servem de molde para a sntese de novas cadeias (-), actuam como mRNA para a sntese de protenas e pertencem aos genomas dos novos viries. Togavirus O togavirus apresenta um genoma dicistrnico, com um cistro a 5 que codifica para a protena vrica e um cistro a 3 que codifica para o precursor das protenas vricas. Geralmente, provoca encefalites vricas equinas, sendo que no ser humano responsvel pela rubola.
Coronavirus O coronavirus usa uma estratgia diferente de replicao relativamente aos outros vrus anteriores, apresentando nested mRNAs, que podem ser traduzidos para formar a protena, apesar de existirem mRNAs com extremidades 3 todas iguais e extremidades 5 todas diferentes. Este vrus utiliza processos de splicing alternativo, e provoca o sndroma respiratrio
| Captulo 11 Vrus
143
Biologia Molecular - 1 Ano - FFUP
2006/2007
agudo severo (SARS) que caracterizado por um aumento da temperatura corporal e tosses agudas ao incio e arrepios e tonturas medida que decorre o processo de infeco. Quando este termina, comeam a detectar-se grandes dificuldades na respirao.
6.6. Replicao na classe V
VSV O VSV apresenta RNA (-) na sua constituio, no segmentado e tem um conjunto de protenas que fazem parte da prpria cultura da partcula vrica que so necessrias para a sntese da descendncia vrica. Nestes vrus, existem as seguintes protenas: Protenas M (matriz) situadas entre a cpside e o invlucro viral; Protenas G (glicoprotenas) interagem com os receptores celulares; Protenas L e NS - protenas que quando so expressas, formam a transcriptase vrica que permite a formao de mRNA a partir de material genmico de RNA (-). Protenas N envolvidas no controlo da infeco e no revestimento do genoma dos vrus. Ao ser expressa vai controlar a replicao ou vai controlar a formao de material genmico para que ele possa actuar na replicao. expressa em elevada quantidade e o vrus entra num processo de replicao, formando sempre partculas viricas para que possa lanar para o exterior a sua descendncia. Quando isto acontece, a concentrao da proteina N mais baixa, deixa de ser sintetizada e faz com que haja tambm menor quantidade de mRNAs, para serem encapsuladas novamente.
Influenza A O vrus influenza A apresenta, geralmente, uma forma esfrica de 100 nm, e tem um genoma de 13 Kb dividido em 8 segmentos, existindo tambm outras formas de influenza como o B e o C que podem ter 9 segmentos. A replicaao ocorre no nucleo e e feita a nvel das 8 nucleocpisdes que formam um virio.
144
Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
O envelope contm protenas no estruturais, e ribonucleoprotenas que esto associadas ao material genmico. As protenas do envelope so: HA (hemaglutinina) trmero de glicoprotenas envolvido na adeso NA (neuraminidase) tetrmero envolvido na libertao do endossoma M1 protena membranar da matriz M2 protena membranar dos canais de ies (ausente na influenza B e C) Relativamente s ribonucleoprotenas (RNPs), as principais caractersticas deste vrus so: 8 segmentos de ribonucleoprotenas por genoma Cada um dos 8 (-) segmentos esto envolvidos por nucleoprotenas Uma cpia do segmento da polimerase (PB1, PB2, PA e produtos dos segmentos 1-3) no terminal 3 de cada segmento. Por fim, quanto etiologia do vrus, os principais factos so: Passa de pessoa em pessoa por aerossol, directa ou indirectamente (no h vectores), animais de criao e aves; Causa dores de cabea, mialgias, febre e tosse; O perodo de incubao era aproximadamente de 1 a 3 dias; Os sintomas durante entre 2 e 7 dias; A intensidade dos sintomas depende da espcie vrica; As espcies so descritas com a anteginicidade de HA e NA (que so designados por nmeros).
| Captulo 11 Vrus
145
Biologia Molecular - 1 Ano - FFUP
2006/2007
6.7. Replicao na classe VI
Retrovrus Os retrovrus caracterizam-se por apresentarem um genoma de RNA (+) que serve de molde para a sntese de DNA de cadeia dupla pela transcriptase reversa. Fazem parte dos retrovrus os agentes causadores do cancro e da SIDA, sendo que os seus genomas integram trs principais genes: Gag codifica para um precursor poliproteico que depois clivado e forma as protenas da cpside; Pol codifica para a transcriptase reversa e para a integrase; Env codifica para o precursor das glicoprotenas do envelope Para alm disto, no genoma dos retrovrus, existem: Duas regies no traduzidas, U5 e U3, nas extremidades 5 e 3, necessrias replicao e transcrio; Uma sequncia directamente repetida (R) em cada extremidade do genoma; Uma molcula de tRNA na extremidade 5.
6.8. Replicao na classe VII
Hepadnavirus O hepadnavirus contm uma cadeia de DNA (-) completa e uma cadeia de DNA (+) incompleta. Este vrus tem uma particularidade no processo de replicao porque, inicialmente, vai entrar dentro da clula, vai completar a sntese do DNA, de forma a ter duas cadeias de DNA completas e depois, durante o processo de infeco vai colocar o RNA dentro da clula, formando o pr-genoma e quando passa a DNA de cadeia incompleta por aco da transcriptase reversa, conduz a hepatites viricas sericas e cancros do figado.
146
Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
No processo de replicao, existe a particula vrica completa que tem no seu interior o DNA incompleto e uma molcula peqena que a DNA polimerase do vrus. Quando o material genomico est todo completo (DNA de cadeia dupla) h a sntese de RNA que vai constituir o pr-genoma e inserido dentro do pre-core, conduzindo passagem para DNA de cadeia dupla incompleta. Por fim, o produto final empactotado, e ocorre a formao de uma cpsula.
7. Ciclo de vida do retrovrus (classe VI)
Os retrovrus inserem cpias de DNA (provirus) de um genoma de RNA viral no interior de cromossomas da clula-hospedeira. Os principais passos que intervm no ciclo so: a converso do RNA viral em DNA, a integrao do DNA no genoma hospedeiro e, por fim, a transcrio desse DNA para RNA. A enzima responsvel pela obteno da cpia inicial de DNA a partir do RNA viral a transcriptase reversa. Esta enzima converte o RNA numa molcula linear dupla de DNA no citoplasma da clula infectada, sendo que o DNA tambm transformado para adquirir formas circulares.
| Captulo 11 Vrus
147
Biologia Molecular - 1 Ano - FFUP
2006/2007
O DNA linear segue o seu percurso no ncleo. Uma ou mais cpias de DNA comeam a ser integradas no genoma, sendo que a enzima integrase responsvel por este mecanismo. O provirus transcrito pela maquinaria da clulahospedeira para produzir RNAs virais que servem os mRNAs e os genomas para serem transportados em viries, fazendo com que a integrao seja uma parte normal do ciclo de vida do retrovrus e seja sempre necessria para a transcrio. A partir do momento em que o DNA entra, o core libertado e h a passagem de RNA a DNA a nvel do citoplasma, que por sua vez vai sintetizar mais molculas de RNA atravs da transcrio da sua informao gentica. Por fim, este DNA j poder sair do ncleo para o citoplasma se for para formar partculas vricas completas, ou pode ser processado uma vez que contm intres para facilitar a infeco do vrus.
7.1. Transcriptase reversa
A transcriptase reversa a enzima que tem a capacidade de sintetizar uma cadeia complementar de DNA relativamente a uma cadeia inicial de RNA. Pode ser utilizada in vitro para originar as cadeias simples de DNA mas no caso particular do retrovrus, tem a capacidade de criar a molcula de cadeia dupla do DNA. Para alm disso, uma enzima que consegue degradar o prprio RNA que serviu de molde para a sntese do DNA, atravs da funo de RNase H. Por fim, tambm apresenta funo de clivagem, visto que consegue cortar a extremidade 5 do RNA que serviu de primer (sequncia U3).
7.2. Sntese de DNA no retrovrus
Na reaco de sntese do DNA no interior do retrovrus, a regio R na extremidade 5 da molcula de RNA degradada pela actividade de transcriptase reversa da RNase H. A sua remoo permite que a regio R da extremidade 3 emparelhe com a nova cadeia de DNA, agora sintetizada, e faz com que a transcrio reversa continue atravs da regio U3 para o interior da molcula de RNA. A fonte da regio R que emparelha com a molcula de DNA (-) pode ser tanto a extremidade 3 de uma mesma molcula de RNA (emparelhamento intramolecular) como a extremidade 3 de uma molcula diferente de RNA (emparelhamento intermolecular). A troca para um molde diferente de DNA utilizada para evidenciar que a sequncia primer de tRNA no derivada do ciclo de vida do retrovrus. 148 Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
O resultado desta troca e da extenso a adio de um segmento U3 extremidade 5. A condensao da sequncia U3-R-U5 designada por LTR (long terminal repeat) devido sequncia semelhante de acontecimento que adiciona um segmento U5 extremidade 3, conferindo-lhe a mesma estrutura de U5-R-U3. , agora, necessrio criar a cadeia complementar (+) de DNA e a sequncia LTR na outra extremidade. A transcriptase reversa sintetiza a cadeia (+) de DNA a partir de um fragmento de RNA que deixado a degradar a molcula original de RNA. Este DNA transferido para a outra extremidade da cadeia (-), podendo ser libertado atravs de uma reaco de desagregao quando a segunda parte da sntese de DNA tem lugar a partir de um fragmento de primer ligeiramente acima na cadeia. Ele usa a regio R para emparelhar com a extremidade 3 da cadeia (-) de DNA e a cadeia dupla de DNA agora formada requer a finalizao para originar um duplo LTR em cada extremidade.
7.3. Elementos genticos do DNA provrico e seus produtos
Um provirus retroviral normal encontra-se, geralmente, integrado no DNA celular. Apesar de as duas sequncias LTR serem idnticas, as suas posies fazem com que a LTR esquerda seja identificada como um promotor na transcrio e a LTR direita como especificador para o local poli-A do transcrito. O transcrito primrio de RNA apresenta trs funes funcionais:
Actua como mRNA para a sntese das protenas do Gag e do Gag-Pol pois o sinal de terminao da sua primeira tradu removido ocasionalmente para dar lugar a algumas protenas Gag-Pol. identificado como genoma do vrus Intervm no splicing pois cerca de 50% das molculas sofrem o processamento.
| Captulo 11 Vrus
149
Biologia Molecular - 1 Ano - FFUP
2006/2007
O transcrito j processado, que no empacotado devido remoo do sinal necessrio para o efeito, serve o mRNA para a protena Env. Todos os trs produtos da traduo so considerados produtos viries: a protena Env inserida no interior da bicamada lipdica que rodeia o vrus, a protena Gag forma como que uma concha interna e a protena Gag-Pol (que contm a transcriptase reversa) tambm se encontra no interior da partcula. Por fim, quando as partculas terminam o seu processo de formao, as protenas Gag e Gag-Pol so extensivamente processadas por protelise.
7.4. Caractersticas das LTR
Embora as duas LTR sejam regies praticamente iguais, tm funes diferentes consoante a extremidade em que se encontra. A LTR da extremidade 5 tem sinais para a RNA polimerase, como a TATA box e a a CAAT box e tem locais de ligao para protenas activadoras, logo uma vez sintetizada e ligada, vai permitir a formao de todos os genes correspondentes regio codificante, mas s se tambm tiver sinais de paragem para a finalizao deste processo. Estes sinais de terminao so os que se vo encontrar na extremidade 3 juntamente com sinais de transcrio e poliadenilao, caractersticas desta extremidade.
150
Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
8. SIDA
8.1. O HIV como retrovrus
O HIV foi detectado e constitudo como vrus causador de doenas no incio dos anos 80 e foi diagnosticado num indivduo que apresentava grandes deficincias do sistema imunitrio. Inicialmente, o HIV infecta clulas vitais do sistema imunitrio, como linfcitos T helpers (especificamente os seus receptores CD4+), macrfagos e clulas dendrticas. A infeco do HIV conduz reduo dos nveis das clulas CD4+ atravs de trs principais mecanismos. Em primeiro lugar, mata directamente as clulas infectadas; depois, aumenta as taxas de apoptose nas clulas infectadas e, por fim, destri as clulas CD4+ atravs de linfcitos citotxicos CD8 que reconhecem facilmente as clulas infectadas pelo vrus. Quando as clulas CD4+ baixam e atingem um nvel crtico, a imunidade mediada pelas clulas perdida totalmente e o organismo comea a tornar-se progressivamente mais vulnervel infeco por doenas oportunistas. Quando no tratado, os indivduos infectados pelo retrovrus HIV desenvolvem a SIDA (Sndroma da Imunodeficincia Adquirida) e morrem. No entanto, um em cada dez indivduos permanece relativamente saudvel durante um maior perodo de tempo, sem quaisquer sintomas assinalveis. As principais patologias associadas ao vrus do HIV so o sarcoma de Karposis e a pneumonia (pneymocystis carinii) e a mdia de partculas vricas de HIV por 1 mL de fluido corporal a seguinte:
A transmisso do HIV feita por uma grande variedade de processos, sendo que as mais comuns so: Via sexual Transfuses (sangue e factores de coagulao) Utilizao de agulhas comuns na administrao de drogas Transmisso materna pela placenta (30% das mes infectadas transmitem o vrus aos descendentes)
| Captulo 11 Vrus
151
Biologia Molecular - 1 Ano - FFUP
2006/2007
8.2. Estrutura do HIV humano
O HIV diferente, em estrutura, de todos os outros retrovrus. Tem cerca de 120 nm de dimetro e uma forma aproximadamente esfrica. Basicamente, o HIV-1 composto por duas cpias de RNA de cadeia simples, fechado no interior de uma cpside de forma cnica que contm a protena viral p24 (tpica dos lentivrus) e que, por sua vez, revestida por uma membrana plasmtica da clulahospedeira. A cadeia simples de RNA est estreitamente ligada s protenas da nucleocpside (p7) e enzimas que so indispensveis para o desenvolvimento do virio, como a transcriptase reversa e a integrase. A nucleocpside (p7 e p6) associa-se com o RNA genmico (uma molcula por hexmero) e protege o RNA da sua digesto pelas nucleases. Uma matriz constituda por uma associao da protena viral p17 envolve a cpside, assegurando a integridade da partcula vrica. Outras partculas tambm includas no interior da partcula vrica so os Vif, Vpr, Nef, p7 e uma protena viral, sendo que o envelope da membrana formado quando a cpside sai da clula-hospedeira e leva consigo alguns elementos da sua membrana, e constitudo pelas glicoprotenas gp120 e gp41.
8.3.
Ciclo de infeco do HIV
A glicoprotena gp120 localizada na superfcie das parculas de HIV e, consequentemente, estabelece uma ligao com a CD4 e um de dois coreceptores da clula (CCR5 ou CXCR4). Desta forma, o contedo viral entra para a clula por endocitose, sofrendo uma catlise pelo RNA viral. A clula-hospedeira sintetiza, de seguida, uma cadeia complementar de DNA formando, ento, uma dupla cadeia de DNA que direcciona a sntese de RNA de HIV e protenas respectivas. Por fim, as partculas de HIV completas so reconhecidas, sendo que nos macrfagos, o HIV sai da clula por exocitose, mas nas clulas T a sada feita atravs da ruptura da clula libertando o HIV na corrente sangunea.
152
Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
8.4.
Ciclo de vida do HIV
No processo de infeco do HIV, o retrovirus passa para o interior dos macrfagos e das clulas CD4, atravs da adsoro das glicoprotenas sua superfcie, e de receptores na clula-hospedeira, seguida da fuso do envelope viral com a membrana celular e da libertao da cpside do HIV para o seu interior. As interaces do complexo trimrico do envelope nuclear, com uma clula CD4 e com um coreceptor de quimocinas (CCR5 ou CXCR4) ocorrem na superfcie da clula. A ponta gp160 contm domnios de ligao para a CD4 e para a quimocina, logo o primeiro passo na fuso de membranas envolve uma ligao de alta afinidade da CD4 gp120. Assim que a gp120 estiver ligada protena CD4, o complexo de envelope viral conduz a uma alterao estrutural, expondo uma quimocina com domnios para a gp120 e permitindo que eles interajam. Assim que a partcula vrica est incorporada na clula, a transcriptase reversa liberta a cadeia simples de RNA (+) das protenas virais anexadas e copia-a para uma molcula complementar de DNA com 9 Kb de tamanho. Este processo de transcrio reversa extremamente susceptvel de formar erros e durante esta fase que a maior parte das mutaes ocorre, podendo causar resistncia s drogas. A transcriptase reversa origina uma cadeia complementar de DNA para formar a cadeia dupla interna de DNA viral (vDNA) que vai ser transportada para o ncleo e a sua integrao do DNA viral no genoma da clula transportada pela integrase. Este DNA viral, agora integrado, mantm-se inactivo, no estado latente da infeco do HIV. Para produzir activamente o vrus, vrios factores de transcrio tm de estar presentes, sendo o mais importante o NF-kB, que regulado quando as clulas T so activadas, significando que estas clulas que sofrem a aco directa da infeco do HIV so intervenientes no combate sua morte. No processo de replicao, o provirus integrado copiado para um mRNA que seguidamente vai sofrer splicing originando partculas mais pequenas. Por sua vez, estas partculas produzem vrias protenas reguladoras da classe Tat e Rev. medida que as Rev se vo acumulando gradualmente, comeam a inibir o
| Captulo 11 Vrus
153
Biologia Molecular - 1 Ano - FFUP
2006/2007
splicing do mRNA. Nesta fase, as protenas estruturas Gag e Env so produzidas a partir do mRNA de maiores dimenses, e o RNA total , agora, o genoma total do vrus, ligando-se s partculas Gag para originar novas partculas vricas. Na fase final do ciclo, ocorre o reconhecimento de novos vrus HIV-1 a partir da membrana plasmtica da clula-hospedeira. A poliprotena Env (gp160) atravessa o retculo endoplasmtico e transportada para o complexo de Golgi onde clivada pela aco de uma protease e processada para a obteno de duas protenas do envelope vrico: gp41 e gp120. Por sua vez, estes passam para a membrana plasmtica da clula-hospedeira, onde se mantm alojados. As poliprotenas Gag e Gag-Pol tambm se associam a essa mesma superfcie, juntamente com o RNA do genoma do HIV para formar o virio. A maturao pode ocorrer na estrutura recentemente formada ou no virio imaturo depois de se ter formado na clulahospedeira. Durante a maturao, as proteases do HIV clivam as poliprotenas em protenas e enzimas individualmente funcionais do HIV e o vrus maduro j pode infectar uma nova clula.
8.5.
Funes dos genes do HIV-1
O HIV tem uma grande parte dos genes que codificam para protenas estruturais que so encontradas em todos os retrovrus e vrios genes no estruturais (acessrios) que so nicos para o HIV. Os principais genes do HIV-1 so: Env codifica para os precursores das glicoprotenas gp120 e gp41, que so protenas localizadas no envelope viral e que ajudam o vrus a ligar-se e a fundir-se com as clulas-alvo. Pol codifica para enzimas virais, sendo as mais importantes a transcriptase reversa, a integrase e a protease que corta as protenas provenientes do Gag para protenas funcionais. Gag codifica para vrias protenas estruturais como a p24 para a cpside viral, pa 6 e a p7 para as protenas da nucleocpside, a p17 para a protena matriz e a p55 que cortada em quatro protenas estruturais menores pela protease. Tat (Trans-activator of Transcription) auxilia o HIV a reproduzir-se atravs da compensao de um determinado defeito no seu genoma, atravs da sntese de RNA. Rev (Regulator of Virion) permite que os fragmentos de mRNA do HIV que contenham RRE (Rev Response Unit) sejam exportados do ncleo para o citoplasma. Na falta deste gene, a maquinaria do splicing de RNA no ncleo rapidamente com o objectivo de obter menores protenas reguladoras. Na presena do Rev, o RNA exportado do ncleo antes de sofrer splicing, fazendo com que as protenas estruturais e o genoma do RNA possam ser produzidos. Nef (Negative Regulatory Factor) a sua expresso no ciclo de vida do vrus assegura a activao das clulas T e o estabelecimento de um estado de infeco persistente. Para alm disso, promove a sobrevivncia das clulas infectadas atravs da modulao da expresso de vrias molculas da superfcie que so importantes para a funo imune da clula. Vif (Viral Infectivity Factor) essencial para a replicao do vrus, sendo que inibie uma protena celular ao virio durante a separao de uma clula-hospedeira atravs da degradao proteossomal. Vpu (Viral Protein U) envolvida na separao e formao final do vrus, no momento em que o virio se desprende da clula.
154
Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
Vpr (Viral Protein R) desempenha um papel importante na regulao da importao nuclear do complexo de pr-integrao do HIV-1 e requerida para a replicao viral em clulas no divisveis como os macrfagos. Para alm disso, induz a interrupo do ciclo celular e a apoptose em clulas proliferativas, provocando disfunes imunitrias.
8.6.
Terapias
8.6.1. Terapia pela AZT e DDI
O AZT actua como um terminador de cadeia durante a sntese do DNA provrico atravs da transcriptase reversa. O AZT uma molcula anloga timidina excepto num grupo hidroxilo do carbono-3 do anel desoxirribonucleico que foi substitudo por um grupo azotado (N3). Os nuclesidos so fosforilados antes de terem sido adicionados na cadeia nascente de DNA. Uma ponte fosfodiester criada entre o grupo hidroxilo do ltimo nucletido incorporado e o trifosfato desoxirribonuclesido que vai ser introduzido. Se o ltimo nucletido incorporado foi o AZT, o grupo hidroxilo est em falta, logo o prximo nucletido no pode ser adicionado e a sntese termina, sendo que isto acontece porque o AZT uma didesoxipirimidina.
8.6.2. Terapia com inibidores de proteases
Esta terapia relaciona-se com o processamento de proteases da Pr55 e da Pr160, que so duas poliprotenas produzidas a partir dos genes Gag e Pol e cortadas pela protease de HIV em protenas maduras.
| Captulo 11 Vrus
155
Biologia Molecular - 1 Ano - FFUP
2006/2007
O precursor Gag-Pol Pr160 cortado em trs protenas mais pequenas: a protease propriamente dita, a transcriptase reversa e a integrase que est envolvida na integrao proviral no interior do DNA ceular. O precursor Gag Pr55 comanda o processamento mais complexo, produzindo inicialmente p17, p24 e p15, sendo que este ltimo segmento comanda o passo seguinte para originar p7 e p6. Todas as protenas Gag so protenas estruturais do HIV inner core.
8.6.3. Terapia com molculas de CD4 solveis
Neste processo de terapia, ocorre o armadilhamento intracelular da gp120, utilizando uma molcula recombinante de CD4. Depois da sua sntese nos ribossomas anexados ao retculo endoplasmtico, as protenas como a gp120 e a CD4 passam para o interior do retculo endoplasmtico e so transportadas para a superfcie celular. Outras molculas que actuam dentro do retculo endoplasmtico so retidas pela aco de uma sequncia de aminocidos especial, no seu grupo carboxilo. Esta sequncia Lys-Asp-Glu-Leu (KDEL). Uma forma solvel da CD4 recombinante foi criada num local onde os domnios transmembranar e citoplsmico foram submetidos pela KDEL (sCD4KDEL). Desta forma, conclui-se que esta molcula ligar-se-ia aos receptores existentes no retculo endoplasmtico, onde ficaria retida. Um plasmdeo expressante da sCD4-KDEL foi transferido para clulas HeLa, juntamente com um plasmdeo expressante da gp120. Assim, as clulas cresceram num meio contendo metionina marcada radioactivamente para marcar protenas agora sintetizadas. A gp120 marcada no citoplasma das clulas e no meio de cultura foi imunoprecipitada e analisada por electroforese, tendo sido encontrada em ambos os locais quando o plasmdeo contendo gp12 foi transferido sozinho ou com CD4. Desta forma, quando modificado com sCD4-KDEL, a gp120 apenas foi encontrada nas clula, sugerindo que os complexos gp120-sCD4-KDEL ainda se encontrassem retidos no retculo endoplasmtico. 156 Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
8.6.4. Terapias para o futuro
Terapia de combinao refere-se administrao simultnea de dois ou mais medicamentos para tratar a mesma doena, ou quando so usados dois tratamentos ao mesmo tempo. No caso do HIV, existem inibidores de proteases que actuam sobre as protenas da cpside, da replicao e do envelope viral e o AZT que actua no processo de replicao, e fazem com que a actividade de determinados genes virais sejam expressos em quantidades mais controladas e de menor nvel de gravidade. Vacina ou Terapia por Drogas os componentes da vacina incorporam o gene nef que apresenta caractersticas defeituosas, sendo que simultaneamente existe uma protena inibidora do gene nef que impede a formao de uma protena crtica para a infeco vrica do HIV. Receptores de bloqueio existe um receptor de quimocina que est bloqueado, impedindo a movimentao do coreceptor CCR5 e CXCR4. Receptores de desactivao o coreceptor est mutado e desta forma inibido e desactivado de exercer as funes de ligao na fase inicial do ciclo de infeco do HIV. Bloqueio de replicao interveno do CAF que impede a replicao das sequncias vricas e a consequente infeco total do indivduo pelo vrus.
9. Agentes com propriedades semelhantes aos vrus
9.1. Virides
Os virides so patgenos de plantas que consistem em pequenos fragmentos (com alguns centenas de nucleobases) de RNA de cadeia simples, circular e com grande complementaridade, no apresentando a cpsula viral que tpica nos vrus. O mais pequeno que se conhece actualmente tem 220 nucletidos de scRNA (small cytoplasmic RNA) e est associado ao RYMV (rice yellow mottle sobenovirus). Comparativamente, o genoma dos mesmos vrus conhecidos capazes de causar uma infeco por mais pequena que seja tem 2 Kb de tamanho. O RNA dos virides no codifica para nenhuma protena conhecida, e alm disso alguns deles nem contm o codo de iniciao AUG. O mecanismo de replicao envolve a interaco com a RNA polimerase II, enzima geralmente associada sntese de mRNA e sntese do rolling circle do novo RNA. Alguns virides so ribozimas, existindo propriedades de enzimas de RNA que permitem a sua auto-clivagem e a ligao de genomas unitrios a partir de maiores intermedirios de replicao. No homem, os virides provocam geralmente a hepatite D, pois o viride em causa est na cpside do vrus da hepatite B, sempre num processo de sequestro ou destruio do 7S RNA heptico, que um componente da SRP.
| Captulo 11 Vrus
157
Biologia Molecular - 1 Ano - FFUP
2006/2007
9.2.
Pries
Os pries so agentes infecciosos com capacidade de modificar outras protenas, tornando-as cpias de si mesmas. Um prio no possui qualquer cido nucleico na sua constituio, sendo actualmente conhecidas 13 espcies de pries, das quais 3 atacam fungos e 10 atacam mamferos. Geralmente, no caso do ser humano, um produto do gene humano PrP localizado no cromossoma 20, fazendo com que essa mesma protena PrP esteja envolvida no mecanismo de diferenciao neuronal e no sequestro de ies Cu2+.
9.2.1. Pries no ser humano
No ser humano, os pries podem surgir em quatro principais caractersticas: Espordico (> 85%) Doena de Creutzfeldt-Jakob (CJD) Insnia fatal espordica Gentico (< 15%) Aparentemente autossmica dominante CJD familiar (fCJD) Sndroma de Gerstmann-Strussler-Scheinker (GSS) Insnia fatal espordica Ambiental / Intrognica Kuru Nova variante da CJD (vCJD) Contaminao com hormona de crescimento humana Transplantes de crnea e dura mater Equipamento contaminado Elctrodos de EEG Instrumentos neurocirrgicos
9.2.2. Transmisso e infeco
Geralmente, a contraco de determinadas doenas atravs de pries feita de forma horizontal, sendo que a transmisso feita directamente de pessoa para pessoa ou atravs da ingesto de carne proveniente de animal previamente infectado. Relativamente infeco em particular, o processo geralmente envolve cirurgias, injeco de hormonas, transplantes de crnea ou alteraes no gene PrP. 158 Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
9.2.3. Tipos de pries
PrPc um PrP celular, constitudo pela glicoprotena da superfcie celular da membrana. Apresenta uma estrutura secundria em -hlice, surgindo como prio solvel e facilmente dirigido por proteases.
PrPsc relativamente estrutura primria, semelhante do PrPc, mas a estrutura secundria apresenta conformao . Desta forma, insolvel, pode ser automaticamente obtida a partir do PrPc e faz com que se possam agregar entre si e depositar-se na forma de placas de amitide para provocar degenerescncia e deteriorao cerebral.
9.2.4. Doenas causadas pelos pries em animais
BSE (encefalopatia espongiforme bovinal) caracterizada por ser a doena das vacas loucas e pelas seguintes reaces: Agitao e agressividade Perda de funes motoras Perda de apetite Convulses Cegueira Auto-mutilao Dificuldade em engolir Scrapie TME (encefalopatia transmissvel da marta) CWD (chronic wasting disease) ocorre em alces e veados
| Captulo 11 Vrus
159
Biologia Molecular - 1 Ano - FFUP
2006/2007
9.2.5. Doenas causadas pelos pries no homem
Kuru a primeira descrio desta doena foi feita em 1957, devido ao canibalismo FFI (insnia familiar fatal) insnia progressiva, com atrofia severa e selectiva do tlamo GSS (sndroma de Gerstmann-Straussler-Scheinker) provoca ataxia cerebral, problemas motores, demncia e ocorre entre os 40 e 50 anos. Sndroma de Alper ocorre nas crianas CJD (doena de Creutzfeld-Jacob) perda do controlo motor, demncia, paralisia e morte, sendo transmitida por instrumentos cirrgicos mal esterilizados ou por transplante de rgos. vCJD ou fCJD nova variante de CJD proveniente do consumo de produtos de animais contaminados com BSE.
9.2.6. Mtodos laboratoriais para confirmao do diagnstico
No h testes sanguneos para deteco de pries para rastreio de portadores; Identificao por exame microscpico de tecido cerebral; Tcnicas de microscopia e colorao para deteco de formas resistentes protelise da protena do prio modificado (PrPres).
160
Captulo 11 Vrus |
Biologia Molecular 1 Ano - FFUP
2006/2007
Captulo 12 Mutaes no DNA
As mutaes so mudanas repentinas que ocorrem nos genes, ou seja, o processo pelo qual um gene sofre uma mudana estrutural. Geralmente, as mutaes distinguem-se das aberraces por serem alteraes a nvel de ponto, envolvendo a eliminao ou substituio de um ou poucos nucletidos da cadeia de DNA. Podem ter uma origem causal ou induzida na informao gentica e s transmitida para os descendentes de organismos complexos se ocorrer em clulas germinativas. Para que haja mutao primeiro necessrio que ocorra um dano na sequncia de nucletidos do DNA. As clulas possuem um conjunto de mecanismos de reparao de DNA encarregues de corrigir o possvel erro, mas ocasionalmente pode ocorrer uma falha nesses mecanismos, ou o dano pode ser irreparvel e as clulas replicam-se nestas condies de mutao. As mutaes podem ter vrias origens: podem ser ocasionais, tomando parte na pequena probabilidade do erro espontneo no momento da duplicao de DNA na mitose ou na meiose; podem ser provocadas por agentes mutagnicos de origem electromagntica, qumica ou biolgica; podem ainda ser induzidas em laboratrio com o uso intencional destes mesmos agentes sobre organismos vivos. As bactrias so ptimos modelos para estudar mecanismos genticos, porque para alm de se reproduzirem rapidamente, tm apenas uma cpia de cada gene. Portanto, qualquer mutao conduz directamente a uma alterao fenotpica. As mutaes actuam de forma crucial na evoluo das espcies. As alteraes morfolgicas, nas quais a teoria da seleco natural se baseia, devem-se a mutaes que promovem o surgimento de novas caractersticas numa determinada populao, que por um motivo ou por outro faz com que os seus portadores sejam mais bem sucedidos que os seus concorrentes e predecessores. Da mesma forma, as mutaes que produzem indivduos menos adaptvis ao seu meio tendem a ser rapidamente eliminadas pelos seus concorrentes, j que a probabilidade de um indivduo menos adaptado se reproduzir menor. Os principais tipos de mutaes incluem: Mutaes espontneas isomerizao, depurinao, desaminao e leso oxidativa Mutaes induzidas atravs da interveno de agentes fsicos ou qumicos Mutaes pontuais Recombinao Rearranjos cromossmicos
| Captulo 12 Mutaes no DNA
161
Biologia Molecular - 1 Ano - FFUP
2006/2007
1. Mutagnese
1.1.Mecanismos de mutagnese
Existem principalmente trs mecanismos de mutagnese que esto envolvidos nas mutaes de DNA referidas anteriormente, estando todos eles relacionados entre si apesar das diferenas existentes nos processos. Desemparelhamento especfico comandado por mutagnios que alteram especificamente uma base no DNA e causam mutaes. devido aco de determinados agentes especficos que podem levar mutao: Agentes alquilantes etilmetano sulfonaoto e nitrosoguanidina Agentes intercalantes proflavina, acridina e compostos ICR Agentes que causam desaminao ies bissulfito e cido nitroso Incorporao de bases anlogas inclui compostos que so semelhantes s bases do DNA, que so incorporadas numa das cadeias do DNA e provocam mutaes: 5-bromouracil 2-amino-purina Perda de especificadade de emparelhamento mutagnios que se vo ligar ao DNA por terem maior afinidade de conexo, provocando a leso de uma ou mais bases que vai impedir o emparelhamento correcto: Luz UV Aflatoxina B Benzopireno
1.2.Consequncias da mutagnese
As consequncias da mutagnese relacionadas com substituio de bases podem ser a nvel da molcula de DNA ou a nvel da protena formada. A nvel da molcula de DNA, podem existir, essencialmente: transverses, transices e mutaes pontuais. As mutaes pontuais apenas ocorrem numa das bases do DNA, ou seja, num local especfico. Por sua vez, as transies incluem a substituio de uma purina por outra purina ou de uma pirimidina por outra pirimidina e nas transverses, ocorre a troca de uma purina por uma pirimidina e vice-versa. Em termos de molcula proteica, traduz-se em alteraes que podem ser de vrios tipos: Silenciosas - quando h uma base mutada fazendo com que o DNA fique diferente mas a protena resultante se mantenha igual, porque existem vrios codes que especificam para o mesmo aminocido e, por isso, pode haver uma substituio a nvel da molcula de DNA que vai originar o mesmo aminocido no final. 162 Captulo 12 Mutaes no DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
Neutras fazem alteraes a nvel da molcula de DNA e a nvel da protena mas no alteram a sua funo, traduzindo-se em mutaes como a substituio de um aminocido de carcter cido por outro semelhante, que conduz alterao da conformao da molcula mas sempre mantendo a sua funo. Absurdas (Nonsense) existem quando aps a troca de uma base se criou um codo stop. Se a sequncia de DNA codificava um pptido de determinado tamanho, aps a formao de um codo stop, o pptido ir ser mais pequeno e ir conferir alteraes em tudo o que est relacionado com a protena. Sem sentido (Missense) ocorrem normalmente por serem responsveis pela formao de mutantes termosensveis, funcionando apenas com temperaturas especficas para cada caso.
Para alm destas consequncias, existem as mutaes frameshift, que podem levar perda de uma base e a ligao das duas subjacentes, traduzindo-se numa modificao do quadro de leitura final. Ocorrendo a insero de uma nova base na molcula de DNA, faz-se com que o quadro de leitura seja lido de uma forma diferente da inicial, podendo conduzir formao de um codo stop e interrupo da cadeia peptdica.
| Captulo 12 Mutaes no DNA
163
Biologia Molecular - 1 Ano - FFUP
2006/2007
Nas adies e deleces, tambm pode haver inverses que resultam quando temos uma sequncia inicial que pode sofrer cortes das duas cadeias e que quando vai ser reparada, no se coloca a molcula na posio correcta mas sim de forma invertida inicial. Por fim, existem os pontos quentes (hotspots). Quando se consideram as mutaes em termos de inactivao de um gene, a maior parte dos genes no interior de determinadas espcies, apresentam aproximadamente taxas de mutao semelhantes s esperadas relativamente ao seu tamanho. Isto sugere que o gene pode ser considerado como alvo para uma determinada mutao e que o dano causado em qualquer parte do gene que pode abolir a sua funo. Como resultado, a susceptibilidade para a mutao torna-se superior ou inferior, e proporcional ao tamanho do gene em causa. A probabilidade estatstica de que mais de uma mutao ocorre num local particular dada pela cintica da actuao de uma determinada mutao. Desta forma, alguns locais adquirem uma ou mais mutaes ao mesmo tempo que outros locais se mantm inalterados e, por isso, intactos relativamente ao efeito das mutaes. Para alm disso, existem ainda outros locais que ganham um nmero de mutaes muito superior ao nmero de mutaes esperadas numa distribuio aleatria, podendo apresentar dez ou cem vezes mais mutaes que o esperado para os casos aleatorios. Estes locais particulares designam-se por hotspots, que so locais especficos da molcula de DNA onde ocorrem alteraes sistemticas, apresentando consequentemente maior tendncia para ser mutadas.
2. Mutaes espontneas
As mutaes espontneas so mutaes que resultam de operaes celulares normais ou de interaces aleatrias com o ambiente em que o indivduo se encontra inserido.
2.1. Isomerizao
No processo de isomerizao, os tautmeros so compostos orgnicos que so interconvertveis atravs de uma reaco qumica designada tautomerizao. Geralmente, esta reaco definida como sendo uma reaco que resulta na migrao formal de um proto ou de um tomo de hidrognio, acompanhada da troca de uma ligao simples e de ligao dupla adjacente.
164
Captulo 12 Mutaes no DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
Em solues em que pode ocorrer tautomerizao, atinge-se um equilbrio qumico dos tautmeros. A sua proporo depende de vrios factores como a temperatura, o solvente utilizado e o pH. O conceito de tautmeros interconvertveis pela tautomerizao designa-se tautomerismo e um caso especial de isomerismo estrutural, podendo desempenhar um papel importante no emparelhamento de bases no DNA e em molculas especficas de RNA. As reaces de tautomerizao so catalisadas por cidos ou por bases, sendo os dois pares tautomricos mais comuns os seguintes:
Um exemplo comum o da guanina que passa da forma ceto forma enol, depois da alterao tautomrica no momento da replicao. Na sua forma enol, a guanina emparelha com a timina em vez de emparelhar com a citosina, fazendo com que na replicao seginte, a guanina volte a trocar para a sua forma ceto, que mais estvel. A timina incorporada na forma enol da guanina direcciona a incorporao da adenina na replicao subsequente, fazendo com que o resultado seja uma mutao G.C - A.T. Se a guanina sofre uma alterao tautomrica no momento de incorporao (como um nuclesido trifosfato), ela vai ser incorporada na timina novamente na cadeia em causa e provoca uma mutao A.T G.C. Isto acontece porque uma das cadeias fica modificada estruturalmente pois uma citosina isomerada passa a estar presente na molcula, fazendo com que haja uma molcula GC e uma molcula GT que no respeita as regras normais de emparelhamento.
| Captulo 12 Mutaes no DNA
165
Biologia Molecular - 1 Ano - FFUP
2006/2007
2.2. Depurinao
Na depurinao, ocorre a perda de uma base da molcula de DNA devido falta da ligao glicosdica entre a base e a desoxirribose, formando um local apurnico. Geralmente, so mutaes que ocorrem em grande nmero, temperatura corporal do indivduo durante cerca de vinte horas, sendo que no final so corrigidas por sistemas de recuperao que possam existir.
2.3. Desaminao
Na desaminao, qualquer uma das bases pode sofrer mutao: Citosina Uracilo 5-metilcitosina Timina Adenina Hipoxantina Guanina Xantina Numa determinada molcula de DNA, por exemplo, pode ocorrer a desaminao de uma citosina para formar um uracilo. O par de bases resultante U-G reconhecido como sendo anormal e pode vir a ser reparado para restaurar o par normal C-G. Alternativamente, a mutao pode ser reparada na replicao de DNA que se segue. Depois de uma replicao de DNA, uma molcula filha de DNA vai apresentar um par de bases U-A (mutante) e a outra um par C-G (normal). O uracilo vai ser removido e substitudo por uma timina, gerando um DNA mutante em que o par T-A substitudo por um par C-G.
166
Captulo 12 Mutaes no DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
2.4. Leso oxidativa
As reaces oxidativas levam formao ou a alteraes que se traduzem por transverses, resultando do metabolismo aerbio normal das bases do DNA. Normalmente, as mutaes de leso oxidativa formam, essencialmente, dois produtos: a timinoglicol e a 8-oxo-7,8-dihidrodioxiguanina, ambos provocados por radicais livres de oxignio, espcies reactivas como radicais superxido (O2-), perxido de hidrognio (H2O2) e radicais hidroxilo (OH). As transverses obtidas atravs desta classe de mutaes so maioritariamente reparadas pelo sistema GO.
3. Mutaes induzidas
As mutaes induzidas resultam da exposio dos organisos a determinados agentes mutagnicos que reagem com o DNA, como a luz UV, radiaes ionizantes ou vrios agentes de carcter qumico.
3.1. Desemparelhamento especfico
No caso da hidroxilamina (HA), esta molcula pode ligar-se citosina para formar uma cetocitosina que passa a ter a capacidade de emparelhamento com a adenina. Este emparelhamento no se traduz num emparelhamento normal e, consequentemente, causa alteraes a nvel do DNA resultante. Isto acontece porque neste tipo de mutaes induzem-se vrias transies, fazendo com que as molculas emparelhadas sejam diferentes das esperadas.
3.2. Agentes alquilantes
Os agentes alquilantes podem ser responsveis pela transformao da timina na O-6-etilguanina, ou da timina noutro composto que tem capacidade de emparelhar com a guanina. No h, de facto, nenhuma
| Captulo 12 Mutaes no DNA
167
Biologia Molecular - 1 Ano - FFUP
2006/2007
incorporao como nas baes anlogos, porque se encontra um grupo adicional que colocado na prpria base que faz com que, se isto no for reparado atempadamente, ocorram determinadas reaces, nomeadamente antes do processo de replicao. Aqui, o DNA vai sofrer modificaes e se for adicionado o grupo metilo e ocorrer a replicao subsequente, uma das cadeias continua GC e a outra passa a G.T, conferindo alteraes a nvel do NA obtido no final. Os principais agentes alquilantes so o etilmetano sulfonato (EMS) e a nitrosoguanidina (NA) que apresenta uma grande afinidade para os garfos de replicao. Todos os agentes alquilantes causam transies na molcula de DNA mas no so incorporados nela.
3.3. Agentes intercalantes
Os agentes intercalantes vo intoduzir-se no meio da molcula de DNA e, com isto, podem criar adies ou deleces. Durante o processo de replicao, este tipo de alteraes ocorre frequentemente. A molcula de DNA, durante o processo de sntese, pode sofrer uma adio de uma paragem comandada pela DNA polimerase, cuja consequncia a formao de um loop porque uma determinada sequncia lida duas vezes, produzindo uma cadeia com mais um tripleto que a inicial. Por paragem da DNA polimerase, seguidamente, pode haver um recuo e uma deleco de uma determinada base.
168
Captulo 12 Mutaes no DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
3.4. Desaminao oxidativa
A desaminao oxidativa pode ser provocada pelo cido nitroso ou pelos ies bissulfito, e pode envolver a citosina ou a 5-metilcitosina. Os efeitos do cido nitroso conduzem a um exemplo clssico de uma transio causada pela converso qumica de uma base noutra. O cido nitroso intervm na desaminao oxidativa que converte a citosina no uracilo. No ciclo de replicao, depois da transio, o uracilo emparelha com a adenina em vez de a guanina emparelhar com a citosina original. Consequentemente, o par CG substitudo por um par T.A quando a adenina emparelha com a timina no ciclo de replicao seguinte. O cido nitroso tambm pode desaminar a adenina, originando a hipoxantina que vai emparelhar com a citosina e conduz transio inversa a partir do par AT para o par GC.
A desaminao da 5-metilcitosina um processo relativamente raro, que processa uma mutao que apenas ocorre porque os sistemas de reparao so muito mais eficientes em reconhecer o par GU que o par G.T. A E. Coli contm uma enzima, a uracilo-DNA-glicosidase, que remove os resduos de uracilo do DNA. Esta aco deixa um resduo guanlico no emparelhado e um sistema de reparao que, mais tarde, insere uma citosina para emparelhar com a guanina anteriormente referida. Isto resulta no restabelecimento da sequncia original de DNA. Este sistema protege o DNA contra asc consequncias das desaminaes espontneas da citosina mas, no entanto, a desaminao da 5-metilcitosin deixa uma timina no lugar. Isto permite a formao de um par de bases anormal G.T, sendo que se esta diferena no for corrigida antes do ciclo de replicao seguinte, vai formar-se uma mutao. Na replicao seguinte, as bases do par G.T separam-se para mais tarde emparelharem com novas bases para produzir um par normal GC e um par mutante A.T. A desaminao da 5-metilcitosina a causa mais comum da produo de pares anormais G.T na molcula de DNA. Os sistemas de reparaao que actuam nestes pares apresentam compatibilidade para substituir as timinas pelas citosinas, ajudando a reduzir as taxas de mutao nas molculas. No entanto, estes sistemas no so to eficientes quanto a remoo do uracilo dos pares G.U. Como consequncia, a desaminao da 5-metilcitosina conduz a mutaes mais frequentemente do que a desaminao da citosina.
| Captulo 12 Mutaes no DNA
169
Biologia Molecular - 1 Ano - FFUP
2006/2007
3.5. Incorporao de bases anlogas
No caso da bromodesoxiuridina (BrdU), esta base apresenta-se na maior parte dos casos na forma ceto (5bromouracil), com especificidade para emparelhar com a adenina. No entanto, depois de passar sua forma oxidada, a ligao dupla com o oxignio deixa de existir, a base passa forma enol e passa a emparelhar com a guanina.
O outro caso engloba a 2-aminopurina (2-AP) que antes de sofrer reaco, tem capacidade de emparelhar com a adenina e depois de estar protonada devido interaco com um tomo de hidrognio ou outro proto, passa a emparelhar com a citosina.
Replicao do DNA na presena de BrdU
A bromodesoxiuridina (BrdU) uma base anloga da timina que contm um tomo de bromo no local do grupo metilo que constitui a timina. A BrdU incorporada no interior do DNA no local da timina, mas contm um conjunto de propriedades de emparelhamento que so ambguas devido presena do tomo de bromo que permite a ocorrncia de uma troca na qual as bases trocam estruturalmente de forma ceto (=O) para a forma enol (-OH). A forma enol pode emparelhar com a guanina, permitindo a substituio do par original AT pelo par GC. O emparelhamento errado pode ocorrer ou durante a incorporao original da base, ou num ciclo de replicao subsequente. A transio induzida por uma certa probabilidade em cada ciclo de replicao, para que a incorporao do BrdU apresente efeitos contnuos na sequncia de DNA.
170
Captulo 12 Mutaes no DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
3.6. Perda de especificidade de emparelhamento
Os agentes mais comuns nesta interveno so a aflatoxina B e o benzopireno, sendo que o primeiro responsvel pelos cancros do fgado e o segundo pelas transverses e pelos locais apurnicos na molcula de DNA. As enzimas que realizam a metabolizao so enzimas hepticas e actuam atravs da via citocromo P450, criando um radical hidroxilo (OH) que posteriormente vai ser cortado para permitir a ocorrncia de uma nova reaco. Aqui, obtm-se uma nova molcula com quatro grupos OH que adquire grande afinidade para o DNA, distorcendo-a e criando a mutao consequente.
4. Mutaes pontuais
As mutaes pontuais podem ser de vrios tipos:
Mutaes silenciosas (ou sinnimas) resultam da substituio de um par de bases por outro, que leva formao de um codo diferente, que continua, no entanto, a codificar o aminocido original; no se observa qualquer alterao na funo da protena.
Mutaes missense resultam da substituio de um par de bases por outro, levando formao de um codo que codifica um aminocido diferente do original. Se o codo obtido por mutao, no entanto, codificar um aminocido quimicamente semelhante ao original, no se vo detectar grandes alteraes na funo da respectiva protena. Assim sendo, numa mutao deste tipo, pode obter-se uma protena com funo alterada ou no.
Mutaes nonsense resultam da substituio de um par de bases por outro, que leva formao de um codo stop; h terminao prematura da cadeia polipeptdica e a respectiva protena (truncada) normalmente no funcional.
Mutaes frameshift resultam de adies ou eliminaes de pares de bases que provocam desfasamento do quadro de leitura, obtendo-se uma sequncia de aminocidos diferente da original a partir do local onde se deu a alterao.
| Captulo 12 Mutaes no DNA
171
Biologia Molecular - 1 Ano - FFUP
2006/2007
5. Anomalias cromossmicas
As anomalias cromossmicas envolvem alteraes em segmentos de DNA de grandes dimenses. Presumivelmente, estas anomalias devem-se maioritariamente a erros em mecanismos de reparao das quebras nas duplas cadeias de DNA. Os cromossomas (I ou II) esto exibidos como linhas nicas com regies envolvidas numa anomalia particular identificada por cor verde ou roxo. Mutao Deleco Esquema representativo Caracterizao Perda de material cromossmico originando falta de genes no centro ou na extremidade do cromossoma.
172
Captulo 12 Mutaes no DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
Translocao
Transferncia de material de um cromossoma para outro no homlogo translocao simples
Troca de segmentos entre 2 cromossomas no homlogos translocao recproca (mais frequentes).
Duplicao
Adio de um segmento cromossmico resultante do cromossoma homlogo. Desta forma, um conjunto de genes surge em duplicado.
Inverso
Inverso da ordem dos genes resultante da soldadura em posio invertida de um segmento cromossmico.
5.1. Perda de especificidade de emparelhamento
No caso das anomalias cromossmicas, integradas nas mutaes cromossmicas estruturais, tambm pode ocorrer perda sucessiva da especificadade de emparelhamento durante o desenvolvimento da mutao. Geralmente, os dois principais factores intervenientes nesta perda so a luz UV e a xerodermia. Luz UV leva formao de dmeros de timina, de 64-fotoprodutos (timina e citosina) e pode causar transies e transverses (em menor quantidade). Xerodermia conduz ao cancro da pele atravs da formao de dmeros de timina.
| Captulo 12 Mutaes no DNA
173
Biologia Molecular - 1 Ano - FFUP
2006/2007
6. Identificao de mutagnios
A identificao de mutagnios baseia-se na capacidade que um determinado compostos tem de reagir com algo para formar revertentes, isto , bactrias que conseguem voltar ao seu estado selvagem. Desta forma, se tivermos uma bactria que no tenha capacidade de sintetizar, quando ela sofre a aco de um mutagnio, as alteraes que ele induz podem ser responsveis pelo regresso ao estado selvagem e original da bactria. H muitos compostos que no so, partida, mutagnicos, passando apenas a esse estado depois do processo de metabolizao. Estes so compostos que inicialmente no tm relao com o DNA, mas que passam a ter depois de possurem grupos metoxi aps a metabolizao. O principal mtodo utilizado o teste de Ames. Este teste um teste de visualizao que utilizado para ajudar a identificar elementos qumicos que afectam a estrutura da molcula de DNA, avaliando o poder mutagnico de compostos carcinognicos por deteco de revertentes, atravs da passagem do estado mutante ao estado selvagem. Apesar disto, muitos dos agentes carcinognicos s passam a mutagnicos depois de terem sido activados pela metabolizao heptica, sendo os exemplos mais comuns de agentes mutagnicos, vrios elementos oxidantes (tabaco, dicloreto de etilo), anlogos de bases, agentes intercalantes e agentes alquilantes. As alteraes verificadas ao longo do teste resultam de mutaes que ocorrem quando a estrutura do DNA alterada em certos lugares, pois vrios elementos qumicos que provocam mutaes causam cancro em animais ou pessoas. Quando o teste foi criado, pensava-se que a maior parte dos elementos qumicos utilizados que produziram resultados no teste podiam tambm induzir a formao de um cancro.
6.1. Teste de Ames com bactrias
No teste de Ames com bactrias, utilizam-se estirpes de Salmonella auxotrfica que foram alteradas para se tornarem mais susceptveis actuao de mutaes do que a Salmonella normal. Para realizar o teste, as estirpes de Salmonella alteradas so combinadas num tubo-teste com o elemento qumico de interesse para a experincia. Como a Salmonella no possui as enzimas que os animais utilizam para metabolizar os elementos qumicos, adicionamse enzimas de fgado ainal ao tubo-teste para poder decorrer a experincia. Desta forma, possvel detectar o que aconteceria se o elemento qumico passasse para o interior de um organismo humano. As estirpes de Salmonella so depois transferidas para uma placa de Petri onde crescem durante cerca de dois dias. As estirpes alteradas usadas para o decorrer do teste requerem histidina proteica para poderem crescer e um resultado positivo neste teste indicado quando, em resposta mutao, a estirpe de Salmonella deixa de necessitar de histidina protieca para se desenvolver e crescer. 174 Captulo 12 Mutaes no DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
6.2. Teste de Ames com enzimas de ratinho
No caso da realizao do teste de Ames com enzimas de ratinho, o procedimento e o decorrer da experincia so diferentes do anterior. Aqui, em primeiro lugar, as enzimas do fgado de ratinho so mobilizadas atravs da injeco em animais com Arochlor. O fgado de ratinho , mais tarde, homogeneizado e o sobrenadante com enzimas solubilizadas (S9) adicionado a uma suspenso de bactria auxotrfica numa soluo de um potencial carcinognio (X). Esta mistura plaqueada num meio sem histidina e os resultados do mutante 1 e 2 so pesquisados simultaneamente, decorrendo um controlo experimental com o potencial carcinognico. Desta forma, a presena de revertentes indica que o elemento qumico um mutagnio e possivelmente um carcinognio ao mesmo tempo.
7. Outras mutaes
Para alm de todas as mutaes referidas at ao momento, existem ainda outras classes particulares de mutaes que podem ocorrer nas diferentes molculas de DNA, sendo que as principais so as seguintes: Mutaes por transposio so provocados por elementos de transposio, podendo causar deleces, inverses ou inseres.
| Captulo 12 Mutaes no DNA
175
Biologia Molecular - 1 Ano - FFUP
2006/2007
Mutaes reversveis so aquelas em que a base alterada reposta pela original, incluindo as mutaes pontuais, as inseres que so reversveis e as deleces que so irreversveis. Mutaes ribossomais substituo de aminocidos que distorcem o ribossoma (subunidade 30S) induzindo a entrada de uma molcula de aminoacil-tRNA errada. Mutaes supressoras as intragnicas incluem a mutao no mesmo gene, mas noutro local e as extragnicas contm tRNA mutante, corrigindo mutaes nonsense, missense e frameshift.
176
Captulo 12 Mutaes no DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
Captulo 13 Reparao do DNA
Apesar de a variao gentica ser importante para a evoluo, a sobrevivncia do indivduo requer estabilidade gentica. A manuteno da estabilidade gentica necessita no s de um mecanismo extremamente preciso para replicar o DNA, mas tambm de mecanismos para reparar as vrias leses acidentais que ocorrem continuamente no DNA. A maior parte da alteraes espontneas temporria, porque imediatamente corrigida por um conjunto de processos a que se chama reparao do DNA.
1. Agentes danificantes
2. Consequncias
As principais consequncias incluem a inibio dos seguintes processos: Transcrio Replicao Segregao de cromossomas apoptose Mutaes Aberraes cromossmicas cancro, envelhecimento, etc.
| Captulo 13 Reparao do DNA
177
Biologia Molecular - 1 Ano - FFUP
2006/2007
3. Reparao
As clulas possuem sistemas de reparao enzimticos, que tm por objectivo a eliminao de mutaes. A falncia destes sistemas est relacionada com a manuteno das mutaes e o desenvolvimento de doenas humanas. O nmero de reparaes dirias por clula humana encontra-se dentro dos seguintes valores: 2000 a 10000 depurinaes 100 a 500 desaminaes 500 a 600 metilaes 100 a 500 oxidaes
4. Classificao
Preveno de erros: Destoxificao Remoo directa de leses: Alquil transferases Fotoliase (fotorreactivao) Reparao por exciso: Geral uvrABCD especfica AP endonucleases DNA glicosilases Sistema GO Reparao ps-replicao: Por emparelhamento incorrecto Por recombinao Sistema SOS Sntese de transleso do DNA Reparao associada transcrio (TCR) Reparao de bases (BER) e nucletidos (NER) Reparao genmica global (GGR) Reparao por juno de extremidades no homlogas (Non-Homologous End Joining) Reparao por tRNAs mutantes 178 Captulo 13 Reparao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
5. Preveno de erros
Superxido dismutase: previne a formao de leses oxidativas Catalase: previne ou elimina a formao de espcies reactivas de oxignio
6. Remoo directa de leses
6.1. Reparao da alquilao
O -metiltransferase Codificada pelo gene ada Transfere grupos metil e etil para a enzima e reverte a mutao Tem de ser sintetizada novamente para remover outros grupos alquilantes
6
| Captulo 13 Reparao do DNA
179
Biologia Molecular - 1 Ano - FFUP
2006/2007
6.2. Fotorreactivao
Fotoliase ou PRE (photoreactivating enzime) gene phr Liga-se ao dmero da timina e na presena da luz do visvel (320-360 nm) usa a energia absorvida para quebrar a estrutura dimrica das duas timinas Existe nas bactrias e em alguns eucariotas inferiores, mas no no homem Na ausncia de luz visvel, esta mutao pode ser corrigida por outros mecanismos de reparao (exciso, sntese por transleso, ps replicao) Certas plantas e a Drosophila usam a fotoliase para reverter o 6-4-produto.
7. Reparao por exciso
7.1. Sistema geral - uvrABCD
Este sistema geral corrige leses que causam distoro na dupla hlice do DNA (fotoprodutos, UV e adio de qumicos) 1 uvrA reconhece a leso e liga-se com uvrB 2 uvrC liga-se e corta o DNA, depois de uvrA ser libertado
180
Captulo 13 Reparao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
3 uvrD desenrola o DNA e liberta o DNA lesado 4 DNA polimerase I sintetiza a cadeia 5 DNA ligase junta o DNA sintetizado
Basicamente, este mecanismo consiste em remover as bases que esto erradas, sendo que no passa pelo tirar das duas bases erradas sozinhas, mas sim um fragmento de DNA muito maior. Se existe um dmero de timina, h enzimas que cortam sequencia de DNA a volta de 20 nucletidos, deixam um espao vazio e como se forma uma cadeia sem nucletidos, intervm a DNA polimerase e a ligase que faz a ligao entre os nucletidos.
7.2. Sistema especfico (BER Base Excision Repair)
O sistema de reparao de bases envolve a remoo da base mutada da hlice de DNA e a reparao da base sozinha. DNA glicosilase Remove a base, cria locais AP (apurnicos sem A ou G; ou apirimidnicos sem C ou T), que depois so corrigidos pela AP endonuclease. Repara bases desaminadas (uracilo e hipoxantina), bases metiladas e oxidadas.
1 A glicosilase retira a timina da hlice, deixando apenas a desoxirribose (ponto preto). 2 Uma endonuclease especfica para o local sem base (AP endonuclease) corta as ligaes fosfodister junto ao local AP. 3 - O fosfato e desoxirribose so removidos por uma endonuclease associada com a DNA polimerase . 4 O local preenchido pela DNA Pol e selado pela DNA ligase.
| Captulo 13 Reparao do DNA
181
Biologia Molecular - 1 Ano - FFUP
2006/2007
Sistema GO Corrige mutaes provocadas pela leso oxidativa mutT hidrolisa 8-oxodGTP a 8-oxodGDP mutY (adenina glicosilase) remove A-GO e reconhece G-A. No fundo uma glicosilase que vai cortar DNA que vai reconhecer este tipo de emparelhamento que no corrido, A com GO e G com A que tambm no correcto. mutM responsvel pela remoo de DNA com leses com emparelhamento C-GO.
Este processo, funciona com uma base que foi oxidada, que passa a 8-oxodGTP, havendo um emparelhamento que no correcto nem normal; ou o mutM e tem capacidade de remover o GO e deixar um vazio e a celula vai utilizar outros sistemas de reparao para colocar o nucleotido em falta e o sistem fica em falta. Mas se ela no actua atempadamente, e aps a replicao, podemos ter na cadeia na mesma a presenaa de agentes oxidantes, ha emparelhamento de C com GO mas o GO tb pode emparelhar com o A. Logo, desta forma, a muTY vai actuar mas em vez de teretir o GO retira o A e ficamos na mesma com a falta de uma base naquele local. Ha sistemas de reparaao que tem la o C mas isto ainda nao ta correcot, logo vai entrar novamente a mutM para retir o GO e repor o GC que ja correcto.
182
Captulo 13 Reparao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
8. Reparao ps-replicao
8.1. Reparao de emparelhamento incorrecto
mutS e mutL interagem com o local G-T mutH e mutL interagem com os locais G-T e C-T mutY interage com os locais G-A mutH cliva a cadeia de DNA recm sintetizada mutU separa as duas cadeias (helicase) SSB estabilizam as cadeias de DNA separadas
1 O local de emparelhamento incorrecto reconhecido pela mutL e mutS. 2 A protena mutH corta a nova cadeia sintetizada. 3 As cadeias so separadas atravs da mutU e so estabilizadas pela SSB. 4 DNA polimerases sintetizam um novo fragmento para substituir o que sofreu exciso e esta metilada.
Nos mamferos:
mutS (Msh2/Msh6) reconhecem o emparelhamento incorrecto de 1 bp mutS (Msh2/Msh3) reconhecem 2-12 bp mutL (Mlh1/Pms2) funo exacta desconhecida PCNA necessrio para a maquinaria replicativa, interage com Msh3 e Msh6 RPA protege DNA de cadeias simples e previne a aco das exonucleases Exonuclease I identificada apensas nos eucariotas Pols / refazem a cadeia de DNA
| Captulo 13 Reparao do DNA
183
Biologia Molecular - 1 Ano - FFUP
2006/2007
8.2. Reparao por recombinao
RecBCD tambm conhecida como exonuclease V, uma protena que inicia a reparao por recombinao de falhas na cadeia dupla. composta por 3 subunidades diferentes, codificadas pelos genes recB, recC e recD. As subunidades RecB e RecD so helicases. RuvABC complexo de trs protenas que medeiam a migrao de cadeias e resolvem a juno Holliday (juno de 4 cadeias de DNA) criada durante a reparao por recombinao. RuvA e RuvB ligamse s quatro cadeias de DNA e fazem com que as cadeias migrem atravs de si mesmas, utilizando um mecanismo rotativo. A ligao do RuvC ao complexo RuvAB faz com que as cadeias se desliguem. Na reparao por recombinao, a reparao passa frente de uma leso bloqueante, deixando um espao na nova cadeia. Uma protena recA-directed preenche o espao, utilizando uma parte da cadeia parental oposta. Finalmente, a protena recA repara o espao na cadeia parental.
8.3. Sistema SOS
um sistema indutvel. Protena recA liga-se ao DNA de cadeia simples e activa o sistema SOS por clivagem proteoltica do repressor lexA. DNA reparado por recombinao, exciso, sntese bypass, alquilao ou sistema GO, cujos genes esto sob controlo de lexA Sistema indutvel. Permite sobrevivncia em situaes de stress. umuC e umuD (UV no mutados) requerem a protena recA e codificam para protenas que, associadas DNA polimerase III, introduzem bases mesmo na ausncia de um molde.
184
Captulo 13 Reparao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
No bypass por SOS, quando a replicao atinge um local que contm uma leso bloqueante, o sistema SOS insere o nmero necessrio de bases (normalmente incorrectas) directamente da leso e a replicao continua. de realar que a leso ainda se mantm e deve ser reparada atravs de outro mecanismo.
9. Sntese de transleso do DNA
Pol eta () Insere adenosinas no local oposto a dmeros TT Baixa fidelidade Baixa processividade Pode dar origem a erros noutro tipo de leses produto do gene XPV
Pol zeta () e Rev I Rev I insere bases ao acaso no local oposto a TT Pol zeta () faz a extenso de vrias bases Baixa processividade Baixa fidelidade
| Captulo 13 Reparao do DNA
185
Biologia Molecular - 1 Ano - FFUP
2006/2007
10. Reparao associada transcrio
Reparao da cadeia codificante durante a transcrio da RNA polimerase II No repara 6-4-fotoprodutos Requer CSA e CSB Requer todos os factores NER com excepo de XPC
11. Reparao de nucletidos (NER Nucleotide Excision Repair)
Factores envolvidos na exciso XPC-HHR23B XPA RPA TFIIH XPB XPD XPG ERCC1-XPF
Factores necessrios para a sntese de reparao Factor de replicao C (RFC) PCNA DNA Pol e DNA ligase I
Factores adicionais para a reparao ligada transcrio CSA CSB
Enquanto que a maquinaria do sistema de reparao de bases consegue reconhecer leses especficas no DNA e corrigir apenas as bases danificadas, as enzimas do sistema de reparao de nucletidos reconhecem distores volumosas na forma da dupla hlice de DNA. O reconhecimento destas distores leva remoo de um curto segmento da cadeia que inclui a leso, criando uma falha numa das cadeias, que posteriormente preenchido pela DNA polimerase, que usa a cadeia no danificada como molde. NER pode ser dividido em duas vias: Global genomic NER (GGR) e Transcription coupled NER (TCR), que diferem apenas no reconhecimento da distoro.
186
Captulo 13 Reparao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
11.1. Reparao genmica global (GGR)
XPC reconhece o dano XPG 3 corte (juno de endonucleases especficas) XPA e RPA confirmam o corte e estabilizam o complexo RPA liga a ssDNA
A GGR repara tanto cadeias de DNA transcritas como no-transcritas em genes activos e inactivos ao longo do genoma. Esta via implica vrias protenas, incluindo os complexos DDB (DNA-damage binding) e XPC-Rad23B, que esto constantemente a verificar o genoma e a reconhecer distores na hlice. Aquando da identificao de um local danificado, as protenas reparadores subsequentes so recrutadas para verificar a presena de um erro, efectuar a exciso da regio envolvente da leso e voltar a preencher a cadeia.
11.2. Reparao ligada transcrio nas vias do BER e do NER (TCR)
CSA e CSB funo no processamento da RNAP II ERCC1-XPF 5 corte
Quando a Rna Pol II est a sintetizar a molcula de RNA e depois encontra algo que ela no conhece e no consegue sintetizar a cadeia complementar porque o sistema muito mais complexo e a transcrio pra. Esta activa ento as enzimas todas que esto relacionadas com o processo de reparao.
| Captulo 13 Reparao do DNA
187
Biologia Molecular - 1 Ano - FFUP
2006/2007
Isto tem duas consequncias importantes: a RNA polimerase torna-se uma ferramenta de reconhecimento de DNA danificado e d-se mais ateno aos genes que esto a ser transcritos. Este mecanismo especialmente importante, j que a paragem persistente da DNA Pol II resulta na paragem do ciclo celular ou na morte celular programada (apoptose). Envolve as MSH, BRCA1 que est envolvido no desenvolvimento do cancro da mama, e envolve ainda outras protenas que so as CSB1 e 2 e as enzimas do sistema XP.
12. Reparao por juno de extremidades no homlogas
188
Captulo 13 Reparao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
13. Reparao por tRNAs mutantes
13.1. Supresso de mutao nonsense por tRNAs mutantes
| Captulo 13 Reparao do DNA
189
Biologia Molecular - 1 Ano - FFUP
2006/2007
13.2. Supresso de mutao missense por tRNAs mutantes
13.3. Supresso de mutao frameshift por tRNAs mutantes
tRNA supressor tem a capacidade de passar uma base (frameshift + 1) ou voltar atrs (frameshift 1) O ribossoma est atrasado em relao frameshift e permite o correcto emparelhamento do tRNA
14. Sndromas humanos associados a defeitos na reparao do DNA
Sndrome de Werner Nmero elevado de deleces cromossomais Atraso no crescimento Progeria Defeitos na reparao por recombinao e na sntese de transleso Protena WRN: DNA helicase da famlia RecQ
190
Captulo 13 Reparao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
Sndrome de Cockayne Defeitos no sistema de reparao associado transcrio (TCR) por alteraes causadas pela radiao UV Atraso mental Envelhecimento precoce Nanismo
Sndrome de Bloom Doentes apresentam um maior nmero de rearranjos no DNA e deleces no DNA Provoca atrasos no crescimento e veias dilatadas (telangiectasias) na pele e em particular na face Doentes tm maior probabilidade de desenvolverem cancro antes do 30 anos Defeitos na recombinao homloga
Ataxia telangiectasia Causada por mutaes ATM H aumento na sensibilidade a radiaes ionizantes H rearranjos cromossmicos e de linfomas
| Captulo 13 Reparao do DNA
191
Biologia Molecular - 1 Ano - FFUP
2006/2007
15. Mutaes, consequncias e reparao
C-G substituda por U-G Corrigida removendo U Par de purinas distorce o duplex corrigida removendo uma das purinas Falta de uma purina corrigida por insero Distoro da dupla hlice devido a um grupo metilo corrigida por alquil transferases Dmero de timinas distorce o duplex Corrigida por exciso
16. Enzimas e protenas dos sistemas de reparao de E. coli
192
Captulo 13 Reparao do DNA |
Biologia Molecular 1 Ano - FFUP
2006/2007
Captulo 14 Tcnicas de DNA recombinante
A recombinao entre molculas de DNA de diferentes organismos um fenmeno comum na natureza. As tcnicas de DNA recombinante permitem estudar processos biolgicos e obter benefcios em vrias reas, nomeadamente na sade, biologia, bioqumica, microbiologia, gentica, biotecnologia, agricultura, etc. estas tcnicas possibilitam a clonagem, identificao e regulao de genes; o estudo de doenas hereditrias e terapia genica; produo de protenas, drogas e vacinas; melhoramento de culturas e animais; estudo da gentica molecular e da gentica reversa. A engenharia gentica consiste na criao de uma nova molcula de DNA, geralmente pela recombinao de DNA de diferentes organismos, utilizando-se enzimas conhecidas como enzimas de restrio, e a consequente produo de muitas cpias de DNA recombinante. Este processo designa-se por clonagem. A amplificao de um gene ou genes especficos clonados, associada ao considervel aumento da produo dos seus produtos proteicos, torna relativamente fcil extrair e purificar as respectivas protenas no laboratrio.
Experincia tpica de clonagem. Selecciona-se um plasmdeo adequado (vector) para a insero de um gene determinado (DNA dador). Tanto o DNA dador como o vector tm sequncias de DNA especficas (4 - 6 pares de bases) nas quais endonucleases de restrio cortam as cadeias do DNA. Em tubos separados, mistura-se por um lado o fragmento de DNA contendo o gene a ser clonado e por outro o plasmdeo no qual o gene ser inserido, com a endonuclease de restrio escolhida (neste caso, EcoRl). A enzima escolhida dever cortar o plasmdeo num s local. O plasmdeo cortado pode ento receber uma poro adicional de DNA. Incuba-se os fragmentos de DNA e os plasmdeos cortados e abertos, com a enzima ligase que liga fragmentos de DNA e plasmdeos. Desta ligao resulta um plasmdeo recombinante que contm o fragmento de DNA desejado. As clulas que adquiriram os plasmdeos dizem-se transformadas e, neste caso, so resistentes ao antibitico amplicilina.
| Captulo 14 Tcnicas de DNA recombinante
193
Biologia Molecular - 1 Ano - FFUP
2006/2007
1. Gentica reversa vs. Gentica molecular
Gentica molecular - estudo da funo genica no controle de actividades celulares e da sua organizao fsica dentro dos genomas. Gentica reversa - na gentica directa o mapeamento genico realizado a partir do fentipo. Na gentica reversa, a associao entre genes e fentipo estabelecida pela manipulao do gene e observao de alteraes ocasionadas no fentipo. A clonagem de genes a partir de marcao com transposes um exemplo de gentica reversa. Neste caso, quando um transposo se integra no gene, a sua expresso resulta num fentipo alterado ou mutante, permitindo o isolamento do gene.
194
Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
Alguns benefcios da biotecnologia Hibridoma Anticorpos
Protenas
Insulina lnterfero Albumina srica humana Hormona humana do crescimento Activador plasminognico de tecidos Antitrombina Factores de coagulao do sangue Luciferase (pirilampo) Linfocinas Factor da necrose tumoral Gonadotropina humana
Agricultura
Cereais resistentes a doenas Tomates hidropnicos Resistncia a pesticidas Bio-insecticidas Fixao do azoto
Vacinas
Hepatite B Herpes Gripe Malria
Clonar um processo em que se insere um fragmento de DNA num plasmdeo ou no cromossoma de um fago sendo-lhe permitido replicar-se para produzir numerosas cpias do DNA. A replicao tem normalmente lugar quando o plasmdeo ou cromossoma fgico se introduzem num hospedeiro apropriado (ex: uma bactria ou uma clula de levedura) e o aparelho de sntese do DNA do hospedeiro replica o DNA inserido na clula hospedeira.
| Captulo 14 Tcnicas de DNA recombinante
195
Biologia Molecular - 1 Ano - FFUP
2006/2007
A finalidade de qualquer experincia de clonagem isolar um determinado gene ou fragmento de DNA de um organismo e introduzi-lo numa clula hospedeira apropriada para obter grandes quantidades do DNA. O DNA a clonar designa-se por DNA dador. Muitas vezes, utiliza-se o DNA dador para a produo em larga escala de protenas importantes (Quadro I). Contudo, este tambm se pode usar na deteco de agentes infecciosos ou clulas anormais. Normalmente, o DNA dador corresponde a uma pequena poro do genoma de uma clula e encontra-se representado por uma ou duas cpias por clula. Logo, antes de se poder extrair o DNA dador tem de se obter, a partir quer de uma pequena poro de tecido, quer de uma cultura de clulas, um nmero suficiente de clulas que contenha DNA desejado. Depois de se ter obtido um nmero suficiente de clulas contendo o material gentico extrado. Este material gentico pode estar presente nos cromossomas ou em plasmdeos.
2. Extraco de DNA
2.1.
Extraco do DNA cromossmico das clulas
As molculas de DNA que formam os cromossomas de uma clula correspondem ao DNA genmico da clula. De modo a libertar o DNA genmico duma clula, tm de se dissolver os invlucros membranares que englobam e protegem o material gentico, e digerir as histonas e outras protenas cromossmicas. As membranas dissolvem-se usando detergentes que solubilizam os fosfolpidos que as constituem. Os detergentes mais comuns na extraco do DNA genmico, so o dodecil sulfato de sdio (SDS), o Triton100 e o Tween. Uma vez dissolvidas as membranas, trata-se o lisado celular resultante (constitudo por material citoplasmtico e nuclear, organitos e cromossomas) com proteases (ex. proteinase K) que digerem as protenas e, como tal, dissociam-nas com solventes orgnicos, como seja uma mistura de fenol e clorofrmio, para remover as protenas e outros detritos celulares, ficando o DNA genmico na fase aquosa. A recolha deste DNA parcialmente purificado o prximo e ltimo passo. Quando se adiciona etanol absoluto soluo aquosa de DNA, desenvolve-se uma interfase entre os dois lquidos devido s suas diferentes densidades. O DNA genmico na fase aquosa precipitar na interfase medida que filamentos finos e brancos se aglomeram numa massa com aspecto opaco. Com a ajuda de uma vareta de vidro, o DNA genmico pode ser retirado da interfase por enrolamento (spooling). Para tal, introduz-se uma vareta de vidro na interfase e roda-se de modo a que as cadeias de DNA se enrolem a esta. O DNA enrolado pode ser seco, redissolvido em soluo aquosa e usado numa variedade de processos.
2.2. Extraco do DNA plasmdico das bactrias
O genoma de uma clula bacteriana consiste numa nica molcula de DNA circular (cromossoma). Os plasmdeos so pequenas molculas de DNA auto-replicativas que podem existir independentemente do cromossoma bacteriano. Os plasmdeos possuem a informao gentica que dirige a sua prpria replicao e alguns dos seus genes, conferem algumas vantagens selectivas clula. Exemplos destes genes incluem a 196 Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
resistncia a metais pesados e antibiticos. Visto muitas clulas bacterianas possurem simultaneamente o cromossoma bacteriano e vrios plasmdeos, o isolamento do DNA plasmdico exige um passo no qual este ter de ser separado do DNA cromossmico bacteriano (genmico). Tal como nas clulas eucariticas, as clulas bacterianas so tratadas com detergentes de modo a romper a membrana plasmtica. Como a bactria possui uma parede celular, tambm necessrio o tratamento com a enzima lisozima para remover este invlucro externo. Este tratamento, juntamente com o dos detergentes, liberta ambos os DNAs, plasmdico e cromossmico, da clula. Para o isolamento selectivo do DNA plasmdico das clulas, adiciona-se hidrxido de sdio (NaOH) soluo detergente para desnaturar o DNA (i.e., fazer com que as duas cadeias se separem). A desnaturao do DNA bacteriano com NaOH e detergentes designa-se por lise alcalina. O DNA cromossmico e muitas protenas e componentes da membrana, so removidos pela adio de potssio (geralmente sob a forma de acetato de potssio) a esta mistura, seguindo-se de uma centrifugao. A precipitao selectiva do DNA cromossmico baseia-se nas maiores dimenses do DNA genmico; a maior dimenso torna-o mais vulnervel precipitao pelo acetato de potssio do que a pequena molcula de DNA plasmdico. Depois deste passo, o DNA plasmdico mantm-se em soluo juntamente com o RNA celular. Esta soluo tambm conhecida como lisado lmpido. O RNA pode ser eliminado pelo tratamento com a enzima RNase, que por sua vez pode ser removida com solventes orgnicos (fenol-clorofrmio). Precipita-se ento o DNA plasmdico com etanol, seca-se e redissolve-se em soluo aquosa para ser utilizado numa variedade de aplicaes.
3. Enzimas de restrio
Endonucleases de restrio so enzimas bacterianas que reconhecem sequncias nucleotdicas especficas da molcula de DNA em cadeia dupla e cortam o DNA nesses locais. Estas enzimas cortam o DNA em fragmentos de vrios comprimentos, dependendo do nmero de vezes que o local de reconhecimento da respectiva enzima se repete ao longo da molcula. As endonucleases de tipo II, que se utilizam em experincias de clonagem, reconhecem sequncias de pares de bases de quatro a oito nucletidos e cortam dentro destas sequncias. Por exemplo, a endonuclease de restrio conhecida como SmaI reconhece a sequncia 5-CCCGGG-3 e corta entre o C adjacente ao G, enquanto que a enzima EcoI reconhece a sequncia 5-GAATTC-3 e corta entre o G e o A. Estes locais designam-se por sequncias de reconhecimento. Consequentemente, qualquer molcula de DNA que seja cortada (ou restringida) por uma endonuclease de restrio de tipo II tem de ter sequncias de reconhecimento conhecidas como palndromas. Palndromas so sequncias de quatro a seis bases (nucletidos) que so idnticas em ambas as cadeias da molcula de DNA quando lidas na mesma direco (5-3). As endonucleases de restrio cortam o DNA gerando fragmentos com extremidades coesivas ou extremidades cegas.
| Captulo 14 Tcnicas de DNA recombinante
197
Biologia Molecular - 1 Ano - FFUP
2006/2007
As endonucleases de restrio so denominadas segundo a espcie bacteriana de onde foram isoladas. Por exemplo, a endonuclease de restrio EcoRI obtm-se a partir da estirpe R da bactria E.coli, e a enzima HindIII a partir da estirpe D da bactria Haemophilus influenzae. A numerao romana indica a ordem pela qual determinada enzima foi isolada do microrganismo. Assim, EcoRI foi a primeira enzima de restrio a ser isolada da estirpe R de E.coli enquanto que HindIII foi a terceira enzima de restrio isolada da estirpe D de Haemophilus influenzae. Diferentes microrganismos produzem endonucleases de restrio que reconhecem a mesma sequncia de reconhecimento, o que significa que duas enzimas de restrio diferentes podem cortar no mesmo local de reconhecimento. Estas designam-se por isosquizmeros, ex: MspI e HpaII.
Enzimas de modificao so aquelas que reconhecem a mesma sequncia de DNA das correspondentes enzimas de restrio e, em vez de a cortarem, modificam-na.
198
Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
4. Mapas de restrio
Tendo em considerao que as endonuclease cortam o DNA em locais especficos, uma dada molcula de DNA de um organismo pode diferenciar-se de uma outra molcula de DNA pelo nmero de vezes que uma dada enzima de restrio corta o DNA. Por exemplo, a enzima de restrio HindIII corta o DNA do bacterifago em sete locais diferentes ao longo do genoma viral, gerando oito fragmentos. Por outro lado, o plasmdio pBR322 s tem um local de restrio para a sequncia de reconhecimento HindIII. Quando a enzima corta este plasmdeo, converte a molcula de DNA circular em linear com parte do local de reconhecimento HindIII em cada extremidade da molcula. A representao grfica dos locais de reconhecimento para duas ou mais endonucleases de restrio corresponde ao mapa de restrio (endonuclease) para essa molcula.
| Captulo 14 Tcnicas de DNA recombinante
199
Biologia Molecular - 1 Ano - FFUP
2006/2007
200
Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
5. Vectores de clonagem
Um vector uma molcula de DNA qual DNA estranho pode ser ligado e posteriormente inserido nas clulas de modo a que o DNA recombinante se possa replicar. Os vectores podem ser introduzidos nas clulas hospedeiras por transformao ou por infeco fgica. Existem vrios tipos de vectores utilizados nas experincias de clonagem. Contudo, os mais frequentes so os plasmdeos e vrus bacterianos (bacterifagos) porque facilmente se introduzem nas clulas e so manipulados no laboratrio. Independentemente do tipo, os vectores tm de ter certas caractersticas adequadas clonagem.
Caractersticas dos vectores de clonagem Caractersticas Serem estveis na clula hospedeira Controlarem a sua prpria replicao Funes Permite a replicao Permite a sua multiplicao dentro da clula com um elevado nmero de cpias Permite uma rpida introduo na clula por transformao, electroporao ou transduo
Possurem pequenas dimenses
Serem cortados num s local por uma enzima Permite a insero do DNA dador e a circularizao do plasmdeo de restrio No serem transferidos por conjugao Evita que o DNA recombinante se dissemine para as populaes naturais de bactrias Torna possvel distinguir as clulas transformadas de no transformadas Aumenta o rendimento de plasmdeos recombinantes
Terem caractersticas facilmente detectveis Serem facilmente isolados das clulas
Um dos principais objectivos da clonagem produzir grandes quantidades de um determinado gene ou do seu produto. A produo da protena pretendida possvel se os vectores de clonagem tiverem locais de regulao para a iniciao da transcrio do gene e traduo do seu mRNA pela clula hospedeira.
| Captulo 14 Tcnicas de DNA recombinante
201
Biologia Molecular - 1 Ano - FFUP
2006/2007
202
Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
As molculas de DNA dador e vector ligam-se atravs de enzimas chamadas ligases de DNA ou ligases de polinucletido. As ligases de DNA so enzimas que catalisam a formao de ligaes fosfodister entre o grupo 3-hidroxilo de um segmento de DNA dador e o grupo 5-fosfato do DNA vector.
| Captulo 14 Tcnicas de DNA recombinante
203
Biologia Molecular - 1 Ano - FFUP
2006/2007
6. Cromossomas artificiais
6.1. Cromossomas artificiais de bactrias (BAC)
Um cromossoma artificial de bactrias (BAC) uma poro de DNA artificial que vai ser transplantado para um tecido alvo ou clula (DNA construct), baseado numa plasmdeo de fertilidade (F-plasmdeo), usado para transformar e clonar em bactrias, geralmente E.coli. Os F-plasmdeos desempenham um papel importante porque contm genes que promovem a distribuio uniforme dos plasmdeos aps a diviso celular da bactria, sendo por isso um vector de clonagem.
6.2. Cromossomas artificiais de leveduras (YAC)
Um cromossoma artificial de leveduras (YAC) um vector de clonagem de grandes fragmentos de DNA (maior de 100 kb e at 3000 kb). uma poro de DNA construdo artificialmente e contm as sequncias telomricas, centromricas e de origem de replicao necessrias replicao e conservao nas clulas das leveduras. 204 Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
7. Vectores de expresso
Os vectores de expresso so vectores que possuem um gene que eficazmente transcrito e traduzido pela clula hospedeira. Para a transcrio, so necessrios um local promotor (P)e um local terminador (TER). A transcrio do gene pretendido comea no local promotor e termina no local terminador. Em muitos vectores de expresso, tambm se encontra presente, a montante do codo de iniciao, um local de ligao ao ribossoma (RBS). Este local necessrio para uma eficiente iniciao da traduo nas bactrias. Os vectores de expresso controlada so muito importantes em Engenharia Gentica,, uma vez que do resposta necessidade de superproduzir, de uma forma controlada, o produto de expresso de um gene (uma protena recombinada - r-protena). Estes vectores alm de terem uma origem de replicao e marcas genticas de seleco, possuem outras caractersticas lhe conferem a especializao. No caso do
| Captulo 14 Tcnicas de DNA recombinante
205
Biologia Molecular - 1 Ano - FFUP
2006/2007
vector de expresso controlada para expresso numa clula bacteriana (usualmente E. coli) devero apresentar: 1. Uma regio promotora reconhecida no hospedeiro e que permita uma eficiente transcrio do gene clonado a usar para superproduo na r-protena e . conveniente que este promotor seja regulvel de modo a que possa ser ligado ou desligado, da o nome de vectores de expresso controlada. 2. O codo de iniciao ATG que inicia a transcrio da maioria dos genes dos procariotas; 3. Um terminador da transcrio a jusante da sequncia codificante para evitar a formao de mRNA demasiado longo; 4. Uma regio, designada Shine-Delgarno, que garanta uma ligao efectiva do mRNA ao ribossoma onde se d a traduo, garantindo que o processo se d eficientemente e que a expresso do gene clonado no fica limitada ao nvel da traduo; 5. Um ou mais codes stop para a terminao da traduo. Para alm destas caractersticas, os plasmdeos especializados para superexpresso podero ser em elevado nmero de cpias de modo a maximizar o nmero de cpias do gene clonado na clula. No entanto, se no houver controlo de expresso tal que poder levar a uma afectao grave da fisiologia do hospedeiro o que resultar num efeito oposto ao desejado.
206
Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
A competncia natural das bactrias para receber o DNA do meio circundante um fenmeno raro (verificando-se apenas em determinadas espcies e em condies fisiolgicas especficas). Assim, necessrio desenvolver essa capacidade, ou seja, necessrio tornar as clulas competentes para viabilizar a introduo do DNA recombinado (rDNA) no hospedeiro seleccionado. Ao processo de entrada de DNA livre (isto , DNA em soluo, fora do invlucro celular) na clula d-se o nome de transformao. A introduo de rDNA em clulas bacterianas (como por exemplo na estirpe Escherichia coli XLIBlue) pode ser realizada recorrendo s tcnicas de transformao clssica ou electroporao. Na transformao qumica (ou clssica) as clulas (principalmente de E. coli) so tornadas competentes, atravs dum mecanismo ainda por esclarecer, por incubao numa soluo fria de cloreto de clcio, a que se segue um choque trmico de curta durao. Se necessrio possvel aumentar a eficincia do processo por recurso a outros ies monovalentes (K+ ou Rb+) ou divalentes (Mg2+ ou Mn 2+). A electroporao ou electrotransformao, como alternativa transformao clssica, assenta na electropermeabilizao, reversvel, das clulas bacterianas, por sujeio a um pulso elctrico de elevada intensidade e curta durao. A alta voltagem altera a estrutura da membrana, formando-se poros temporrios que permitem a entrada do plasmdeo na clula. Esta tcnica, que permite uma maior eficincia face transformao qumica, garante tambm a introduo de DNA recombinado (rDNA) em espcies refractrias transformao clssica, desde que as condies experimentais sejam devidamente optimizadas. A conjugao um processo natural de transferncia de material gentico entre bactrias, que pressupe o contacto fsico entre estas. A clula dadora a clula que possui o rDNA, que queremos transferir para uma outra clula, dita receptora. Neste caso, o vector de clonagem a usar tem de poder ser mobilizvel, ou seja, apresentar parte das funes de um vector conjugativo, que autotransmissvel. Por questes de segurana em Engenharia Gentica, no permitido o uso de vectores baseados em plasmdeos conjugativos, de modo a no viabilizar a sua transferncia descontrolada para outras clulas bacterianas fora do laboratrio e do controlo do manipulador. As funes que so retiradas aos plasmdeos conjugativos para os tornar no conjugativos tero de se encontrar presentes, durante a transferncia por conjugao em laboratrio, num plasmdeo ajudante, que pela sua co-presena, permitir a associao entre a clula receptora e a clula dadora atravs do tubo de conjugao (ver figura). Este processo, envolvendo trs clulas (a dadora com o rDNA, a receptora e a ajudante com o plasmdeo ajudante que
| Captulo 14 Tcnicas de DNA recombinante
207
Biologia Molecular - 1 Ano - FFUP
2006/2007
apresenta as funes em falta no vector com o rDNA). De salientar que a clula receptora no apresenta o rDNA mas que, no final de um processo bem sucedido, ambas as clulas de conjugao apresentam o rDNA, podendo assim a clula receptora funcionar como dadora num novo processo de conjugao. A transduo recorre tambm a um fenmeno natural: a infeco viral em bactrias. Assim, este processo utiliza um bacterifago ou fago (vrus de bactrias) que serve de veculo para a introduo do rDNA, ao infectar a clula receptora, de uma forma controlada, no laboratrio. No caso da bactria E. coli, possvel utilizar o fago (fago desta espcie) num processo engenhoso que permite introduzir plasmdeos de grandes dimenses (cerca de 50 kb ou seja 50000 pares de bases). Para tal, estes plasmdeos tm que incluir uma regio dita cos, de reconhecimento pelo fago. Estes vectores de clonagem designam-se, por isso, cosmdeos. A transfeco consiste na infeco de uma clula hospedeira por DNA ou RNA viral.
208
Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
8. Bibilioteca de genes
Uma biblioteca de genes uma coleco de fragmentos de DNA de uma determinada espcie de organismo, obtidos a partir da aco de endonucleases de restrio, ligados aos vectores apropriados e clonados aps terem sido inseridos num hospedeiro. suposto que uma biblioteca seja representativa de todos os genes do organismo, sendo muito til quando se pretende isolar um gene especfico. Assim, tornase claro que quando se quer clonar um determinado gene ou o cDNA (DNA complementar) das mensagens por ele codificadas (mRNA) necessrio a construo de coleces de genes recombinantes. Se estes forem obtidos a partir do DNA genmico, sendo portadores de molculas representantes de todo o genoma, constituem uma biblioteca genmica; uma biblioteca de cDNA uma coleco de clones de DNA complementar derivados de toda a populao de mensageiros da clula ou tecido de interesse. Uma biblioteca de genes pode ser uma colnia de bactrias (se for construda utilizando um plasmdeo ou um cosmdeo) ou placas de fagos (se o vector utilizado for um fago) com DNA recombinante. Esta inclui sequncias de um conjunto muito grande, como o genoma inteiro de um organismo, sequncias expressas de um tipo celular particular ou sequncias presentes num fragmento de DNA.
Figura A1: Bibliotecas genmicas. Biblioteca de plasmdeos (a) e biblioteca de fagos (b)
Figura A2: Biblioteca de cDNA: de RNA ao cDNA.
| Captulo 14 Tcnicas de DNA recombinante
209
Biologia Molecular - 1 Ano - FFUP
2006/2007
Uma biblioteca genmica deve conter vrias cpias/clones representativos de cada fragmento de DNA para aumentar a probabilidade de se conseguir isolar genes de cpia nica, ou seja, a maior parte dos genes codificantes de protenas. A utilizao de uma biblioteca o modo mais eficiente para se conseguir isolar uma determinada poro de DNA. A separao da poro do DNA genmico que contem toda a unidade de transcrio com as regies limitadoras onde esto localizados os elementos controladores da expresso desse gene, permite-nos obter informaes acerca da estrutura molecular de um determinado gene de eucariotas. O ponto ao qual se d mais relevo na construo de uma biblioteca genmica a obteno aleatria de um elevado nmero de clones contendo fragmentos do genoma. Para garantir que um dado fragmento de interesse esteja entre os fragmentos clonados, necessrio estimar o tamanho necessrio da biblioteca atravs do tamanho do fragmento clonado e do tamanho do genoma. Assim, na construo destas bibliotecas o objectivo bsico procurar reduzir o nmero de clones necessrios, aumentando o mximo possvel o tamanho dos fragmentos de DNA clonados, e, utilizando vectores de clonagem baseados no fago , maximizar a eficincia da clonagem. A construo de uma biblioteca genmica envolve basicamente os seguintes passos: 1 - isolamento de DNA de alto peso molecular, que posteriormente quebrado de modo a produzir fragmentos de tamanho compatvel com o vector. 2 - ligao desses fragmentos ao vector e introduo dos recombinantes obtidos nas clulas hospedeiras.
8.1. Rastreio de uma bibilioteca de genes por hibridizao com sonda
A desnaturao do DNA pode ser provocada por aquecimento de uma soluo aquosa de DNA a 100C ou por exposio a pH elevado (pH13). Contudo, a desnaturao do DNA no irreversvel: se mantidas a 65C por um perodo prolongado de tempo, as cadeias complementares de DNA voltam a formar duplas hlices por via de um processo designado por hibridizao permitindo assim a renaturao do DNA. Similarmente, a hibridizao pode ocorrer entre quaisquer duas cadeias complementares de cidos nucleicos (DNA/DNA, RNA/RNA, DNA/RNA). 210 Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
Este mecanismo amplamente utilizado para detectar e caracterizar sequncias especficas de nucleotdeos quer no RNA quer no DNA. No caso particular das bibliotecas de genes, as sondas permitem descobrir, por exemplo, se uma determinada sequncia est presente ou mesmo caracterizar a sequncia de um dado clone. As molculas de DNA de cadeia simples usadas para detectar sequncias complementares designamse por sondas; estas apresentam radioactividade ou marcadores qumicos para facilitar a sua deteco e podem ter desde 15 at milhares de nucleotdeos. As reaces de hibridizao com sondas de DNA so extremamente selectivas e sensveis pois permitem detectar sequncias complementares presentes em concentraes extremamente baixas (at uma molcula por clula). As sondas so obtidas por polimerizao de nucleotdeos (marcados radioactivamente e detectveis por auto-radiografia; ou com um marcador qumico que pode ser detectado pelo uso de um anticorpo especfico) por aco de uma DNA polimerase. As bibliotecas genmicas e de cDNA podem conter mais de um milho de clones, no caso dos eucariotas. Procurar um clone especfico tem que obedecer a um mtodo preciso, caso contrrio seria praticamente impossvel encontr-lo. De forma geral existem duas formas para examinar (fazer o screening) uma biblioteca por forma a identificar os clones que transportam um determinado gene ou outra regio de DNA de interesse: 1) deteco com uma sonda de oligonucleotdeos que se ligue ao clone de interesse 2) deteco baseada na expresso da protena que o gene codifica. No ensaio de hibridizao esquematizado na figura F6.1, uma sonda de cadeia simples usada para detectar os fragmentos de DNA complementares sonda, presentes numa mistura. Inicialmente, a amostra de DNA desnaturada originando cadeias simples que se ligam a um suporte slido, geralmente filtro de nitrocelulose ou membrana de nylon. A membrana ento incubada com uma soluo contendo as sonda marcadas radioactivamente. Em condies apropriadas (pH aproximadamente neutro, 40-65C, 0.3-0.6 M NaCl) a sonda hibridiza com as cadeias complementares. As sondas em excesso, isto , que no hibridizem, so extradas e os hbridos so detectados por autoradiografia. Figura F6.1: Ensaio de hibridizao em membrana detecta complementaridade a uma sonda. Este ensaio pode ser usado para detectar DNA e RNA e a sonda marcada radioactivamente pode ser de DNA ou RNA.
| Captulo 14 Tcnicas de DNA recombinante
211
Biologia Molecular - 1 Ano - FFUP
2006/2007
A figura F6.2 consiste na aplicao deste procedimento ao caso duma biblioteca de cDNA. Neste caso, uma rplica da placa de Petri contendo os clones reproduzida na superfcie duma membrana de nitrocelulose. Usa-se ento uma sonda marcada radioactivamente e especfica para o DNA recombinante contendo o fragmento de interesse. A hibridizao com sondas radioactivas usada para examinar bibliotecas de cDNA; quando obtido um clone de cDNA que codifica uma dada protena, o cDNA pode ento ser marcado radioactivamente e usado como sonda dos clones de bibliotecas genmicas contendo fragmentos do gene correspondente.
Figura F6.2: Bibliotecas de cDNA podem ser examinadas com sondas radioactivas para identificao do clone de interesse. A identificao de clones especficos pela tcnica de hibridizao em membrana est contudo dependente da disponibilidade de sondas complementares marcadas radioactivamente. Para que um oligonucleotdeo seja til como sonda, deve ser suficientemente grande para que a sua sequncia ocorra unicamente no clone de interesse e em nenhum dos outros clones. Habitualmente esta condio satisfeita para oligonucleotdeos de cerca de 20 nucleotdeos. Estes oligonucleotdeos podem ser sintetizados quimicamente e posteriormente marcados radioactivamente usando a cnase dos polinucleotdeos, a qual transfere fsforo radioactivo do ATP para a extremidade 5 de cada nucleotdeo. Tendo em conta que, se a sequncia (ou parte dela) dos aminocidos da protena codificada pelo clone conhecida, ento pode construir-se uma sonda complementar a uma pequena regio do gene, 212 Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
baseando-se apenas no cdigo gentico. Contudo, como este cdigo degenerado (alguns aminocidos so codificados por mais que um codo), uma sonda baseada na sequncia de aminocidos deve incluir todas as possveis sequncias de oligonucleotdeos que teoricamente podem codificar a sequncia de interesse. Nesta mistura de oligonucleotdeos est presente a que hibridizar perfeitamente com o clone de interesse.
9. Bibilioteca de cDNA
Uma biblioteca de cDNA , muitas vezes, preferencialmente utilizada para se iniciar a clonagem de um gene. Isto, deve-se ao facto de, sendo o cDNA (DNA complementar) derivado do mRNA (RNA mensageiro), para uma protena expressa na clula, obtm-se bibliotecas em que a sua representao significativamente maior do que em bibliotecas genmicas, o que auxilia o seu isolamento. Outra vantagem poder deduzir-se facilmente a sequncia da protena pois o cDNA de eucariotas uma cpia do mRNA processado. Finalmente, bastante importante, para vrios trabalhos de investigao, possuir um clone de DNA completo (com todas as sequncias codificantes da protena). O cDNA uma cpia da mensagem, no possuindo assim intres e apresentando poucas sequncias que no codificam protenas. Esta ausncia de intres permite a directa transcrio e traduo dos cDNA de
| Captulo 14 Tcnicas de DNA recombinante
213
Biologia Molecular - 1 Ano - FFUP
2006/2007
clulas eucariticas superiores, em bactrias e outros microorganismos, o que permite a produo em massa de polipeptdeos importantes. A inexistncia de intres facilita igualmente a determinao directa da sequncia equncia da mensagem codificada e deduo subsequente da sequncia de aminocidos da protena por ela codificada, o que se tem demonstrado extremamente til na identificao de inmeras protenas que ainda no o tinham sido gentica ou bioquimicamente.
Figura C1: DNA genmico versus cDNA.
A sntese, clonagem e isolamento de cDNAs especficos d d-se, se, resumidamente em 4 etapas: Sntese in vitro de cDNAs de cadeia dupla partindo de mRNAs isolados do tecido de interesse, utilizando a enzima trancrptase e reversa; Preparao dos cDNAs para ligao com o vector escolhido; Introduo dos cDNAs recombinantes numa clula hospedeira para serem replicados. Dar Dar-se- a amplificao dos recombinantes e, consoante o vector utilizado, a expresso das protenas codificadas pelas mensagens que deram origem aos cDNAs; Identificao de clones especficos.
Figura C2: Construo de uma biblioteca de cDNA. 1. Isolar mRNA das clulas; 2. Usar a transcrptase reversa para fazer cadeias de DNA complementares a cada mRNA. Usar a DNA polimerase para tornar o DNA de cadeia simples em DNA de cadeia dupla. 3. Inserir em plasmdeo e transformar em E. coli.
214
Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
10. Sntese de cDNAs de cadeia dupla
A elaborao de uma biblioteca de cDNA inicia-se inicia se com o isolamento do mRNA (subs (substrato) total do tecido e com a seleco de parte de RNA poliadenilado que possui grande parte das mensagens contidas na clula. Este isolamento efectuado por cromatografia de afinidade, em colunas contendo oligo-dT. oligo A escolha deste fragmento de RNA det determinante erminante para o processo restante, pois, se estiver degradado, afectar a representatividade das sequncias na biblioteca. Aps este passo, efectua-se efectua a sntese dos cDNAs atravs de diversos processos, sendo o abaixo referido bastante comum: O mRNA utilizado como molde para a sntese da primeira cadeia de cDNA, pela enzima trancrptase reversa. Esta enzima tem a capacidade de catalizar in vitro a polimerizao de DNA a partir de RNA, necessitando de um primer com a extremidade 3OH livre, para poder comear a sntese. Normalmente o primer utilizado um pequeno oligonucleotdeo de desoxitimidinas (poli-dT) (poli que vai hibridar na cauda de poli-A A do mRNA. Resultam, desta reaco, molculas hbridas de DNA/RNA que vo funcionar para a sntese da segunda cadeia cadeia de cDNA, como um complexo de molculas molde (cDNA) e primers (mRNA). Depois da sntese da cadeia de cDNA, a enzima RNase A vai remover parcialmente o mRNA, pois esta vai cortando pedaos do RNA da molcula hbrida RNA RNA-DNA, DNA, formando nesta espaos va vazios. Os fragmentos no digeridos pela RNase A servem como primers para que a DNA polimerase substitua eficientemente o RNA por DNA, originando uma molcula de DNA de cadeia dupla. Este DNA de cadeia dupla tratado com T4 polimerase que possui actividade activ 3-5, 5, sendo utilizada para remover possveis nucleotdeos extras nas extremidades 3 do cDNA. Obtemos assim uma molcula de DNA de cadeia dupla que a cpia exacta de uma determinada mensagem (mRNA).
Figura C3: Sntese de cDNA de cadeia dupla. mRNA extrado de um dado tecido e cDNA sintetizado a partir do mRNA, por aco cataltica da transcrptase reversa: Um oligonucleotdeo complementar cauda poli-A A da extremidade 3' do mRNA hibridiza com este, servindo como primer da transcrptase reversa, ersa, que copia ento o RNA para uma cadeia complementar de DNA, formando um hbrido DNA/RNA. Tratando Tratando-o com RNase H, esta faz-lhe lhe cortes e cria espaos na cadeia de RNA. A cadeia complementar de DNA ento copiada, formando-se se cDNA de dupla cadeia por aco aco de uma DNA polimerase. O primer para esta reaco consiste no fragmento de mRNA original.
| Captulo 14 Tcnicas de DNA recombinante
215
Biologia Molecular - 1 Ano - FFUP
2006/2007
216
Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
11. Sntese qumica de oligonucletidos
12. Radioistopos
Marcao de sondas para pesquisa de DNAs ou RNAs homlogos Determinao do tempo de semi-vida e estabilidade de RNAs Estudos de sequenciao Determinao do tamanho de protenas e cidos nucleicos Reunio de macromolculas e sua distribuio na clula Tempo de semi-vida especfico tempo em que a actividade ou concentrao passa para a metade da original Actividade especfica quantidade de radioactividade por unidade de material Deteco de radioistopos: Autorradiografia Ensaios quantitativos Pulse-chase
| Captulo 14 Tcnicas de DNA recombinante
217
Biologia Molecular - 1 Ano - FFUP
2006/2007
13. Autorradiografia
Neste procedimento, as clulas vivas so expostas brevemente a um pulso de um composto radioactivo especfico e ento incubadas por um perodo varivel para lhes dar tempo de incorporarem o composto antes de serem fixadas e processadas por microscopia ptica ou electrnica. Cada preparao ento coberta com uma camada fina de emulso fotogrfica e deixada no escuro durante vrios dias, durante os quais o radioistopo decai. A emulso revelada e, a posio da radioactividade em cada clula indicada pela posio dos grnulos de prata revelados.
218
Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
Ensaios quantitativos - Os electres (partculas beta) podem ser detectados com um contador Geiger, pela ionizao que eles produzem num gs, ou podem ser medidos num contador de cintilaes pela pequena quantidade de luz que induzem no fluido de cintilao. Estes mtodos tornam possvel medir, com acuidade, a quantidade de um radioistopo em particular presente numa amostra biolgica.
Pulse-chase - As alteraes na localizao ou na forma qumica das molculas radioactivas podem ser seguidas em funo do tempo. Utiliza-se para esse efeito um protocolo de marcao de tipo pulse-chase, no qual o material radioactivo (o pulso) adicionado somente por um breve perodo de tempo e ento retirado e substitudo por molculas no-radioactivas (a caa). So retiradas amostras em intervalos regulares, e a forma qumica ou a localizao da radioactividade identificada para cada amostra.
| Captulo 14 Tcnicas de DNA recombinante
219
Biologia Molecular - 1 Ano - FFUP
2006/2007
220
Captulo 14 Tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
Captulo 15 Aplicaes das tcnicas de DNA recombinante
1. Sequenciao do DNA
A sequenciao de cidos nucleicos revela a ordem dos nucletidos ao longo de uma molcula de DNA. Pode ser realizada por um de dois mtodos, cada um dos quais resulta na produo de fragmentos de DNA de diversos comprimentos, diferindo uns dos outros por uma nica ase, e a partir dos quais se pode inferir a sequncia nucleotdica da molcula. Isto conseguido mediante a utilizao de gis desnaturantes de poliacrilamida, que discriminam molculas de DNA que diferem por uma nica base. Estes gis fazem com que as cadeias da molcula de DNA se separem e assim permaneam todo o tempo da electroforese. Como as molculas de DNA so invisveis para o observador, geralmente marcam-se, na maior parte dos casos, com istopos radioactivos. Aps electroforese, as molculas marcadas visualizam-se colocando um filme de raios-X junto do gel de poliacrilamida seco e conservando-o numa cassete durante algumas horas. Durante esse tempo, a radiao emitida pelo istopo em cada molcula de DNA impressiona o filme e, aps revelao, v-se uma banda escura na posio em que a banda de DNA estava localizada. Esta tcnica denomina-se por auto-radiografia.
1.1.
Sequenciao automtica
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
221
Biologia Molecular - 1 Ano - FFUP
2006/2007
Os didesoxinucletidos no possuem um grupo 3OH mas sim 3H. Na presena de um didesoxinucletido a sntese de DNA pra porque no se pode dar a ligao difosfato. A cadeia em crescimento termina nesse ponto e a ltima base na extremidade 3 conhecida como sequncia de terminao didesoxi. Na sequenciao pelo mtodo de Sanger, quatro misturas diferentes servem para sequenciar um fragmento de DNA pelo mtodo dos terminadores didesoxi de Sanger. Cada mistura de reaco contm a molcula de DNA molde a sequenciar, primers marcados radioactivamente, os quatro desoxinucletidos, polimerase de DNA, e um terminador didesoxi diferente (ddA, ddC, ddG ou ddT). Quando um destes terminadores incorporado na nova cadeia de DNA sintetizada, pra a sntese da cadeia; o resultado que nessa mistura de reaco as cadeias de diversos comprimentos terminam com a mesma base. Os produtos radioactivos so separados por electroforese e visualizados por auto-radiografia. A leitura do gel feita da base (fragmentos mais pequenos terminados mais prximo da extremidade 5) para o topo revela a sequncia de bases 3TACGATACGAGG5, complementar da cadeia-molde.
2. Deleces unidireccionais no DNA
222
Captulo 15 Aplicaes das tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
3. Construo de primers
4. Anlise computorizada de sequncias de DNA e protenas
Assim que sequenciar protenas e DNA se tornou banal, foram criadas bases de dados de sequncias para catalogar esta informao. Assim, tambm foram criados programas computacionais para explorar as sequncias nessas bases de dados, podendo ajudar a identificar novas sequncias de cDNA clonado. A sequncia de DNA determinada a partir de um cDNA pode ser traduzida e usada para procurar a base de dados de protenas, para determinar se essa protena conhecida ou se o cDNA foi previamente clonado. Uma pesquisa pode descobrir protenas que esto relacionadas na base da sua primeira sequncia de aminocidos (homlogas). Isto importante para ajudar a determinar a funo de uma protena desconhecida.
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
223
Biologia Molecular - 1 Ano - FFUP
2006/2007
5. Chromosome walking Hibridao sobreposta
A anlise de DNA genmico e da sua organizao tm de ser feitas de modo sistemtico. Uma maneira eficaz de o fazer usando um fago recombinante para isolar outro que contenha informao sobreposta do genoma. Esta tcnica, conhecida como Chromossome walking, depende da obteno de um pequeno segmento de DNA a partir de um fim do primeiro recombinante e usando este pedao de DNA reanalisa-se a biblioteca para obter recombinantes adicionais que contenham o pedao de DNA e a poro seguinte de genoma. O segundo recombinante usado para obter um terceiro e por a fora, at obter um conjunto de segmentos clonados sobrepostos. Chromossome walking a nica forma de procurar um gene quando a sua posio no genoma apenas aproximadamente conhecida. utilizado para a deteco de clones de bibliotecas que tm sequncias sobrepostas a partir de um local conhecido, por hibridao, sub-clonagem e mapa de restrio.
224
Captulo 15 Aplicaes das tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
6. Southern blot
Assim que um cDNA clonado isolado, este pode ser usado para analisar a expresso gnica e a organizao do DNA genmico. O mtodo mais utilizado o Southern blot, que uma ferramenta extremamente til para analisar a estrutura de um gene.
Deteco de sequncias particulares com sondas especficas Mapa fsico dos locais de restrio de um gene Nmero de cpias de um gene no genoma Grau de semelhana de um gene com outros homlogos Anlise da expresso de genes e organizao de DNA genmico Deteco de mutaes e rearranjos num determinado gene Deteco de polimorfismos genticos designados por restriction fragment lenght polymorphism (RFLP)
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
225
Biologia Molecular - 1 Ano - FFUP
2006/2007
7. Northern blot
Deteco de molculas especficas de RNA Avalia a presena, a quantidade e tamanho de um mRNA Compara mRNAs especficos com os de outros organismos diferentes
8. Western blot
Deteco de protenas especficas com anticorpos radioactivos ou fluorescentes
226
Captulo 15 Aplicaes das tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
9. Polimorfismo dos fragmentos de restrio (RFLP)
O RFLP pode ter dois significados: como uma caracterstica de molculas de DNA (partindo das suas diferentes sequncias de nucletidos) atravs dos quais podem ser distinguidos e como tcnica laboratorial que usa esta caracterstica para comparar molculas de DNA. Permite a deteco de fragmentos nicos numa populao Avaliam a presena de mutaes (inseres ou deleces) em diferentes alelos Caracterizados pela presena ou ausncia de locais de restrio particulares no DNA Correspondem impresso digital de cada indivduo (regies hipervariveis e repetidas
10. Fingerprinting
Permite a comparao de duas amostras de DNA e RNA ou de protenas, sem a necessidade de sequenciao das mesmas, por anlise do padro de fragmentos obtidos aps digesto enzimtica
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
227
Biologia Molecular - 1 Ano - FFUP
2006/2007
utilizado para estudos de paternidade e em medicina forense (identificao de suspeitos ou vtimas)
Estudos de paternidade Por anlise de elementos hipervariveis com sequncias core caractersticas VNTR (Variable Number Tandem Repeats) classe especial de repeties de nmero varivel em diferentes locais e diferentes indivduos
228
Captulo 15 Aplicaes das tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
11. Mutagnese in vitro
A mutagnese in vitro de genes clonados tornou-se uma ferramenta tradicional na anlise funcional de cidos nucleicos e protenas. Permite a adio, deleco ou mutao pontual de uma ou mais bases na sequncia de DNA, alm de possibilitar uma mutagnese direccionada.
mutao pontual
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
229
Biologia Molecular - 1 Ano - FFUP
2006/2007
12. Single Stranded Conformation Polymorphism (SSCP)
SSCP definido como uma diferena conformacional de cadeias simples de sequncias de nucletidos de tamanho idntico induzida por diferenas nestas sob determinadas condies experimentais. Esta tcnica permite detectar diferenas de um nucletido no DNA de cadeia simples e pode ser visualizada pela alterao da mobilidade das cadeias normal e mutante em gel de electroforese.
13. Determinao do local do incio da transcrio
230
Captulo 15 Aplicaes das tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
S1 nuclease usa DNA de cadeia simples (cerca de 100 nucletidos), radioactivonuma extremidade, com sequncia conhecida do promotor que permite a ligao do mRNA.
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
231
Biologia Molecular - 1 Ano - FFUP
2006/2007
Primer extension usa oligodesoxinucletidos (cerca de 10 bases), radioactivo numa extremidade, que corresponde regio a jusante do promotor
14. Hibridizao in situ com fluorocromos (FISH)
Baseia-se na complementaridade de bases enre DNA/RNA, DNA/DNA e RNA/DNA. Localiza sequncias especficas de DNA ou RNA, marcados com corantes fluorescentes (FISH) em cromossomas, ncleo e citoplasma de clulas ou tecidos
15. Anlise de cromossomas por Chromosome painting
Usa sondas de DNA de regies especficas de cromossomas com corantes fluorescentes cujo alvo so as repeties de segmentos de DNA que so especficas de cada cromossoma.
232
Captulo 15 Aplicaes das tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
16. DNA microarrays - DNA chips
Consistem em milhares de sequncias codificantes de vrios genes, previamente amplificados por PCR ligados suerfcie de uma lmina Mltiplos oligonucletidos de DNA (cerca de 20 nucletidos) sintetizados a partir de um nucletido inicial Permitem a anlise de milhares de genes em simultneo Preparados e usados para analisar a expresso de genes em diferentes rgos e tecidos Podem ser utilizados para anlise de padres de expresso em doenas e durante a diferenciao celular
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
233
Biologia Molecular - 1 Ano - FFUP
2006/2007
17. Polimerase Chain Reaction (PCR)
A PCR uma tcnica que permite amplificar exponencialmente o DNA, via replicao enzimtica, sem precisar de usar um organismo vivo. Como uma tcnica in vitro, pode ser realizada sem restries na forma de DNA e pode ser altamente modificada para que ocorram manipulaes genticas. Permite a amplificao de pequenas sequncias conhecidas de DNA a partir de dois primers Pequenas quantidades de DNA (<1g) Dois primers Magnsio, desoxinucletidos Taq polimerase e banho programvel: desnaturao (94 - 5), ligao dos primers (30 a 65 - 30), sntese de DNA (72 - 3 a 4) e controlos
18. PCR assimtrico
Para se obterem cadeias simples de DNA Um dos primers est numa concentrao maior A acumulao de DNA linear Sequenciao pelo mtodo de Sanger
234
Captulo 15 Aplicaes das tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
19. RT-PCR
A Reverse transcription polymerase chain reaction uma tcnica para amplificar um pedao definido de uma molcula de RNA. A cadeia de RNA transcrita pela transcriptase reversa para o seu DNA complementar, seguido de amplificao do DNA resultante atravs de PCR.
Isolar RNA de clulas ou tecidos Sntese de cDNA com gene-specific primers (GSP), oligo-dT-primers ou random-hexamer primers Transcriptase reversa e Taq DNA polimerase sntese de DNA de cadeia dupla
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
235
Biologia Molecular - 1 Ano - FFUP
2006/2007
20. Ligase Chain Reaction (LCR)
Requer Tap DNA polymerase e ligase termoestvel 2 primers especficos para cada cadeia de DNA 1 primer comum a cada cadeia de DNA Deteco de mutaes pontuais
21. Produo de ratinhos transgnicos
Permitem o estudo da funo e desenvolvimento de um organismo Possuem DNA proveniente de outra espcie nas clulas germinativas DNA com o gene de interesse injectado no pr-ncleo de um ovo fertilizado, antes de ter sofrido a fuso, e transferido para a me receptora 236 Captulo 15 Aplicaes das tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
22. Ratinhos Knockout
Permitem o estudo da embriognese e comportamento destes animais Avaliam certas doenas genticas do homem (fibrose cstica) Gene normal ou mutado tem um marcador gentico (cor) Possuem DNA proveniente de outra espcie nas clulas germinativas
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
237
Biologia Molecular - 1 Ano - FFUP
2006/2007
23. RNA interference (RNAi)
RNAi um mecanismo para regulao da expresso genica guiada por RNA, na qual dupla cadeia de cidos nucleicos inibe a expresso de genes com sequncias de nucletidos complementares
Determinar a funo de um gene e expresso gnica (transcrio, traduo e morte celular) Estudo das vias metablicas Identificao e validao de alvos para drogas Constitui um mecanismo celular de defesa contra certos vrus e elementos genticos mveis em plantas e animais Nas plantas, mutaes nos genes que codificam para Dicer e protenas RISC aumentam a susceptibilidade a infeces por vrus com RNA e a movimentao de transposes dentro do genoma
24. Small interference RNA (siRNA)
siRNA uma classe de molculas de cadeia dupla de RNA com 20 a 25 nucletidos que tm uma srie de papis em biologia. O mais importante que est envolvido na via RNA interference onde interfere com a expresso de um gene especfico.
238
Captulo 15 Aplicaes das tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
Funes especficas: Inibio da expresso de genes (gene knock down) Desenvolvimento e organognese Apoptose e controlo do crescimento celular Regulam negativamente a expresso genica aps a transcrio Complexo RITS (RNA induced transcriptional silencing) que liga as histonas metiladas e induz a degradao do pr-mRNA transcrito pela RNA polimerase II
Deteco: Imunoblotting Imunofluorescncia
25. RNA-induced silencing complex (RISC)
RISC um complexo siRNA multi-proteico de siRNA que quebra a dupla cadeia de RNA e liga pequenas cadeias de RNA antisense, fazendo com que fiquem capazes de se ligar cadeia complementar. Quando encontra a cadeia complementar, activada a RNase, que vai saparar o RNA.
miRNAs reprimem a traduo siRNAs conduzem clivagem do mRNA Dicer ribonuclease requerida para a formao de siRNAs
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
239
Biologia Molecular - 1 Ano - FFUP
2006/2007
26. miRNA
Em gentica, miRNAs so molculas de RNA de uma cadeia que regulam a expresso de outros genes. miRNAs so codificados por genes transcritos do DNA, mas no traduzidos para protena (RNA nocodificante), pelo contrrio, so processados a partir de transcriptos primrios conhecidos por pri-miRNA a estruturas chamadas pr-miRNA e finalmente a miRNA funcional. As molculas de miRNA so parcialmente complementares a uma ou mais molculas de mRNA e funcionam regulando negativamente a expresso gnica.
Famlias de pequenos RNAs no-codificantes Formados por 21-25 nucletidos, depois de processados de pri-mRNAs para pr-mRNAs miRNAs interagem com mRNAs alvo : sequncias especficas (3UTR) que podem apresentar complementariedade imperfeita em vrios locais o grau de complementariedade afecta a clivagem, a supresso e a degradao induzem a clivagem e inibem a traduo Drosha e Pasha: medeiam a produo de miRNAs por processamento dos miRNAs no ncleo
240
Captulo 15 Aplicaes das tcnicas de DNA recombinante |
Biologia Molecular 1 Ano - FFUP
2006/2007
| Captulo 15 Aplicaes das tcnicas de DNA recombinante
241
Biologia Molecular - 1 Ano - FFUP
2006/2007
Captulo 16 Regulao gentica nos procariotas
O termo expresso gentica refere-se ao processo atravs do qual a informao codificada num gene particular descodificada numa protena especfica. Normalmente, uma clula expressa apenas uma fraco dos seus genes, de acordo com um esquema geralmente definido durante o desenvolvimento embrionrio. Alm disso, a maioria das clulas de um organismo so capazes de alterar o seu padro de expresso gentica em resposta a sinais extrnsecos. Assim se explica como diferentes tipos de clulas num mesmo organismo diferem dramaticamente entre si. O controlo da expresso gentica difere entre procariotas e eucariotas, sendo o processo mais complexo nos ltimos. Neste captulo vamo-nos debruar sobre a regulao da expresso gentica em procariotas, que pelo facto de se tratar de clulas mais simples, a regulao ocorrer essencialmente ao nvel da transcrio, em detrimento da traduo. As regies reguladoras podem ser simples ou complexas mas todas funcionam como interruptores, com dois componentes fundamentais: curtos segmentos de DNA, que definem uma sequncia, e protenas reguladoras, que reconhecem e se ligam ao DNA afectando a transcrio. Nos mecanismos de regulao coexistem controlos negativos e positivos atravs da aco de repressores e activadores, respectivamente. Ou seja, as protenas que se ligam ao DNA, vo afectar a transcrio atravs da produo de enzimas indutveis ou repressveis. Estes mecanismos so, ento, constitudos por dois domnios um de ligao, que reconhece uma sequncia especfica de DNA ao qual se liga, e um inibidor ou activador, que diminui ou acelera o nvel da iniciao da transcrio, respectivamente. a ligao entre a transcrio e a traduo que rege esta expresso gentica, e o controlo est directamente relacionado com modificaes nutricionais ligadas ao crescimento e diviso celular. Tipicamente um gene procariota (ex: bactria) controlado por apenas uma ou duas protenas reguladoras. As regies reguladoras so simples. Muitos dos genes ao serem transcritos encontram-se em locais adjacentes no cromossoma formando um opero. A expresso dos genes depende de protenas que regulam o acesso da RNA polimerase ao promotor. Este modelo composto, ento pelo: opero (unidade funcional constituda pelos genes estruturais, promotor e operador) e pelos genes reguladores/repressores.
242
Captulo 16 Regulao gentica nos procariotas |
Biologia Molecular 1 Ano - FFUP
2006/2007
- Genes reguladores codificam para protenas reguladores que ajudam a sentir os sinais do meio exterior e regulam a velocidade de transcrio dos genes estruturais. Ou seja, este controla o operador, que por sua vez controla os genes estruturais. Isto porque os genes reguladores produzem uma protena chamada repressor, que se liga ao operador, impedindo a ligao da RNA polimerase ao promotor e portanto impedindo a transcrio dos genes estruturais que codificam determinadas protenas. O gene regulador vai ter o seu prprio promotor. - Operador local onde se liga o repressor; - Promotor - sequncia especfica de nucletidos do DNA qual se liga a RNA polimerase e onde tem incio a transcrio; - Genes estruturais codificam para as protenas requeridas;
1. Regulo
Existem situaes em que um grupo de operes controlado por um nico tipo de regulador. Esse grupo de operes toma a designao de regulo. O regulo activado pela ligao de uma molcula especfica. Na sua forma activa, o regulador liga-se a um local a montante do promotor, facilitando o acesso da RNA polimerase ao promotor e induzindo a transcrio. A existncia deste regulo permite controlar simultaneamente um conjunto de operes envolvidos em determinada tarefa. Ao activar estes operes com o mesmo regulador (que funciona como repressor ou indutor) tm-se uma eficincia maior nessa tarefa, permitindo satisfazer mais rapidamente as necessidades da clula.
2. Protenas activadoras ou reguladoras positivas
As protena reguladoras vo estimular directamente a expresso dos genes, ligando-se ao DNA e facilitando assim o acesso da RNA polimerase. Algumas destas protenas activadoras necessitam da ligao de uma molcula especfica, o efector, para ficarem activas e assim se poderem ligar a um local a montante do promotor, atraindo a RNA polimerase ao promotor e induzindo a transcrio dos genes. Quando uma protena activadora que no necessita de efector est presente em grande concentrao, esta vai-se ligar tambm a um local a montante do promotor seguindo todo o processo anteriormente descrito.
| Captulo 16 Regulao gentica nos procariotas
243
Biologia Molecular - 1 Ano - FFUP
2006/2007
3. Protenas repressoras ou reguladoras negativas
As protenas repressoras, ou repressores, vo se ligar ao DNA e impedindo o acesso da RNA polimerase. O opero , ento, bloqueado pela forma activa do repressor. A ligao do repressor activo ao operador impede a ligao da RNA polimerase ao promotor e inibe a transcrio. Este tipo de regulao depende da aco dos efectores: * Indutores - diminuem a capacidade de ligao dos repressores ( ex.: Opero da lactose) * Co-repressores - conjugam-se com os repressores e facilitam a ligao dos mesmos (ex.: Opero do triptofano)
a) Protena repressora encontra-se activa, tendo a capacidade de se ligar ao operador, impedindo assim a ligao da RNA polimerase ao promotor, no se efectuando a transcrio. No entanto, quando no meio est presente um indutor, este vai-se ligar ao repressor e alterar a sua forma, o que far com que o repressor se desligue da regio operadora e a RNA polimerase se possa ligar ao promotor efectuando-se a transcrio. b) O repressor encontra-se na forma inactiva, logo no tem capacidade para se ligar ao operador, podendo a RNA polimerase ligar-se ao promotor e fazer a transcrio dos genes estruturais. No entanto, quando um co-repressor est presente no meio, este vai-se ligar protena repressora inactiva, formando este complexo uma protena repressora activa que se vai poder ligar ao operador, bloqueando assim a transcrio dos genes estruturais do opero.
244
Captulo 16 Regulao gentica nos procariotas |
Biologia Molecular 1 Ano - FFUP
2006/2007
4. Opero da lactose
Estrutura: O opero da lactose formado por trs genes estruturais: - lacZ - -galactosidase - lac Y - galactose permease - lac A - galactose transcacetilase Estes codificam as enzimas necessrias ao metabolismo da lactose. Este tambm constitudo pelo promotor (P) e pelo operador (O). Fora do opero lac existe o gene regulador que codifica o repressor (lac I), e que possui igualmente um promotor. Da ele ser constantemente transcrito e traduzido, produzindo a bactria, continuamente, pequenas quantidades de protena repressora.
Regulao do opero da lactose: Como j se referiu o gene regulador est continuamente a produzir protena repressora activa. Quando no existe lactose no meio, o repressor liga-se regio operadora, bloqueando o acesso da RNA polimerase ao promotor, no ocorrendo assim a transcrio do opero. Quando existe lactose no meio, esta molcula liga-se ao repressor, altera a sua conformao de tal forma que este se torna inactivo, desligando-se do operador. A RNA polimerase pode-se agora ligar ao promotor efectuando assim a transcrio dos genes estruturais, que so posteriormente traduzidos, formando-se as enzimas necessrias ao metabolismo da lactose. A lactose funciona como um indutor, pois a sua presena permite activar o opero. O opero da lactose pode ser designado assim por opero indutvel. Estes fenmenos de auto regulao garantem uma poupana de recursos.
| Captulo 16 Regulao gentica nos procariotas
245
Biologia Molecular - 1 Ano - FFUP
2006/2007
4.1. Anlise de mutaes
Mutaes no operador (O) Oc = operador constitutivo O operador no permite a ligao de um repressor.
Mutaes no repressor (I) I- = mutao que impede a ligao do repressor ao DNA Is = mutao no gene I que impede a ligao do indutor Iq = mutao no gene I que permite a formao de repressor e que impede a expresso basal dos genes estruturais
Mutaes no promotor (P) P- = impedem a ligao da RNA polimerase
Mutaes cis-acting ou cis-dominantes - afectam o promotor e o operador
246
Captulo 16 Regulao gentica nos procariotas |
Biologia Molecular 1 Ano - FFUP
2006/2007
- afectam a expresso dos genes sob o seu controlo no mesmo DNA, mas no afectam no DNA onde no h mutao
Mutaes trans-acting ou trans-dominantes
Mutaes no operador (Oc): Clulas diplides heterozigticas O+/Oc demonstram que os operadores so cis-acting dominantes. Isto porque se o operador Oc, o repressor no se pode ligar a este, e assim os genes estruturais deste opero so expressos mesmo na ausncia de um indutor. No entanto, se o operador da outra cadeia de DNA O+, os genes estruturais adjacentes a este operador no so expressos pois o repressor liga-se a este.
Mutaes no repressor (I-): A natureza recessiva das mutaes I- demonstra que o repressor trans-acting. Pois, embora nenhum repressor activo seja sintetizado a partir do gene I-, o gene I+ sintetiza um repressor funcional, que se ir ligar a ambos os operadores da clula diplide e bloquear a transcrio do opero da lactose.
Mutaes no repressor (Is): A dominncia da mutao Is deve-se inactivao do local alostrico do repressor da lactose numa clula Is/I+, que impede que o indutor se ligue a este. Em contraste com o repressor normal, proveniente de I+, o repressor Is no inactivado pelo indutor, devido mudana de conformao do seu local alostrico. Assim, mesmo na presena de um indutor o repressor Is vai-se ligar a ambos os operadores da clula diplide (mesmo no DNA em que o repressor no sofreu mutao), e impede a expresso dos genes do opero da lactose.
| Captulo 16 Regulao gentica nos procariotas
247
Biologia Molecular - 1 Ano - FFUP
2006/2007
4.2. Controlo catablico do opero da lactose
A protena CAP (catabolite activator protein) em conjunto com o AMP cclico (cAMP) um controlador positivo do opero da lactose. Ou seja, s ocorre transcrio quando a CAP e o cAMP formam um complexo que actua como um activador da transcrio dos genes do opero da lactose, permitindo que a RNA polimerase se ligue ao promotor. Quando o cAMP est presente vai actuar como um efector alostrico, complexando com CAP. A glucose um dos catabolitos do opero lactose na E.coli. Quando presente em elevadas concentraes a glucose inactiva a enzima adenilato ciclase impedindo que esta catalise a converso de ATP em cAMP. Com isto a glucose vai impedir a expresso do opero lactose pois no existe cAMP para formar o complexo activador com a CAP. Quando a concentrao de glucose baixa o opero lactose activado, pois a adenilato ciclase j pode catabolizar a converso de ATP em cAMP. Um aumento da concentrao de cAMP leva formao do complexo com a CAP e ao incio da transcrio dos genes do opero.
O controlo do opero da lactose feito pelo repressor da lactose, pela CAP e pela cAMP. No caso a) no est presente lactose no meio logo no ocorre sntese de mRNA, quer exista glucose ou no, porque o repressor est ligado ao operador. Em b) est presente lactose, que se liga ao repressor e altera a sua forma fazendo com que este se desligue do operador. Contudo existe glucose no meio o que faz com que a 248 Captulo 16 Regulao gentica nos procariotas |
Biologia Molecular 1 Ano - FFUP
2006/2007
concentrao de cAMP seja baixa, logo este no pode fazer um complexo com a CAP e esta no se pode ligar ao promotor. Como resultado ocorre sntese de mRNA, mas uma cadeia pequena. Em c) com a presena de lactose e ausncia de glucose feita a transcrio mxima. Isto porque, ao contrrio do caso anterior, a ausncia de glucose faz com que a concentrao de cAMP aumente. Esta vai complexar com a CAP permitindo que esta se ligue ao promotor na posio-35, activando a transcrio do opero lac. A ligao deste complexo ao DNA na regio promotora faz com que este curve 90 nesta zona. Aparentemente, a RNA polimerase liga-se/trabalha mais eficazmente quando o promotor est nesta forma. Como efeito ocorre sntese de vrias cadeias de lac mRNA, intensificando a transcrio.
5. Opero da arabinose
A arabinose um acar que uma fonte de energia e de carbono para as bactrias. O opero arabinose constitudo por trs genes estruturais: - gene A (araA)- cinase - gene B (araB)- isomerase - gene D (araD)- epimerase Estes codificam para trs enzimas envolvidas na degradao catablica da arabinose. Estes genes so transcritos a partir de um nico promotor, PBAD. A transcrio dos trs genes requer a presena simultnea da arabinose, da RNA polimerase e da protena araC, uma protena que actua na forma de dmero, funcionando como activadora ou repressora, produzida pelo gene C. O gene C um gene auto regulado que tem o seu prprio promotor (PC). No modelo do opero da lactose existe tambm dois operadores: o O1 e O2, e dois genes reguladores: o I1 e o I2, que so quatro stios de ligao que tambm regulam a transcrio dos genes BAD.
| Captulo 16 Regulao gentica nos procariotas
249
Biologia Molecular - 1 Ano - FFUP
2006/2007
5.1. Controlo positivo e negativo do opero da arabinose
Quando existe arabinose, a bactria utiliza a arabinose que vai interagir directamente com a protena araC. Esta interaco faz com que a araC altere a sua conformao o que favorece a ligao da RNA polimerase ao promotor PBAD e, consequentemente, a transcrio dos genes araA, araB e araC. As trs enzimas hidrolticas catalizadas por estes genes so produzidas e vo degradar a arabinose. Quando a arabinose se esgota a protena araC volta sua forma original e deixa de ocorrer transcrio dos genes do opero arabinose. Ou seja no opero da arabinose vo existir dois tipos de controlos: Controlo negativo sem arabinose, monmeros de araC ligam-se a O2 e I1, dobrando o DNA e bloqueando o acesso da RNA polimerase ao promotor PBAD. Para alm disso existe glucose no meio o que faz com a que as concentrao de cAMP baixem no se podendo formar o complexo com a CAP. No h transcrio. Controlo positivo a arabinose existente no meio liga-se a araC modificando a sua conformao de forma que araC passa ligar-se como dmero a I1 e I2 (regio araI) e no a O2. Isto permite a ligao da RNA polimerase ao promotor e a transcrio. No havendo glucose no meio o complexo CAP-cAMP existe em concentrao suficientemente alta para se ligar ao activador o que estimula a melhor ligao da RNA polimerase ao promotor e uma maior taxa de transcrio.
5.2. Regulao do opero da arabinose
Quando no existe arabinose no meio, o dmero araC assume uma conformao que lhe permite ligar-se a O1, O2 e regio I apenas no local I1. os dmeros ligados a O2 e I1 interagem fazem com que se forme um loop no DNA. Esta conformao impossibilita o acesso da RNA polimerase ao promotor PBAD, no ocorrendo assim a transcrio dos genes BAD. A araC tambm vai controlar a sua prpria transcrio, sendo por isso o gene C auto regulado. Esta conformao de DNA faz com que sejam sintetizados baixos nveis de araC mRNA. Na presena de arabinose, formado um complexo entre este acar e o dmero araC. Isto faz com que a conformao de araC modifique e consequentemente os dmeros ligados a O2 e I1 vo deixar de interagir passando o DNA a ter outra conformao. A araC+arabinose vai se poder ligar agora tanto a I1 como a I2 o que vai activar a transcrio dos genes BAD. Como os dmeros de O1 e O2 vo interagir isto possibilitar a sntese elevada de araC mRNA.
250
Captulo 16 Regulao gentica nos procariotas |
Biologia Molecular 1 Ano - FFUP
2006/2007
6. Opero do triptofano
O opero do triptofano formado por 5 genes estruturais: trp E, trp D, trp C, trp B e trp A, que codificam as enzimas necessrias sntese do aminocido triptofano. Estes esto associados a uma regio reguladora que contm: - Promotor (P) - Operador (O) - Sequncia leader (L), que tem na sua extremidade 3 um terminador de transcrio formado por um lao de 8 uridinas que designado por atenuador. O lao pode ser formado entre a regio 3 e 4 (muito triptofano), entre a regio 2 e 3 (pouco triptofano) ou entre a regio 1 e 2 e 3 e 4 (o ribossoma no faz a traduo do peptdo sinal). O peptdo leader tem 14 aa e 2 so triptofanos. Esta sequncia responsvel pelo processo de atenuao. semelhana do opero da lactose, tambm existe um gene mais distante que codifica um repressor, que neste caso apresenta-se na forma inactiva, no podendo assim ligar-se ao operador do opero do triptofano. Esta situao verifica-se quando os nveis intracelulares de triptofano so baixos e, assim, as enzimas necessrias sntese deste aa so produzidas, conduzindo a um aumento da concentrao de triptofano.
| Captulo 16 Regulao gentica nos procariotas
251
Biologia Molecular - 1 Ano - FFUP
2006/2007
Quando a concentrao deste aa comea a atingir nveis elevados, algumas molculas de triptofano ligam-se ao repressor, modificam a sua forma e tornam-no activo, diz se por isso que o triptofano funciona como co-repressor. Desta forma, o repressor liga-se ao operador, bloqueando a transcrio dos genes estruturais do opero. O opero triptofano ter ento dois mecanismos de regulao: um mecanismo que envolve o repressor e outro de atenuao transcripcional que veremos de seguida.
6.1. Interveno de uma atenuao
O mRNA produzido constitudo por 4 sequncias importantes representadas a vermelho, que tm uma particularidade interessante: podem parear duas a duas, 1 com 2, 2 com 3 ou 3 com 4, formando sempre hairpins. Apenas o hairpin 3-4 um hairpin de terminao, pois seguido de um poli U. Por outro lado, apenas a primeira sequncia possui codes para triptofano (dois seguidos, neste caso). A sntese do mRNA inicia-se assim que o operador liberado. O ribossoma liga-se sequncia de Shine Delargno acima da regio 1 e desliza at encontrar o primeiro AUG. Ao entrar na regio 1 o ribossoma vai encontrar dois codes para triptofano. A sequncia leader serve para interromper precocemente a sntese de mRNA caso ainda haja algum triptofano no citoplasma bacteriano, insuficiente para activar o repressor mas suficiente para garantir a produo de protenas. Assim, a concentrao de triptofano na clula vai determinar a velocidade de traduo do peptdo leader. Desta forma a velocidade de movimento do ribossoma na regio leader do mRNA reflecte a concentrao de triptofano-tRNA no citoplasma da bactria.
A) Alta concentrao de triptofano: trp-tRNA Em condies de excesso de triptofano (tRNAs carregados com triptofano), o ribossoma que traduz o RNA lder no para na regio 1, traduzindo os codes para o triptofano, e progride para a sequncia 2 bloqueando esta regio atenuadora. Assim, o ribossoma ir impedir a formao do hairpin 1-2 ou 2-3, e com isto a sequencia 3 pode emparelhar coma sequncia 4 formando o hairpin terminao seguido de uma regio poli U. A transcrio pra e as enzimas necessrias para a sntese do triptofano no so expressas.
252
Captulo 16 Regulao gentica nos procariotas |
Biologia Molecular 1 Ano - FFUP
2006/2007
B) Baixas concentraes de triptofano: Nesta situao no h tRNA carregado com triptofano. O ribossoma faz uma pausa nos codes adjacentes para o triptofano, no incio da sequncia 1 (regio que codifica o pptido leader). Como a sequencia 1 esta ligada ao ribossoma a sequncia 2 est livre para emparelhar coma sequncia 3, formando-se um hairpin 2-3, que previne a formao do hairpin de terminao 3-4. Com isto a RNA polimerase pode prosseguir o seu percurso no opero, ocorrendo assim a transcrio e a sntese de enzimas para formao de triptofano.
C) No h sntese de protenas Em condies nas quais o peptdo lder no traduzido, forma-se o hairpin 1-2, impedindo formao do hairpin 2-3 e permitindo formao do hairpin de terminao 3-4. no ocorre transcrio.
A atenuao da transcrio ter como objectivo economizar energia da bactria, que no precisa sintetizar enzimas para a produo de um aminocido que ela ainda dispe, embora em pequena quantidade.
| Captulo 16 Regulao gentica nos procariotas
253
Biologia Molecular - 1 Ano - FFUP
2006/2007
7. Outras formas de regulao
7.1. Regulao dos sistemas de reparao
Os genes SOS, que so cerca de 15 (recA, uvrA, uvrB), so induzidos quando ocorrem leses severas que impedem a replicao do DNA. O repressor LexA controla a resposta SOS em E. coli sendo capaz de reconhecer e ligar-se com diferente especificidade nas regies que controlam a transcrio dos genes cujos produtos desempenham funo de reparo de danos no DNA. Em clulas no danificadas, um grupo de operes que constituem o regulo SOS regulado por um repressor comum, LexA. Esse regulo inclui o prprio gene LexA (auto-regulao) e o gene recA, juntamente com muitos genes que codificam enzimas que agem reparando leses. H uma transcrio baixa, constitutiva de muitos desses genes, incluindo LexA e recA, mesmo quando a protena LexA est presente. Durante a replicao, quando h dmeros de pirimidina, a DNA polimerase impedida de replicar esta sequncia e, ento, a replicao reinicia-se mais adiante. Aqui a protena RecA liga-se ao DNA de cadeia simples lesada e activada, fazendo uma clivagem proteoltica do repressor dos genes SOS, o repressor LexA. Os fragmentos clivados de LexA no se podem ligar aos operadores, de modo que todo o conjunto de operes induzido. Muito mais RecA feita, juntamente com outras protenas, incluindo LexA. Enquanto existirem leses, RecA permanecer activada, e a LexA continuar a ser clivada para que os operes permaneam ligados. Quando as leses so reparadas, RecA desactivada e no mais ajuda a clivar LexA, de modo que LexA activa se liga ao operador dos operes e desliga os genes desse regulo.
254
Captulo 16 Regulao gentica nos procariotas |
Biologia Molecular 1 Ano - FFUP
2006/2007
7.2.
Regulao da terminao da transcrio
- Terminao dependente do factor Rho durante a transcrio o factor Rho ligado ao RNA persegue a RNA polimerase. Quando a RNA sofre um abrandamento, ou pra, devido formao de um hairpin o factor Rho consegue apanh-la e induzir terminao da transcrio; - Terminao independente do factor Rho a sequncia de terminao do triptofano um exemplo de terminao independente de factor Rho. Neste caso, o mRNA forma um hairpin rico em G-C, procedido de uma srie de Us, caracterstica de um local de terminao independente de Rho. - Polaridade gentica na transcrio e na traduo - Anti-terminao: capacidade de passar os locais de terminao pode ser usado um mecanismo de antiterminao para controlar a transcrio. Os factores de anti-terminao actuam como subunidades da RNA polimerase fazendo com que esta ignore os locais de terminao e continue a transcrio.
7.3. Regulao de sistemas repressveis
Esta regulao requer o reconhecimento de promotores especficos e requer tambm a ligao de RNA polimerases especficas.
7.4. Regulao na traduo
Os genes das protenas ribossomais esto organizados em 4 operes. O controlo primrio feito a nvel da traduo do mRNA (por reduo da actividade do mRNA como molde) e no na sntese de protenas.
7.5. Regulao atravs da aco de alarmonas
As alarmonas formam-se na resposta aguda face a sinais de esgotamento da fonte de aminocidos, hidratos de carbono, folatos, dano oxidativo, etc. Estas podem ser definidas como sensores de determinadas situaes de stress. Nos eucariontes a aco de alarmonas est associada paragem da replicao do DNA.
| Captulo 16 Regulao gentica nos procariotas
255
Biologia Molecular - 1 Ano - FFUP
2006/2007
Composto
Condies que induzem a sua sntese Ausncia de fontes de carbono (hidratos de carbono)
Funo
cAMP (AMP cclico)
Estimular a sntese de enzimas que metabolizem a sntese de outros acares; Controlar a expresso de outras protenas, necessrias quando h falta de carbono Inibir a sntese de RNA estvel; controlar outros processos celulares, de forma a que a clula se adapta as condies de falta de aa ?
ppGpp, pppGpp
Falta de aminocidos
Appppa
Bactria: dano oxidativo (?) Eucariontes: forquilha de replicao presa; transio normal para a fase S do ciclo celular
Estimular a sntese de DNA e proliferao celular ?
ZTP
Falta de folatos
7.6. Regulao do ciclo de vida do fago
O genoma do fago tem cerca de 50Kb. Como j se viu a sua replicao inicial feita de forma bidireccional e a replicao tardia feita em crculos rolantes. O a regio cos presente no DNA do fago que vai determinar a quantidade de genoma introduzido no capsdio. Dependendo das condies os fago pode ter um ciclo ltico (lise celular) ou um ciclo lisognico (integrao do DNA do fago no cromossoma da bactria). Existem duas protenas responsveis pela entrada do fago ou no ciclo ltico ou no ciclo lisognico. Quando ocorre a infeco inicial, h uma activao dos genes iniciais. - Gene N (esquerda dos genes iniciais) tem capacidade de anti-terminao (permite a expresso dos genes esquerda de N direita de cro); associado ao ciclo lisognico. - cro (direita dos genes iniciais) um repressor; gene Q activa a expresso dos genes tardios; associado ao ciclo ltico.
256
Captulo 16 Regulao gentica nos procariotas |
Biologia Molecular 1 Ano - FFUP
2006/2007
- Expresso dos genes iniciais:
Quando o DNA de entra numa clula o ciclo comea sempre do mesmo modo, produzindo protenas N e cro, genes iniciais. Eles so expressos a partir dos promotores PL (esquerda) e PR (direita), respectivamente. No inicio da infeco pelo fago, a transcrio destes genes para em TL e TR, devido a sinais de terminao ai presentes (nut site). Como consequncias so transcritos pequenos fragmentemos de mRNA que depois da traduo do origem as protenas N e cro. Com o prosseguimento da infeco a concentrao da protena N vai aumentando at que esta ganha capacidade de anti-terminao, fazendo com que a RNA polimerase passe os sinais de terminao e continue a expresso dos genes esquerda de TL. Esta protena vai tambm permitir a expresso dos genes direita de TR, sendo assim expressos os genes seguintes. Esta aco de anti terminador da protena N vai permitir, ento, a transcrio de uma cadeia de mRNA maior e a expresso de um maior nmero de genes.
| Captulo 16 Regulao gentica nos procariotas
257
Biologia Molecular - 1 Ano - FFUP
2006/2007
- Genes iniciais transcritos aps a infeco do fago : Com a anti terminao, o fago lambda entrou na fase de expresso de genes intermdios do ciclo, que incluem os genes cII e cIII. H esquerda de TL comeam a ser expressos os genes da protena cIII. Esta ao ser expressa vai inibir a expresso de HfL, que uma protena que feita pela bactria. A HfL inibia a expresso da protena cII, ao ser inibida HfL a cII expressa. Esta protena vai activar a transcrio a partir de PE, comeando a protena cI ser expressa. As protenas cI e cro vo competir pela regio operadora (Or3, Or2, Or1), onde se encontra esquerda o promotor PRM, que promove a transcrio de cI, e direita o promotor PR, que promove a transcrio de cro. Neste caso a protena cI vai ter maior afinidade para a regio operadora. O promotor PRM ficar livre enquanto que promotor PR ser bloqueado. cI ainda vai auxiliar a ligao da RNA polimerase ao promotor PRM. Com isto a protena cI sintetizada, e como o promotor de cro ficou bloqueado no h sntese dos genes intermedirios e o fago entra na via lisognica. A protena cI age como um estimulador da sntese de mais cI, ao ligar-se ao operador, mas tambm age como um repressor da sntese a partir de PR e PL. Com isto, na via lisognica o nico gene expresso a partir de lambda cI, a partir de PRM.
- Manuteno do ciclo lisognico: Como j se viu, a protena cI tem mais afinidade para a regio operadora OR. Esta regio pode ser dividida em 3 partes: Or1, Or2 e Or3. Os dmeros de cI tm uma ordem de afinidade Or1> Or2> Or3, enquanto que os dmeros de cro ligam-se na ordem Or3> Or2> Or1. Como a protena cI tem 10 vezes mais afinidade para o DNA do que cro, d-se a ligao de cI a Or1 e Or2, activando a transcrio de mais cI a partir de PRM e reprimindo a transcrio de cro a partir de PR.
258
Captulo 16 Regulao gentica nos procariotas |
Biologia Molecular 1 Ano - FFUP
2006/2007
- Activao do ciclo ltico: Quando ocorre a clivagem do dmero de cI, ligado a Or1 e Or2, devido a radiao UV, a protena cro comea a ser expressa e liga-se regio Or3, bloqueando o acesso da RNA polimerase ao promotor PRM. Com isto, a sntese de cI pra e o fago comea a sintetizar as protenas necessrias para a via ltica.
- Efeito dos nutrientes no estabelecimento do ciclo ltico ou lisognico:
Meio pobre em nutrientes Hf1 inibida por cIII, logo os nveis da protena cII aumentam, o que faz com que aumente tambm a expresso de cI a partir de PE. Com isto no h expresso de cro e o fago entra na via lisognica. Meio rico em nutrientes HfI no inibida por cIII, logo os nveis da protena cII so baixos, o que faz com que no ocorra expresso de cI a partir PE. Como no h cI no meio, ocorre a expresso de cro, entrando o fago na via ltica que levar posteriormente lise da bactria.
| Captulo 16 Regulao gentica nos procariotas
259
Você também pode gostar
- Genética Médica para Médicos e Estudantes de Medicina: OncologiaNo EverandGenética Médica para Médicos e Estudantes de Medicina: OncologiaAinda não há avaliações
- Tradução ProtéicaDocumento49 páginasTradução Protéicacarolsous100% (1)
- Aula 1 Visão Geral Da CélulaDocumento33 páginasAula 1 Visão Geral Da Célulaapi-19738142Ainda não há avaliações
- Bioquimica Da CelulaDocumento19 páginasBioquimica Da CelulaEmília RodriguesAinda não há avaliações
- Casos Clínicos em BioquímicaDocumento132 páginasCasos Clínicos em BioquímicamartaAinda não há avaliações
- Histologia e EmbriologiaDocumento45 páginasHistologia e Embriologiaadrienerosceli100% (3)
- Ciclo celular e replicação do DNADocumento62 páginasCiclo celular e replicação do DNAFrancielli Valerio de OliveiraAinda não há avaliações
- Homeostase e Termorregulação - PHDocumento26 páginasHomeostase e Termorregulação - PHPaulo Henrique0% (1)
- Propriedades das respostas imunes veterináriasDocumento5 páginasPropriedades das respostas imunes veterináriasLara BorgesAinda não há avaliações
- Biologia Celular: Estruturas e FunçõesDocumento11 páginasBiologia Celular: Estruturas e FunçõesDiogo RibasAinda não há avaliações
- Via das pentoses produz NADPHDocumento8 páginasVia das pentoses produz NADPHPedro Henrique Azevedo100% (1)
- 2-Introdução A Fisiologia e Homeostasia-1Documento46 páginas2-Introdução A Fisiologia e Homeostasia-1Kássia BortolettoAinda não há avaliações
- Anomalias LeucocitáriasDocumento95 páginasAnomalias LeucocitáriasGregroy86% (7)
- Núcleo, Diferenciação e Morte CelularDocumento69 páginasNúcleo, Diferenciação e Morte CelularAmanda CristinaAinda não há avaliações
- Biologia PPT - Aula 06 Proteínas e EnzimasDocumento26 páginasBiologia PPT - Aula 06 Proteínas e EnzimasBiologia ppt100% (14)
- Biologia Celular e Molecular: Estudo das estruturas e funções das célulasDocumento98 páginasBiologia Celular e Molecular: Estudo das estruturas e funções das célulasD CostaAinda não há avaliações
- Fisiologia cardiovascular e contração muscular cardíacaDocumento18 páginasFisiologia cardiovascular e contração muscular cardíacaJairo Azevedo100% (2)
- ElisaDocumento18 páginasElisaOtávio Gomes Gueiros100% (4)
- Apostila de Imunologia Clínica 2013Documento87 páginasApostila de Imunologia Clínica 2013sergiomolivAinda não há avaliações
- Biofísica – Escala BiológicaDocumento26 páginasBiofísica – Escala BiológicaRodrigo DuarteAinda não há avaliações
- Prova Bioquímica ENFERMAGEMDocumento6 páginasProva Bioquímica ENFERMAGEMSLANGIEAinda não há avaliações
- Patologia - Resumo Robbins 1 - Adaptação, Dano e Morte CelularDocumento11 páginasPatologia - Resumo Robbins 1 - Adaptação, Dano e Morte Celularwiller cassiano100% (2)
- INFLAMAÇÃO E RESPOSTA IMUNEDocumento5 páginasINFLAMAÇÃO E RESPOSTA IMUNEmarisademouraAinda não há avaliações
- Roteiro de Estudo EmbriologiaDocumento8 páginasRoteiro de Estudo EmbriologiaMarcos Marzano100% (1)
- Tema 7 - Transcrição Do Dna - Versão Final PDFDocumento50 páginasTema 7 - Transcrição Do Dna - Versão Final PDFPedro SilvaAinda não há avaliações
- Estrutura dos ácidos nucleicos e nucleotídeosDocumento42 páginasEstrutura dos ácidos nucleicos e nucleotídeosJulioCesardeSouzaAinda não há avaliações
- Embriologia: Desenvolvimento Humano e TerminologiaDocumento52 páginasEmbriologia: Desenvolvimento Humano e TerminologiaAmarildo Henrique Junior100% (4)
- Sistema imune: respostas inata e adaptativaDocumento8 páginasSistema imune: respostas inata e adaptativamarcial_pinazoAinda não há avaliações
- Bases moleculares do câncerDocumento64 páginasBases moleculares do câncerbrenoAinda não há avaliações
- DNA Transcrição ProcessoDocumento2 páginasDNA Transcrição ProcessoÉrika FernandesAinda não há avaliações
- Cromossomos GenéticaDocumento3 páginasCromossomos GenéticaJosival JuniorAinda não há avaliações
- Replicação DNADocumento7 páginasReplicação DNAarturpanda panzoAinda não há avaliações
- Bioquímica Fisiológica IDocumento85 páginasBioquímica Fisiológica IBeatriz SPAinda não há avaliações
- DNA e RNA: estrutura e funçãoDocumento53 páginasDNA e RNA: estrutura e funçãoBetânia AraújoAinda não há avaliações
- Gabarito Estudo Dirigido de Trascricao e TraducaoDocumento6 páginasGabarito Estudo Dirigido de Trascricao e TraducaoNelson Jardim FilhoAinda não há avaliações
- Fisiologia do trato gastrointestinalDocumento16 páginasFisiologia do trato gastrointestinalJuan David TovarAinda não há avaliações
- Estudo Dirigido 02-Bioquímica MetabólicaDocumento2 páginasEstudo Dirigido 02-Bioquímica MetabólicaMarcio Chao0% (1)
- Aula 01 DNA, RNA, História Da Genética. Genomas.Documento58 páginasAula 01 DNA, RNA, História Da Genética. Genomas.Davi ÁlefeAinda não há avaliações
- Resumo sobre aminoácidos e estruturas de proteínasDocumento13 páginasResumo sobre aminoácidos e estruturas de proteínasIvanilda MedAinda não há avaliações
- PEROXISSOMOSDocumento12 páginasPEROXISSOMOSelizangelacarAinda não há avaliações
- Aula 1. Introdução À Biologia MolecularDocumento29 páginasAula 1. Introdução À Biologia MolecularVanessa OliveiraAinda não há avaliações
- DNA Transcription and Translation ProcessDocumento36 páginasDNA Transcription and Translation ProcessSilvestre Viana100% (1)
- CarboidratosDocumento8 páginasCarboidratosDeivid AlencarAinda não há avaliações
- Da Antiguidade à Redescoberta das Leis de MendelNo EverandDa Antiguidade à Redescoberta das Leis de MendelAinda não há avaliações
- No caminho certo após uma cirurgia cardíaca: Um guia completo com orientações e recomendações para o pós-operatórioNo EverandNo caminho certo após uma cirurgia cardíaca: Um guia completo com orientações e recomendações para o pós-operatórioAinda não há avaliações
- Apostila GENETICADocumento66 páginasApostila GENETICAChristiane BarbiAinda não há avaliações
- 04 - Genoma BacterianoDocumento6 páginas04 - Genoma BacterianoJoão LuccasAinda não há avaliações
- Genoma bacteriano e mecanismos de resistênciaDocumento6 páginasGenoma bacteriano e mecanismos de resistênciaSauloMachadoAinda não há avaliações
- Roteiro de Estudo 01Documento3 páginasRoteiro de Estudo 01Artur Fiais100% (1)
- ESTUDO DIRIGIDO - Núcleo Celular e Divisão CelularDocumento3 páginasESTUDO DIRIGIDO - Núcleo Celular e Divisão CelularGUILHERME HOOPER DE AVELOS SANTOS100% (1)
- Crescimento e Renovação CelularDocumento7 páginasCrescimento e Renovação CelularJoana TeixeiraAinda não há avaliações
- Nivel Celular 10 ClasseDocumento10 páginasNivel Celular 10 ClasseLunguequi Júlio De Jesus MuzolaAinda não há avaliações
- Organização CelularDocumento9 páginasOrganização CelularAna Carolina SanaiottiAinda não há avaliações
- CURSO DE EDUCAÇÃO FÍSICADocumento6 páginasCURSO DE EDUCAÇÃO FÍSICAGrazi MoreiraAinda não há avaliações
- Ângulos e TriângulosDocumento3 páginasÂngulos e TriângulosVânia Maria SantosAinda não há avaliações
- Ficha1 - Fisica 11Documento3 páginasFicha1 - Fisica 11Vânia Maria SantosAinda não há avaliações
- Ficha Sobre Sismicidade PDFDocumento4 páginasFicha Sobre Sismicidade PDFVânia Maria SantosAinda não há avaliações
- Teste de Ciências Naturais 8o anoDocumento4 páginasTeste de Ciências Naturais 8o anosovilela100% (1)
- Análise de gráficos de movimentosDocumento5 páginasAnálise de gráficos de movimentosDiana Isabel VieiraAinda não há avaliações
- Funções Sintáticas 1Documento1 páginaFunções Sintáticas 1Vânia Maria SantosAinda não há avaliações
- Flyer Original ImpressãoDocumento1 páginaFlyer Original ImpressãoVânia Maria SantosAinda não há avaliações
- Funcionalidades MVODocumento7 páginasFuncionalidades MVOVânia Maria SantosAinda não há avaliações
- Análise de Gráficos MovimentosDocumento2 páginasAnálise de Gráficos MovimentosVânia Maria SantosAinda não há avaliações
- Aquecimento Global + Correção PDFDocumento3 páginasAquecimento Global + Correção PDFCarla100% (2)
- 12 Antraquinonas NaftodiantronasDocumento24 páginas12 Antraquinonas NaftodiantronasVânia Maria SantosAinda não há avaliações
- Acidos CarboxilicosDocumento40 páginasAcidos CarboxilicosVânia Maria SantosAinda não há avaliações
- Processos de FossilizaçãoDocumento1 páginaProcessos de FossilizaçãoVânia Maria SantosAinda não há avaliações
- O relógio de ShmuelDocumento5 páginasO relógio de ShmuelmarcopereiraeslAinda não há avaliações
- Apontamentos Sobre o Império RomanoDocumento7 páginasApontamentos Sobre o Império RomanoVânia Maria SantosAinda não há avaliações
- Ciências Naturais Objetivos7 Nov2016Documento1 páginaCiências Naturais Objetivos7 Nov2016Vânia Maria SantosAinda não há avaliações
- FichaAvaliação7 GlobalDocumento6 páginasFichaAvaliação7 GlobalVânia Maria SantosAinda não há avaliações
- A.2.7 - Impulsão - Ficha de TrabalhoDocumento1 páginaA.2.7 - Impulsão - Ficha de TrabalhoVânia Maria SantosAinda não há avaliações
- Recurso PDFDocumento7 páginasRecurso PDFVânia Maria SantosAinda não há avaliações
- 2017 - Vírus Da Hepatie CDocumento16 páginas2017 - Vírus Da Hepatie CVânia Maria SantosAinda não há avaliações
- Sebenta HematologiaDocumento386 páginasSebenta HematologiaTiago Teixeira100% (3)
- 2017 - 4 Casos Clínicos HBVDocumento7 páginas2017 - 4 Casos Clínicos HBVVânia Maria SantosAinda não há avaliações
- Aula MacroinvertebradosDocumento33 páginasAula MacroinvertebradosVânia Maria SantosAinda não há avaliações
- 3 FamiliaPapovaviridae PapimomavirusHumanosDocumento18 páginas3 FamiliaPapovaviridae PapimomavirusHumanosVânia Maria SantosAinda não há avaliações
- GPS e queda de corpos na LuaDocumento8 páginasGPS e queda de corpos na LuaVânia Maria SantosAinda não há avaliações
- Livro 1Documento1 páginaLivro 1Vânia Maria SantosAinda não há avaliações
- Vírus da Hepatite B (HBV): Características e ReplicaçãoDocumento25 páginasVírus da Hepatite B (HBV): Características e ReplicaçãoVânia Maria SantosAinda não há avaliações
- Lei Dos Senos e Lei Dos Cossenos Aula 07Documento11 páginasLei Dos Senos e Lei Dos Cossenos Aula 07Vânia Maria SantosAinda não há avaliações
- BF - Aula TP 2 - JCDocumento6 páginasBF - Aula TP 2 - JCVânia Maria SantosAinda não há avaliações
- FT4 - Ciclo CelularDocumento5 páginasFT4 - Ciclo CelularPatricia PedrosoAinda não há avaliações
- Teste de Avaliação Escrita de Biologia 12C 3Documento10 páginasTeste de Avaliação Escrita de Biologia 12C 3Renato Costa100% (6)
- Membrana Plasmática e A PermeabilidadeDocumento39 páginasMembrana Plasmática e A PermeabilidadeWesleyJúniorAinda não há avaliações
- Mduloa3 3 ObtenodeenergiaDocumento7 páginasMduloa3 3 ObtenodeenergiaPatricia PedrosoAinda não há avaliações
- Francisco Prosdocimi e Fabrícia Santos - Sobre Bioinformática, Genoma e CiênciaDocumento4 páginasFrancisco Prosdocimi e Fabrícia Santos - Sobre Bioinformática, Genoma e Ciênciatocanay123Ainda não há avaliações
- GenéticaDocumento80 páginasGenéticaTamaraBuenoAinda não há avaliações
- Aminoácidos, proteínas e enzimas: estrutura, função e regulaçãoDocumento9 páginasAminoácidos, proteínas e enzimas: estrutura, função e regulaçãoAlandersonJosuéVieiraDosReisAinda não há avaliações
- Transmissões hereditáriasDocumento6 páginasTransmissões hereditáriasAna RitaAinda não há avaliações
- MI 1º ANO Unid 3 - Célula - Cap. 7a - Membrana PlasmáticaDocumento4 páginasMI 1º ANO Unid 3 - Célula - Cap. 7a - Membrana PlasmáticaEriczinAinda não há avaliações
- Bancos de Dados Biológicos - Dna, Rna, ProteínasDocumento77 páginasBancos de Dados Biológicos - Dna, Rna, ProteínasVerônica MenezesAinda não há avaliações
- ProteínasDocumento3 páginasProteínasgiovanna ferpAinda não há avaliações
- Plano de Ensino 2010-2 Bioquímica (Cerejeiras)Documento3 páginasPlano de Ensino 2010-2 Bioquímica (Cerejeiras)Fabio José Das DoresAinda não há avaliações
- Farmacologia Resumo ReceptologiaDocumento15 páginasFarmacologia Resumo ReceptologiaPedro MeirelesAinda não há avaliações
- Síntese de NeurotransmissoresDocumento45 páginasSíntese de NeurotransmissoresJaqueline SerraoAinda não há avaliações
- Biologia - Morte CelularDocumento17 páginasBiologia - Morte CelularBiologia Concurso Vestibular100% (2)
- Urease RelatorioDocumento6 páginasUrease RelatorioValter OliveiraAinda não há avaliações
- Ativação de Linfócitos T: Primeiro e Segundo SinaisDocumento5 páginasAtivação de Linfócitos T: Primeiro e Segundo SinaisInara NogueiraAinda não há avaliações
- Via Das Pentoses FosfatoDocumento24 páginasVia Das Pentoses FosfatoCesar SchmollerAinda não há avaliações
- 1 Prova de Avaliaçao Sumativa - 11º AnoDocumento9 páginas1 Prova de Avaliaçao Sumativa - 11º AnoHelena Alves100% (1)
- ProteínasDocumento1 páginaProteínassamuel borgesAinda não há avaliações
- Lista de Exercícios I - Estrutura, Compactação e Duplicação Do Material Genético PDFDocumento3 páginasLista de Exercícios I - Estrutura, Compactação e Duplicação Do Material Genético PDFTaciannaEmídio0% (1)
- As Proteínas Composição, Estrutura e Funções - 3º AnoDocumento26 páginasAs Proteínas Composição, Estrutura e Funções - 3º AnoIgor CajatyAinda não há avaliações
- CitocininasDocumento27 páginasCitocininasCristina Martinho CarrollAinda não há avaliações
- Biologia Celular e Molecular - Moléculas e Mecanismos Na Informação GenéticaDocumento11 páginasBiologia Celular e Molecular - Moléculas e Mecanismos Na Informação GenéticaallanHFAinda não há avaliações
- Trombose e HemostaseDocumento56 páginasTrombose e HemostaseSérgio TeixeiraAinda não há avaliações
- Replicação Do DnaDocumento44 páginasReplicação Do DnaMiriãAinda não há avaliações
- Base cromossômica da hereditariedadeDocumento6 páginasBase cromossômica da hereditariedadeAna Crystina MaiaAinda não há avaliações
- Aula Tempo RealDocumento426 páginasAula Tempo RealNaiaraCP100% (1)
- Avaliação Bimestral - BIOLOGIA - 2º ANODocumento2 páginasAvaliação Bimestral - BIOLOGIA - 2º ANOYasmim RaphaelAinda não há avaliações
- Respiração aeróbia vs fermentaçãoDocumento5 páginasRespiração aeróbia vs fermentaçãoIsabel Henriques0% (1)